图神经网络:(化学领域)再次认识图神经网络
文章说明:
1)参考资料:PYG官方文档。超链。
2)博主水平不高,如有错误还望批评指正。
3)我在百度网盘上传了这篇文章的jupyter notebook和有关文献。超链。提取码8848。
文章目录
-
- Mutagenicity数据集
- 搭建模型
- 训练模型
- 文献阅读
- 重新回来
Mutagenicity数据集
Mutagenicity数据集是用于图分类的数据集。判断该化合物是否具有致突变性。具体信息见代码吧。
导库
from torch_geometric.datasets import TUDataset
下载数据,导入数据,打印信息
dataset=TUDataset(root="/DATA/TUDataset",name='Mutagenicity').shuffle()
print(dataset.num_edge_features,dataset.num_node_features,dataset.num_classes,len(dataset))
#输出如下:
#3 14 2 4337
PS:下载有问题就直接手动下载
观察具体的图
data=dataset[0];print(data.is_directed());data=dataset[1];print(data.is_directed())
#输出如下:
#False
#False
我们把这两幅图画出来
导库
from torch_geometric.utils import to_networkx
定义格式转换函数
def to_molecule(data):
ATOM_MAP=['C','O','Cl','H','N','F','Br','S','P','I','Na','K','Li','Ca']
g=to_networkx(data,node_attrs=['x'])
for u,data in g.nodes(data=True):
data['name']=ATOM_MAP[data['x'].index(1.0)]
del data['x']
return g
PS:我来描述一下g.nodes(data=True)长啥样吧,不然这段代码虽然很好理解但是过于黑箱: [ ( 元素 1 , { “ n a m e ” : 列表 1 } ) , ( 元素 2 , { “ n a m e ” : 列表 2 } ) , … … ( 元素 n , { “ n a m e ” : 列表 n } ) ] [(元素1,\{“name”:列表1\}),(元素2,\{“name”:列表2\}),\dots\dots(元素n,\{“name”:列表n\})] [(元素1,{“name”:列表1}),(元素2,{“name”:列表2}),……(元素n,{“name”:列表n})]。元素 n n n对应 u u u,这里没用;描述一下列表 n n n: [ i = 1 i f n [ i ] = = A T O M _ M A P [ i ] e l s e 0 f o r i i n r a n g e ( 14 ) ] [i=1 \;if \;n[i]==ATOM\_MAP[i] \;else\;0\; for\;i\;in\;range(14)] [i=1ifn[i]==ATOM_MAP[i]else0foriinrange(14)]。
导库
import matplotlib.pyplot as plt
import networkx as nx
绘图函数
def draw_molecule(g,edge_mask=None,draw_edge_labels=False):
g=g.copy().to_undirected()
node_labels={}
for u,data in g.nodes(data=True):
node_labels[u]=data['name']
pos=nx.planar_layout(g)
pos=nx.spring_layout(g,pos=pos)
if edge_mask is None:
edge_color='black'
widths=None
else:
edge_color=[edge_mask[(u,v)] for u,v in g.edges()]
widths=[x*10 for x in edge_color]
nx.draw(g,pos=pos,labels=node_labels,width=widths,edge_color=edge_color,edge_cmap=plt.cm.Blues,node_color='azure')
if draw_edge_labels and edge_mask is not None:
edge_labels={k:('%.2f' % v) for k,v in edge_mask.items()}
nx.draw_networkx_edge_labels(g,pos,edge_labels=edge_labels,font_color='red')
plt.show()
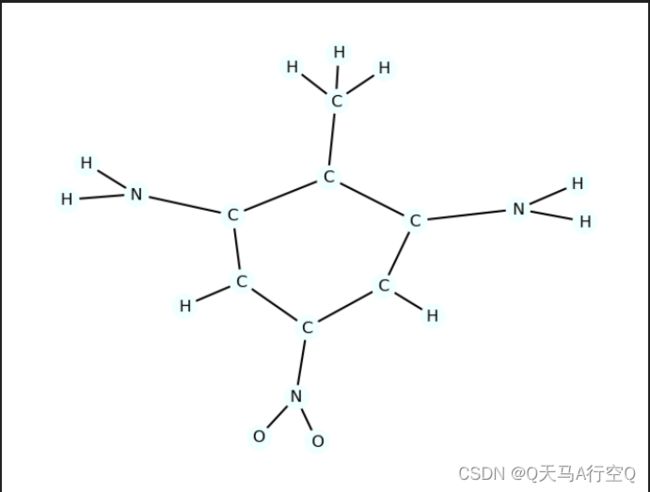
第一幅图
g1=to_molecule(dataset[0])
draw_molecule(g1)
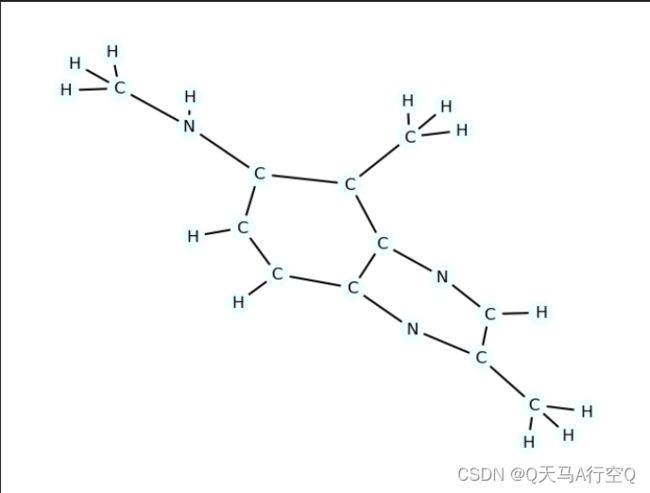
第二幅图
g2=to_molecule(dataset[1])
draw_molecule(g2)
搭建模型
回到正题,目标是图的二分类。
导库
from torch_geometric.nn import GraphConv,global_add_pool
import torch.nn.functional as F
from torch.nn import Linear
import torch
搭建模型
class Net(torch.nn.Module):
def __init__(self,dim):
super(Net,self).__init__()
self.conv1=GraphConv(dataset.num_features,dim)
self.conv2=GraphConv(dim,dim)
self.conv3=GraphConv(dim,dim)
self.conv4=GraphConv(dim,dim)
self.conv5=GraphConv(dim,dim)
self.lin1=Linear(dim,dim)
self.lin2=Linear(dim,dataset.num_classes)
def forward(self,x,edge_index,batch,edge_weight=None):
x=self.conv1(x,edge_index,edge_weight).relu()
x=self.conv2(x,edge_index,edge_weight).relu()
x=self.conv3(x,edge_index,edge_weight).relu()
x=self.conv4(x,edge_index,edge_weight).relu()
x=self.conv5(x,edge_index,edge_weight).relu()
x=global_add_pool(x,batch)
x=self.lin1(x).relu()
x=F.dropout(x,p=0.5,training=self.training)
x=self.lin2(x)
return F.log_softmax(x,dim=-1)
训练模型
训练准备1:定义设备模型与优化器
device=torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model=Net(dim=32).to(device)
optimizer=torch.optim.Adam(model.parameters(),lr=0.001)
print(model)
#输出如下:
#Net(
# (conv1): GraphConv(14, 32)
# (conv2): GraphConv(32, 32)
# (conv3): GraphConv(32, 32)
# (conv4): GraphConv(32, 32)
# (conv5): GraphConv(32, 32)
# (lin1): Linear(in_features=32, out_features=32, bias=True)
# (lin2): Linear(in_features=32, out_features=2, bias=True)
#)
训练准备2:导库训测拆分以及批量划分
from torch_geometric.loader import DataLoader
test_dataset=dataset[:len(dataset)//10]
train_dataset=dataset[len(dataset)//10:]
test_loader=DataLoader(test_dataset,batch_size=128)
train_loader=DataLoader(train_dataset,batch_size=128)
训练准备3:定义训练以及测试
def train(epoch):
model.train()
if epoch==51:
for param_group in optimizer.param_groups:
param_group['lr']=0.5*param_group['lr']
loss_all=0
for data in train_loader:
data=data.to(device)
optimizer.zero_grad()
output=model(data.x,data.edge_index,data.batch)
loss=F.nll_loss(output,data.y)
loss.backward()
loss_all+=loss.item()*data.num_graphs
optimizer.step()
return loss_all/len(train_dataset)
def test(loader):
model.eval()
correct=0
for data in loader:
data=data.to(device)
output=model(data.x,data.edge_index,data.batch)
pred=output.max(dim=1)[1]
correct+=pred.eq(data.y).sum().item()
return correct/len(loader.dataset)
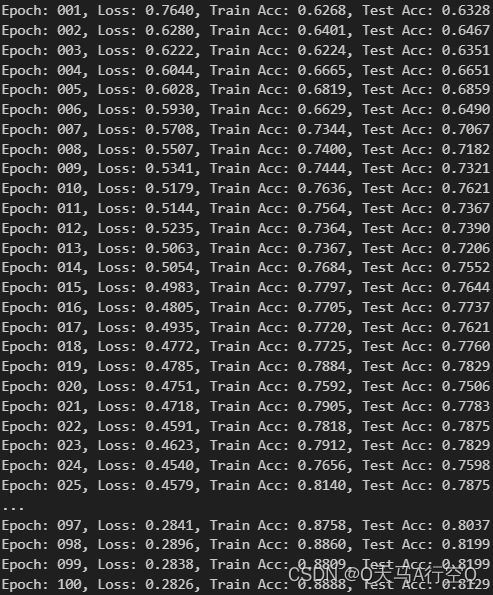
开始训练
for epoch in range(1,101):
loss=train(epoch)
train_acc=test(train_loader)
test_acc=test(test_loader)
print(f'Epoch: {epoch:03d}, Loss: {loss:.4f}, 'f'Train Acc: {train_acc:.4f}, Test Acc: {test_acc:.4f}')
文献阅读
参考文献:Axiomatic Attribution for Deep Networks
文章目的:解释深度神经网络输出与输入的关联(这里下面统称解决这个问题的方法为归因方法)
文章概述:文章证明了两个新定理 S e n s i t i v i t y Sensitivity Sensitivity以及 I m p l e m e n t a t i o n I n v a r i a n c e Implementation\;Invariance ImplementationInvariance并且说明所有归因方法应该满足两个定理。之前大多方法均不满足所以作者提出一种新的归因方法满足两个定理—— I n t e g r a t e d G r a d i e n t s Integrated\;Gradients IntegratedGradients。
S e n s i t i v i t y Sensitivity Sensitivity:要求对于输入变化输出变化应该一致。举个反例模型存在梯度消失。
I m p l e m e n t a t i o n I n v a r i a n c e Implementation\;Invariance ImplementationInvariance:要求对于相同输入无论使用哪种网络输出不变。否则不是等效网络。
I n t e g r a t e d G r a d i e n t s IntegratedGradients IntegratedGradients: I n t e g r a t e d G r a n d s i ( x ) = ( x i − x i ′ ) ∫ α = 0 1 ∂ F ( x ′ + α × ( x − x ′ ) ) ∂ x i d α IntegratedGrands_i(x)=(x_i-x_i')\int_{\alpha=0}^{1}\frac{\partial F(x'+\alpha\times(x-x'))}{\partial x_i}d\alpha IntegratedGrandsi(x)=(xi−xi′)∫α=01∂xi∂F(x′+α×(x−x′))dα。这里 x ′ x' x′是基准输入。


PS1:这个图展示了积分梯度的优越性。
PS2:文章结论可以用于多种神经网络模型并且效果出类拔萃。文章还有许多其他工作这里并不展开因为时间篇幅原因还请读者自行阅读。这篇文章是很好的。
重新回来
阅读文献之后我们重新回来。现在有了两种方法求解输出相对边权重的梯度1. A t t r i b u t i o n e i = ∣ ∂ F ( x ) ∂ ω e i ∣ Attribution_{e_i}=|\frac{\partial F(x)}{\partial \omega_{e_i}}| Attributionei=∣∂ωei∂F(x)∣ 2. A t t r i b u t i o n e i = ∫ α = 0 1 ∂ F ( x α ) ∂ ω e i d α Attribution_{e_i}=\int_{\alpha=0}^1\frac{\partial F(x_{\alpha})}{\partial \omega_{e_i}}d\alpha Attributionei=∫α=01∂ωei∂F(xα)dα。这里我们初始边缘权重为1基线为0简化公式否则就是十分复杂。
导库
from captum.attr import Saliency,IntegratedGradients
import numpy as np
定义函数
def model_forward(edge_mask,data):
batch=torch.zeros(data.x.shape[0],dtype=int)
out=model(data.x,data.edge_index,batch,edge_mask)
return out
def explain(method,data,target=0):
input_mask=torch.ones(data.edge_index.shape[1]).requires_grad_(True)
if method=='ig':
ig=IntegratedGradients(model_forward)
mask=ig.attribute(input_mask,target=target,additional_forward_args=(data,),internal_batch_size=data.edge_index.shape[1])
elif method=='saliency':
saliency=Saliency(model_forward)
mask=saliency.attribute(input_mask,target=target,additional_forward_args=(data,))
else:
raise Exception('Unknown explanation method')
edge_mask=np.abs(mask.cpu().detach().numpy())
if edge_mask.max()>0:
edge_mask=edge_mask/edge_mask.max()
return edge_mask
导库
from collections import defaultdict
import random
定义函数
def aggregate_edge_directions(edge_mask,data):
edge_mask_dict=defaultdict(float)
for val,u,v in list(zip(edge_mask,*data.edge_index)):
u,v=u.item(),v.item()
if u>v:
u,v=v,u
edge_mask_dict[(u,v)]+=val
return edge_mask_dict
开始绘制
data=random.choice([t for t in test_dataset if not t.y.item()])
mol=to_molecule(data)
for title,method in [('Integrated Gradients','ig'),('Saliency','saliency')]:
edge_mask=explain(method,data,target=0)
edge_mask_dict=aggregate_edge_directions(edge_mask,data)
plt.figure(figsize=(10,5))
plt.title(title)
draw_molecule(mol,edge_mask_dict)
jupyter notebook内的输出如下
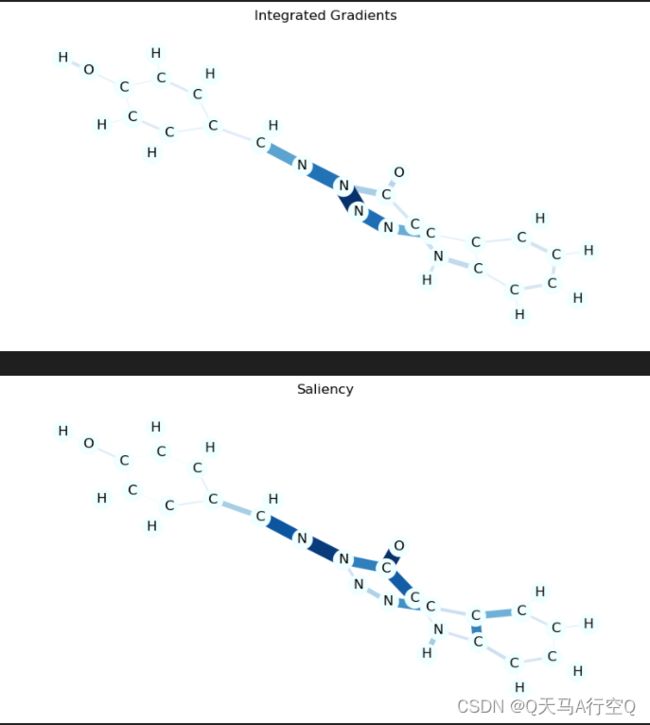
PS1:如果报错发现多个运行设备让它们全在CPU上跑就好。
PS2:越深说明对于目标影响越大。