医学图像分割 nnUNetV2 分割自定义2d数据集
文章目录
- 1 环境安装(Pytorch)
-
- 1.1 安装conda
- 1.1 安装pytorch
- 1.3 安装nnUNet
- 1.4 安装隐藏层(可选)
- 2 配置自定义数据集
-
- 2.1 数据集格式
- 2.2 创建需要目录
- 2.3 数据格式转换
-
- 2.3.1 修改路径与数据集名称
- 2.3.2 修改训练集与测试集
- 2.3.3 修改掩码所在的文件夹,并修改后缀
- 3 数据预处理
-
- 3.1 配置环境变量
- 3.2 对数据集进行预处理
- 4 训练 Train
- 5 推理 Inference
-
- 5.1 寻找合适的配置
- 5.1 运行 `find_best_configuration.py`
- 5.2 模型推理
- 5.3 执行后处理
- 6 评估 evaluation
-
-
-
- label为RGB格式需要进行额外处理
-
-
- 7 自定义划分数据集
-
- 7.1 `final_split.json`自定义
- 7.2 执行训练
- 参考
nnUNet: https://github.com/MIC-DKFZ/nnUNet/tree/master
1 环境安装(Pytorch)
1.1 安装conda
使用Anaconda或者 Miniconda,下载https://mirrors.tuna.tsinghua.edu.cn/anaconda/
创建虚拟环境,并激活,这里建议python>=3.9
conda create -n nnUNet python=3.10 -y
conda activate
1.1 安装pytorch
安装Pytorch: https://pytorch.org/get-started/previous-versions/
pip install torch==2.0.0+cu117 torchvision==0.15.1+cu117 torchaudio==2.0.1 --index-url https://download.pytorch.org/whl/cu117
1.3 安装nnUNet
参考: https://github.com/MIC-DKFZ/nnUNet/blob/master/documentation/installation_instructions.md
- 作标准化基线、开箱即用的分割算法或使用预训练模型进行推理
pip install nnunetv2
- 当作框架使用
git clone https://github.com/MIC-DKFZ/nnUNet.git
cd nnUNet
pip install -e .
解释pip install -e .:
- 最后
pip install -e .相当于python setup.py,也就是运行上图这个setup.py文件 - 向终端添加几个新命令。这些命令用于运行整个
nnU-Net pipeline。您可以从系统上的任何位置执行它们。所有nnU-Net命令都带有前缀“nnUNet_”,以便于识别
可见setup.py中的命令行格式
entry_points={
'console_scripts': [
'nnUNetv2_plan_and_preprocess = nnunetv2.experiment_planning.plan_and_preprocess_entrypoints:plan_and_preprocess_entry', # api available
'nnUNetv2_extract_fingerprint = nnunetv2.experiment_planning.plan_and_preprocess_entrypoints:extract_fingerprint_entry', # api available
'nnUNetv2_plan_experiment = nnunetv2.experiment_planning.plan_and_preprocess_entrypoints:plan_experiment_entry', # api available
'nnUNetv2_preprocess = nnunetv2.experiment_planning.plan_and_preprocess_entrypoints:preprocess_entry', # api available
'nnUNetv2_train = nnunetv2.run.run_training:run_training_entry', # api available
'nnUNetv2_predict_from_modelfolder = nnunetv2.inference.predict_from_raw_data:predict_entry_point_modelfolder', # api available
'nnUNetv2_predict = nnunetv2.inference.predict_from_raw_data:predict_entry_point', # api available
'nnUNetv2_convert_old_nnUNet_dataset = nnunetv2.dataset_conversion.convert_raw_dataset_from_old_nnunet_format:convert_entry_point', # api available
'nnUNetv2_find_best_configuration = nnunetv2.evaluation.find_best_configuration:find_best_configuration_entry_point', # api available
'nnUNetv2_determine_postprocessing = nnunetv2.postprocessing.remove_connected_components:entry_point_determine_postprocessing_folder', # api available
'nnUNetv2_apply_postprocessing = nnunetv2.postprocessing.remove_connected_components:entry_point_apply_postprocessing', # api available
'nnUNetv2_ensemble = nnunetv2.ensembling.ensemble:entry_point_ensemble_folders', # api available
'nnUNetv2_accumulate_crossval_results = nnunetv2.evaluation.find_best_configuration:accumulate_crossval_results_entry_point', # api available
'nnUNetv2_plot_overlay_pngs = nnunetv2.utilities.overlay_plots:entry_point_generate_overlay', # api available
'nnUNetv2_download_pretrained_model_by_url = nnunetv2.model_sharing.entry_points:download_by_url', # api available
'nnUNetv2_install_pretrained_model_from_zip = nnunetv2.model_sharing.entry_points:install_from_zip_entry_point', # api available
'nnUNetv2_export_model_to_zip = nnunetv2.model_sharing.entry_points:export_pretrained_model_entry', # api available
'nnUNetv2_move_plans_between_datasets = nnunetv2.experiment_planning.plans_for_pretraining.move_plans_between_datasets:entry_point_move_plans_between_datasets', # api available
'nnUNetv2_evaluate_folder = nnunetv2.evaluation.evaluate_predictions:evaluate_folder_entry_point', # api available
'nnUNetv2_evaluate_simple = nnunetv2.evaluation.evaluate_predictions:evaluate_simple_entry_point', # api available
'nnUNetv2_convert_MSD_dataset = nnunetv2.dataset_conversion.convert_MSD_dataset:entry_point' # api available
],
},
1.4 安装隐藏层(可选)
nnU-net能够给出其生成的网络拓扑图
- 使用pip 安装
pip install --upgrade git+https://github.com/FabianIsensee/hiddenlayer.git@more_plotted_details #egg=hiddenlayer
- 源码安装
网站: https://github.com/FabianIsensee/hiddenlayer.git
cd nnUNet
git clone https://github.com/FabianIsensee/hiddenlayer.git
cd hiddenlayer
pip install -e .
2 配置自定义数据集
2.1 数据集格式
- 这里数据集中的文件均为
RGB三通道图片png图片 - Image为RGB,Label也为RGB,
单通道灰度图会报错,访问三个通道只有一个
2.2 创建需要目录
- 创建
nnUNet_raw保存格式转换后的数据集 - 创建
nnUNet_result保存结果文件
2.3 数据格式转换
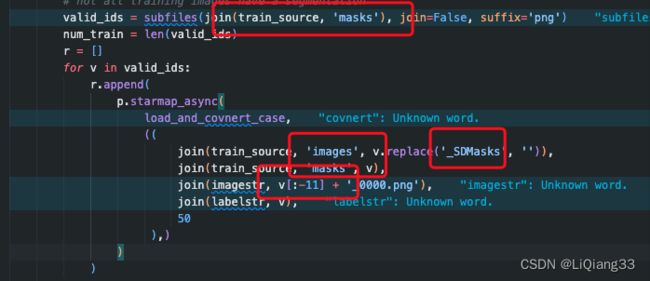
修改文件nnunetv2/dataset_conversion/Dataset120_RoadSegmentation.py
2.3.1 修改路径与数据集名称
Note:
- 数据集名称
dataset_name的格式:Dataset数字_名称, 数字大于10,为数据集的ID - 转换后数据集中原图
image与mask的图片名称去掉_0000相对应
2.3.2 修改训练集与测试集
2.3.3 修改掩码所在的文件夹,并修改后缀
- 根据自己的图片命名规则修改即可
之后运行 Dataset120_RoadSegmentation.py
3 数据预处理
3.1 配置环境变量
vim .bashrc
在最后添加
export nnUNet_raw="/sharefiles1/hanliqiang/GitCode/nnUNet/nnUNet_raw"
export nnUNet_preprocessed="/sharefiles1/hanliqiang/GitCode/nnUNet/nnunetv2/preprocessing"
export nnUNet_results="/sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results"
使其生效
source .bashrc
3.2 对数据集进行预处理
- 将2d格式进行转换
nnUNetv2_plan_and_preprocess -d DATASET_ID --verify_dataset_integrit
DATASET_ID就是数据转换步骤中你设置的datasetname中的id
# nnUNetv2_plan_and_preprocess -d 120 --verify_dataset_integrity
nnUNetv2_plan_and_preprocess -d 110 --verify_dataset_integrity
4 训练 Train
nnUNetv2_train DATASET_NAME_OR_ID UNET_CONFIGURATION FOLD
DATASET_NAME_OR_ID这里为数据集的id- 二维图像的话
UNET_CONFIGURATION就填2d FOLD代表几折交叉验证,nnUNet中为5
我的
nnUNetv2_train 110 2d 5
5 推理 Inference
5.1 寻找合适的配置
按照官方文档:https://github.com/MIC-DKFZ/nnUNet/blob/master/documentation/how_to_use_nnunet.md
运行与数据集有关的ID
nnUNetv2_find_best_configuration 110 -c CONFIGURATIONS
报错
best_score = max([i['result'] for i in all_results.values()]) ValueError: max() arg is an empty sequence[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-tnfUtFVi-1686187402399)(null)]
不知道什么原因,也没有深究
于是看他main 函数,按照数据集的ID修改,修改源码后:
5.1 运行 find_best_configuration.py
位置: nnunetv2/evaluation/find_best_configuration.py
main 文件的内容
if __name__ == '__main__':
# find_best_configuration(110,
# default_trained_models,
# True,
# 8,
# False,
# (0, 1, 2, 3, 4))
find_best_configuration(
dataset_name_or_id=110, # 指定数据集编号,其中有对应的模型
allowed_trained_models=default_trained_models,
allow_ensembling=True,
num_processes=8,
overwrite=False,
folds=(5, ),
strict=False
)
修改的参数:
- 数据集ID为110,数据集名称符合命名规范
Dataset110_StentSegmentation - 交叉验证为5
等待数据集训练完成,运行修改后的文件
修改后输出
Configuration 3d_fullres not found in plans nnUNetPlans.
Inferred plans file: /sharefiles1/hanliqiang/GitCode/nnUNet/nnunetv2/preprocessing/Dataset110_StentSegmentation/nnUNetPlans.json.
Configuration 3d_lowres not found in plans nnUNetPlans.
Inferred plans file: /sharefiles1/hanliqiang/GitCode/nnUNet/nnunetv2/preprocessing/Dataset110_StentSegmentation/nnUNetPlans.json.
Configuration 3d_cascade_fullres not found in plans nnUNetPlans.
Inferred plans file: /sharefiles1/hanliqiang/GitCode/nnUNet/nnunetv2/preprocessing/Dataset110_StentSegmentation/nnUNetPlans.json.
***All results:***
nnUNetTrainer__nnUNetPlans__2d: 0.9095163742866601
*Best*: nnUNetTrainer__nnUNetPlans__2d: 0.9095163742866601
***Determining postprocessing for best model/ensemble***
WARNING: Not all files in folder_ref were found in folder_predictions. Determining postprocessing should always be done on the entire dataset!
Removing all but the largest foreground region did not improve results!
***Run inference like this:***
nnUNetv2_predict -d 110 -i INPUT_FOLDER -o OUTPUT_FOLDER -f 5 -tr nnUNetTrainer -c 2d -p nnUNetPlans
***Once inference is completed, run postprocessing like this:***
nnUNetv2_apply_postprocessing -i OUTPUT_FOLDER -o OUTPUT_FOLDER_PP -pp_pkl_file /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/postprocessing.pkl -np 8 -plans_json /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/plans.json
- 可见会输出预测的命令
- 按照要求修改相应的文件夹即可
5.2 模型推理
模版命令
nnUNetv2_predict -d 110 -i INPUT_FOLDER -o OUTPUT_FOLDER -f 5 -tr nnUNetTrainer -c 2d -p nnUNetPlans
INPUT_FOLDER输入文件夹,这里指定为nnUNet_raw/Dataset110_StentSegmentation/imagesTsOUTPUT_FOLDER输出文件夹,这里指定为nnUNet_predict_result/Dataset110_result
修改后的命令为:
nnUNetv2_predict -d 110 -i nnUNet_raw/Dataset110_StentSegmentation/imagesTs -o nnUNet_predict_result/Dataset110_result -f 5 -tr nnUNetTrainer -c 2d -p nnUNetPlans
5.3 执行后处理
这里只需要替代相应的文件夹即可
原命令(根据find_configure)
nnUNetv2_apply_postprocessing -i OUTPUT_FOLDER -o OUTPUT_FOLDER_PP -pp_pkl_file /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/postprocessing.pkl -np 8 -plans_json /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/plans.json
- 输入文件夹为预测的结果,修改为:
nnUNet_predict_result/Dataset110_result - 输出文件夹为集成之后的结果,修改为:
nnUNet_predict_result/Dataset110_result_pp
执行命令:
nnUNetv2_apply_postprocessing -i nnUNet_predict_result/Dataset110_result -o nnUNet_predict_result/Dataset110_result_pp -pp_pkl_file /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/postprocessing.pkl -np 8 -plans_json /sharefiles1/hanliqiang/GitCode/nnUNet/nnUnet_results/Dataset110_StentSegmentation/nnUNetTrainer__nnUNetPlans__2d/crossval_results_folds_5/plans.json
6 评估 evaluation
这个没有在官方文档里看到命令,这里使用其提供的源码文件进行
位置:nnunetv2/evaluation/evaluate_predictions.py
修改main函数,使得路径对应
if __name__ == '__main__':
folder_ref = '/sharefiles1/hanliqiang/GitCode/nnUNet/nnUNet_raw/Dataset110_StentSegmentation/labelsTs'
folder_pred = '/sharefiles1/hanliqiang/GitCode/nnUNet/OUTPUT_FOLDER'
output_file = '/sharefiles1/hanliqiang/GitCode/nnUNet/eval_result/summary.json'
image_reader_writer = SimpleITKIO()
file_ending = '.png'
regions = labels_to_list_of_regions([1])
ignore_label = None
num_processes = 12
compute_metrics_on_folder(folder_ref, folder_pred, output_file, image_reader_writer, file_ending, regions, ignore_label,
num_processes)
floder_ref测试集的GT文件floder_pred预测后的文件,即推理结果output_file输出评价结果的json文件labels_to_list_of_regions([1])前景是1,指定需要评估的区域为label1- 文件后缀,修改为
.png文件
label为RGB格式需要进行额外处理
数据集中的标签为RGB格式,三个通道中的数值一致,因此这里只需要去其中一个通道即可
修改自定义的文件路径之后,运行文件,即可得到最终的结果
没有后处理的结果:
{
"foreground_mean": {
"Dice": 0.9050778866067624,
"FN": 31.09931506849315,
"FP": 27.187214611872147,
"IoU": 0.8313335684356011,
"TN": 261787.04223744292,
"TP": 298.67123287671234,
"n_pred": 325.8584474885845,
"n_ref": 329.77054794520546
},
"mean": {
"(1,)": {
"Dice": 0.9050778866067624,
"FN": 31.09931506849315,
"FP": 27.187214611872147,
"IoU": 0.8313335684356011,
"TN": 261787.04223744292,
"TP": 298.67123287671234,
"n_pred": 325.8584474885845,
"n_ref": 329.77054794520546
}
}
}
对我这个数据集来说,好像没有改变,原因待分析,猜测可能是patch的问题
7 自定义划分数据集
参考官方文档: https://github.com/MIC-DKFZ/nnUNet/blob/master/documentation/manual_data_splits.md

可以看到,nnUNet在训练的时候是使用do_split函数来进行数据集划分和加载的,如果没有则自动创建

因此我们需要自定义一个final_split.json ,并在训练的时候指定为0-fold交叉验证即可
7.1 final_split.json自定义
其格式为:
[
{'train': ['la_003', 'la_004', 'la_005', 'la_009', 'la_010', 'la_011', 'la_014', 'la_017', 'la_018', 'la_019', 'la_020', 'la_022', 'la_023', 'la_026', 'la_029', 'la_030'],
'val': ['la_007', 'la_016', 'la_021', 'la_024']},
{'train': [...], 'val': ....}
]
每一个字典为一个交叉验证的方式,注意其中的文件是文件名,可以观察之前生成的文件形式
7.2 执行训练
运行命令,交叉验证选择0,表示使用自定义划分数据集
nnUNetv2_train 110 2d 0
输出信息:
2023-06-23 12:33:07.749127: unpacking dataset...
2023-06-23 12:33:07.951198: unpacking done...
2023-06-23 12:33:07.951562: do_dummy_2d_data_aug: False
2023-06-23 12:33:07.956370: Using splits from existing split file: /sharefiles1/hanliqiang/GitCode/nnUNet/nnunetv2/preprocessing/Dataset110_StentSegmentation/splits_final.json
2023-06-23 12:33:07.956599: The split file contains 1 splits.
2023-06-23 12:33:07.956617: Desired fold for training: 0
2023-06-23 12:33:07.956630: This split has 1690 training and 286 validation cases.
2023-06-23 12:33:12.000968: Unable to plot network architecture:
2023-06-23 12:33:12.001016: failed to execute PosixPath('dot'), make sure the Graphviz executables are on your systems' PATH
2023-06-23 12:33:12.037173:
可以看到已经使用自定义数据集进行训练了
未完待续…
参考
nnUNetv2训练二维图像数据集 https://blog.csdn.net/Halloween111/article/details/130928829
nnUNet保姆级使用教程!从环境配置到训练与推理(新手必看)
https://blog.csdn.net/m0_68239345/article/details/128886376