PSP - MMseqs2 编译最新版本源码 (14-7e284) 支持 MPI 功能 MSA 快速搜索
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/131966061
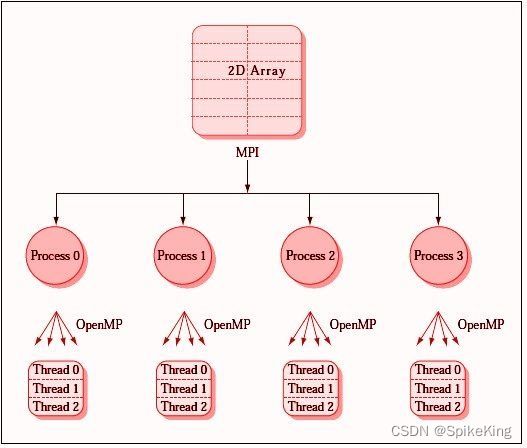
MPI (Message Passing Interface) 是用于并行计算的标准化和可移植的消息传递接口,可以在分布式内存的多台计算机上运行并行程序。MPI 标准定义一些库函数的语法和语义,这些函数对于编写可移植的消息传递程序非常有用,支持 C、C++ 和 Fortran 语言。MPI 的主要功能是实现多个进程之间的数据交换和同步,以及提供一些集合操作,如广播、归约、扫描等。MPI 还支持一些高级特性,如动态进程管理、单边通信、I/O 等。MPI 是目前最广泛使用的并行计算接口之一,有多种开源的实现,如 Open MPI、MPICH、MVAPICH 等。MPI 也有多种语言的绑定,如 Java, Python, R 等。
其他配置,参考:使用 MMseqs2 工具快速搜索蛋白质序列数据库 (GMGC)
可执行文件下载地址:CSDN Downloads - MMseqs2最新版本可执行文件
构建 GMGC 蛋白质基因的测试数据库,建议去 CPU 与 内存 较多的机器中,运行:
# Time for processing: 0h 17m 9s 510ms
mmseqs createdb data/gmgc.fa gmgc.db
mmseqs createindex gmgc.db tmp
创建索引的日志,如下:
indexdb gmgc.db gmgc.db --seed-sub-mat nucl:nucleotide.out,aa:VTML80.out -k 0 --alph-size nucl:5,aa:21 --comp-bias-corr 1 --max-seq-len 65535 --max-seqs 300 --mask 1 --mask-lower-case 0 --spaced-kmer-mode 1 -s 7.5 --k-score 0 --check-compatible 0 --search-type 0 --split 0 --split-memory-limit 0 -v 3 --threads 232
Target split mode. Searching through 5 splits
Estimated memory consumption: 691G
... # 5 个 splits 执行 5 次计算
Index table: counting k-mers
[=================================================================] 100.00% 59.49M 37s 155ms
Index table: Masked residues: 122957738
Index table: fill
[=================================================================] 100.00% 59.49M 1m 6s 231ms
...
GitHub: MMseqs2
1. Conda 配置低版本 (13.45111)
使用 conda 安装 mmseqs2:
conda install -c conda-forge -c bioconda mmseqs2
配置:
if [ -f /Path to MMseqs2/util/bash-completion.sh ]; then
source /Path to MMseqs2/util/bash-completion.sh
fi
版本号:
conda list mmseqs2
# packages in environment at miniconda3/envs/torch-def:
#
# Name Version Build Channel
mmseqs2 13.45111 h95f258a_1 bioconda
版本号较低是 13.45111,而 GitHub 最新版本是 MMseqs2 Release 14-7e284
2. Make 编译高版本 (14-7e284)
较低版本的命令支持不充分,建议使用最新版本,最新版本可以下载,也可以编译,建议通过编译的方式。
下载项目:
git clone [email protected]:soedinglab/MMseqs2.git
编译命令,如下:
cd MMseqs2
mkdir build && cd build
cmake .. -DHAVE_AVX2=1
make
# make install
如需编译 mpi 版本 (OpenMP and Message Passing Interface),建议使用命令:
cd MMseqs2
mkdir build-mpi && cd build-mpi
cmake .. -DHAVE_MPI=1 -DCMAKE_BUILD_TYPE=Release -DCMAKE_INSTALL_PREFIX=.
make
日志如下:
...
-- Performing Test ATOMIC_LIBRARY_NATIVE
-- Performing Test ATOMIC_LIBRARY_NATIVE - Failed # 正常错误
...
编译完成的文件,位于:MMseqs2-master/build/src/mmseqs
Make 与 CMake 的关系:
- Make 是智能的批处理工具,可以根据 Makefile 文件中的规则来编译和链接源文件,生成可执行文件或者库文件。Makefile 文件需要手动编写,指定每个目标文件如何从源文件生成,以及如何链接目标文件生成最终的产品。Make 的优点是简单、灵活、通用,可以用于任何语言和平台。
- CMake 是跨平台的构建系统生成器,可以根据 CMakeLists.txt 文件中的指令来生成不同平台和工具的构建系统,比如 Makefile、Visual Studio、Xcode等。CMakeLists.txt 文件也需要程序员手动编写,但是相对于 Makefile 来说,更加简洁和高级,不需要指定具体的编译器或者链接器命令,只需要描述项目的结构和依赖关系。CMake 的优点是可以自动发现和配置工具链、系统库、编译选项等,支持多种生成器和平台,方便移植和扩展。
3. 测试 MMseqs2
在 .bashrc 中添加,启动命令:
export PATH=~/bin:$PATH
查看: which mmseqs
需要安装包 libatomic1,即:
apt-get install libatomic1
测试搜索 MSA 的命令,蛋白质序列长度1029,数据库60G,5个分片:
- 耗时:[Info] Time taken to execute commands is 138 seconds.
# params
#=========================================
mmseqs=mmseqs
tmp=tmp_my
query_fasta=T1157s1_A1029.fasta
query_db=$tmp/queryDB
target_db=virus4/gmgc/gmgc.db
target_db_index=${target_db}.idx
result_db=$tmp/res # 文件
result_db_realign=$tmp/res_realign
result_db_realign_filter=$tmp/res_realign_filter
a3m_db=result.a3m
tmp_db=$tmp/tmp
#=========================================
mkdir -p $tmp
time_start=$(date +%s)
#=========================================
$mmseqs createdb ${query_fasta} ${query_db}
$mmseqs search ${query_db} ${target_db} ${result_db} ${tmp_db} --db-load-mode 2 --num-iterations 1 -s 1 --max-seqs 10000 -e 0.1 -a
$mmseqs align ${query_db} ${target_db_index} ${result_db} ${result_db_realign} --db-load-mode 2 -e 10 --max-accept 100000 --alt-ali 10 -a
$mmseqs filterresult ${query_db} ${target_db_index} ${result_db_realign} ${result_db_realign_filter} --db-load-mode 2 --qid 0 --qsc 0.8 --diff 0 --max-seq-id 1.0 --filter-min-enable 100
$mmseqs result2msa ${query_db} ${target_db_index} ${result_db_realign_filter} ${a3m_db} --msa-format-mode 6 --db-load-mode 2 --filter-msa 1 --filter-min-enable 1000 --diff 3000 --qid 0.0,0.2,0.4,0.6,0.8,1.0 --qsc 0 --max-seq-id 0.95
$mmseqs rmdb ${result_db_realign_filter}
$mmseqs rmdb ${result_db}
$mmseqs rmdb ${result_db_realign}
#=========================================
time_end=$(date +%s)
time_take=$(( time_end - time_start ))
echo "[Info] MMseqs2 path is ${mmseqs} ."
echo "[Info] Time taken to execute commands is ${time_take} seconds."
搜索出 426 个结果:426 result.a3m,数据如下:
>A
DRVRALRRETVEMFYYGFDNYMKVAFPEDELRPVSCTPLTRDLKNPRNFELNDVLGNYSLTLIDSLSTLAILASAPAEDSGTGPKALRDFQDGVAALVEQYGDGRPGPSGVGRRARGFDLDSKVQVFETVIRGVGGLLSAHLFAIGALPITGYQPLRQEDDLFNPPPIPWPNGFTYDGQLLRLALDLAQRLLPAFYTKTGLPYPRVNLRHGIPFYVNSPLHEDPPAKGTTEGPPEITETCSAGAGSLVLEFTVLSRLTGDPRFEQAAKRAFWAVWYRKSQIGLIGAGVDAEQGHWIGTYSVIGAGADSFFEYALKSHILLSGHALPNQTHPSPLHKDVNWMDPNTLFEPLSDAENSAESFLEAWHHAHAAIKRHLYSEREHPHYDNVNLWTGSLVSHWVDSLGAYYSGLLVLAGEVDEAIETNLLYAAIWTRYAALPERWSLREKTVEGGLGWWPLRPEFIESTYHLYRATKDPWYLYVGEMVLRDITRRCWTPCGWAGLQNVLSGEKSDRMESFFLGETTKYMYLLFDDDHPLNKLDASFVFTTEGHPLILPKPKSARRSRNSPRSSQKALTVYQGEGFTNSCPPRPSITPLSGSVIAARDDIYHPARMVDLHLLTTSKHALDGGQMSGQHMAKSNYTLYPWTLPPELLPSNGTCAKVYQPHEVTLEFASNTQQVLGGSAFNFMLSGQNLERLSTDRIRVLSLSGLKITLQLVEEGEREWRVTKLNGIPLGRDEYVVINRAILGDVSDPRFNLVRDPVIAKLQQLHQVNLLDDTTTEEHPDNLDTLDTASAIDLPQDQSSDSEVPDPANLSALLPDLSSFVKSLFARLSNLTSPSPDPSSNLPLNVVINQTAILPTGIGAAPLPPAASNSPSGAPIPVFGPVPESLFPWKTIYAAGEACAGPLPDSAPRENQVILIRRGGCSFSDKLANIPAFTPSEESLQLVVVVSDDEHEGQSGLVRPLLDEIQHTPGGMPRRHPIAMVMVGGGETVYQQLSVASAIGIQRRYYIESSGVKVKNIIVDDGDGGVDG
>GMGC10.285_640_775.MNL1|built-environment 832 0.483 2.234E-258 38 991 1029 0 953 1046
--------------------------------------LTRDRVNPAHIEVNDVLGNYSLSVVDSLSTLAILASDPESDL-DHYNALDDFQEHVELVIEEYGDGSPGPAGQGRRARGFDLDSKVQVFETTIRGLGGLLSAHLFAIGELPIRGYEPDIQKDG------IHWPNGFVYDGQLLRLAQDLGERLLPAFHTPTGLPYPRVNLRYGTPFYENSPLNNDAEhgqchKTQKPKGAREITETCSAGAGSLVLEFTTLSRLTNDDRFERLAKRAFWAVWERRSASGLIGAGIDAETGAWIGPWTGIGAGIDSFFEYAFKSHILLS--ALTGDSY--------------------NLTEDSPDAFLQTWKDAHSAIMRHVYRDAyfTHPHYAQNDLYTGGPRLTWIDSLSAYYPGLLVLAGELDEAMTAHLLYTALWSRYGALPERWDATTGTIHGGLKWWGGRPEFIESTWYIYHATKDPWYLHIGEMALRDIKRRCYTKCGWAGLQDVRTGEQSDRMESFFLGETAKYMYLLFDPDHPLNNIDAPWVFTTEGHPLIIPK---ANRTRTRRYHAEKDTSLSAAIPPTaEQCPLPPPLLPLTISSTAARSDIFHAASLARLHLMPVgNKPGAPSLDWASDHpsvtmlgpESPTNFTFYPWTLPLDLIPADGYSTKLSNKPTFDLTFPTTVGTGLEIGTLQKIDGG----------VLVNSISGLRFGMVLEDSGpdEDEYRIYTLGNFALGRDEHIALSRDTLSQInpTDPHFTRMRDveamdliidvphpaePAIEAL--VYNNSALNDHVFDFNLDfDLDALDATSSpgemvLDsLPKALFADvhrlAEQLD--GLVGVLPDADSIDDALRDAAKQLRSKSPAASSSTKMPYQqpkglqrFTTPAMLPIGPGAAPLPATIDSAVDPRSLPT------GHLPYTSILVVdSDLCnTSPLPLDLVSTHNVLIIRRGGCSFSKKLAAIPSFPPSAKALQIVLVVSFGSDEG----TRPLVDEAQLTPKGLPRRHPICLALVPGGQTVWE-------------------------------------
>GMGC10.241_341_405.MNL1|soil 756 0.569 1.592E-232 2 658 1029 54 704 740
--IKVLRQETVELFYHGYDNYLRHAFPEDELRPLTCGALTRDRENPAHIELNDALGNYSLTLIDSLSTLAILASsadAKQKPNGwlsTATTPLEDFQEGIKLLVEYYGDGTDGPDGEGKRARGFDLDSKVQVFETVIRGVGGLLSAHLFAVGDLPIRGYVPkLKTRHGKHG---IHWRNGLVYDGQLLRLAQDLADRLVPAFYTPTDLPYPRVNLRHGVSFYPNSPYNAD---SGTgmcskqQGGAQEITETCSAGAGSLVLEFTTLSRLTGNDLYERLAKQAFYAVWNRRSSIGLIGAGIDSETGDWVNSITGIGAGIDSFFEYAFKSHILLS----------------------NLPFDHEEEDIHPSDEFLETWQEAHEAIKRHVYRSdiLQHPHFAQVDMNTGASKYWWIDSLSAFYPGLLTLSGELDEAITVHLLYTALWTRYSAMPERWSTYTGEIESGLRWWGGRPEFIESSWYLYRATMDPWYLHVGEMALRDIKRRCWTKCGWAGLQDVRTGEKSDRMESFFLGETAKYLFLLFDVDHPLNSLDAPFIFTTEGHPLVIPQRVIPRKSRDGiPKRFARRTT----RGADNAmCSIPPSFVPLSLSPTAARDDLFHAASLARLDLMPSIEETESSLVEFNNHhpsismadiRSPSNYTYYPWTLPLELVPQNGSCSRI----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
测试多进程:
- 进程设置为 10,即
--threads 10 - 提速至 65 秒,
[Info] Time taken to execute commands is 65 seconds.
即
$mmseqs createdb ${query_fasta} ${query_db}
$mmseqs search ${query_db} ${target_db} ${result_db} ${tmp_db} --db-load-mode 2 --num-iterations 1 -s 1 --max-seqs 10000 -e 0.1 -a --threads 10
$mmseqs align ${query_db} ${target_db_index} ${result_db} ${result_db_realign} --db-load-mode 2 -e 10 --max-accept 100000 --alt-ali 10 -a --threads 10
$mmseqs filterresult ${query_db} ${target_db_index} ${result_db_realign} ${result_db_realign_filter} --db-load-mode 2 --qid 0 --qsc 0.8 --diff 0 --max-seq-id 1.0 --filter-min-enable 100 --threads 10
$mmseqs result2msa ${query_db} ${target_db_index} ${result_db_realign_filter} ${a3m_db} --msa-format-mode 6 --db-load-mode 2 --filter-msa 1 --filter-min-enable 1000 --diff 3000 --qid 0.0,0.2,0.4,0.6,0.8,1.0 --qsc 0 --max-seq-id 0.95 --threads 10
非 MPI 版本的日志,如下:
MMseqs Version: GITDIR-NOTFOUND
测试 MPI:
- 一定要使用支持
MPI的版本。 mmseqs search命令可以使用, MPI 设置为 10,即--mpi-runner "mpirun --allow-run-as-root -np 10- 提速至 56 秒,
[Info] Time taken to execute commands is 56 seconds.
即
$mmseqs createdb ${query_fasta} ${query_db}
$mmseqs search ${query_db} ${target_db} ${result_db} ${tmp_db} --db-load-mode 2 --num-iterations 1 -s 1 --max-seqs 10000 -e 0.1 -a --threads 10 --mpi-runner "mpirun --allow-run-as-root -np 10"
$mmseqs align ${query_db} ${target_db_index} ${result_db} ${result_db_realign} --db-load-mode 2 -e 10 --max-accept 100000 --alt-ali 10 -a --threads 10
$mmseqs filterresult ${query_db} ${target_db_index} ${result_db_realign} ${result_db_realign_filter} --db-load-mode 2 --qid 0 --qsc 0.8 --diff 0 --max-seq-id 1.0 --filter-min-enable 100 --threads 10
$mmseqs result2msa ${query_db} ${target_db_index} ${result_db_realign_filter} ${a3m_db} --msa-format-mode 6 --db-load-mode 2 --filter-msa 1 --filter-min-enable 1000 --diff 3000 --qid 0.0,0.2,0.4,0.6,0.8,1.0 --qsc 0 --max-seq-id 0.95 --threads 10
--allow-run-as-root避免 MPI 的 root 错误
MPI 版本的日志,如下:
MMseqs Version: GITDIR-NOTFOUND-MPI
Bug
遇到 Bug,MMseqs库的索引构建问题:
Invalid database read for database data file=virus4/gmgc/gmgc.db.idx, database index=virus4/gmgc/gmgc.db.idx.index
getData: local id (4294967295) >= db size (50)
需要重新构建数据库的索引:
mmseqs createdb data/gmgc.fa gmgc.db
mmseqs createindex gmgc.db tmp
遇到 Bug,缺少 libatomic.so.1 文件:
error while loading shared libraries: libatomic.so.1: cannot open shared object file: No such file or directory
需要安装包:
apt-get install libatomic1
参考
- 获取程序运行时间 - Linux Shell 脚本