【MATLAB】matlab多目标遗传算法的用法
函数名:gamultiobj
详细的不多说,帮助文档写的很清楚,这里仅作为备忘简要写出常用用法
基于的算法是改良的NSGA-II
用于求解多目标优化问题
object1 和object 2有一定的竞争关系
得到的解是一组解,称为帕累托前沿 pareto front
options = optimoptions('gamultiobj','PlotFcn',...
{@gaplotpareto},'UseParallel',true);
func=@recomp;
nvar=4;
lb=[0.2,32+273.15,7.4e3,10e3];
ub=[0.4,50+273.15,8e3,15e3];
[var,fval,exitflag,output] = gamultiobj(func,nvar,[],[],[],[],lb,ub,options);
关键是最后一句,前面都是为了写着换行方便定义的变量
从接受的参数开始说:
func是要优化的函数,优化后得到的是函数最小值,如果求最大值,加个负号即可。@代表获取函数句柄(在C语言里就是函数指针)。
nvar是待优化变量的个数,必填。
后面四个空着的,分别代表线性约束不等式的A,b, 等式的A,b,如果没有空着
lb是变量最小值,ub是变量最大值。一一对应。
options是其他附加选项
这里加了:画出帕累托前沿图(实时更新的),并且使用并行(matlab会自动给定使用的核心数)
使用并行会大大加速求解过程,但是启动并行需要几分钟。
控制选项optimoptions
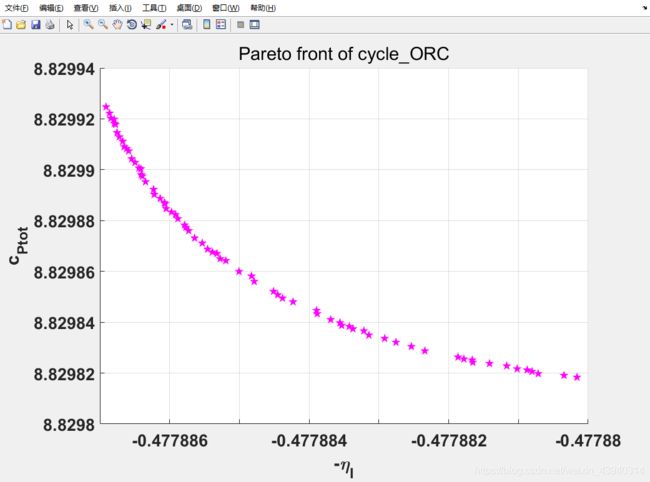
如何让帕累托前沿变宽?
问题描述
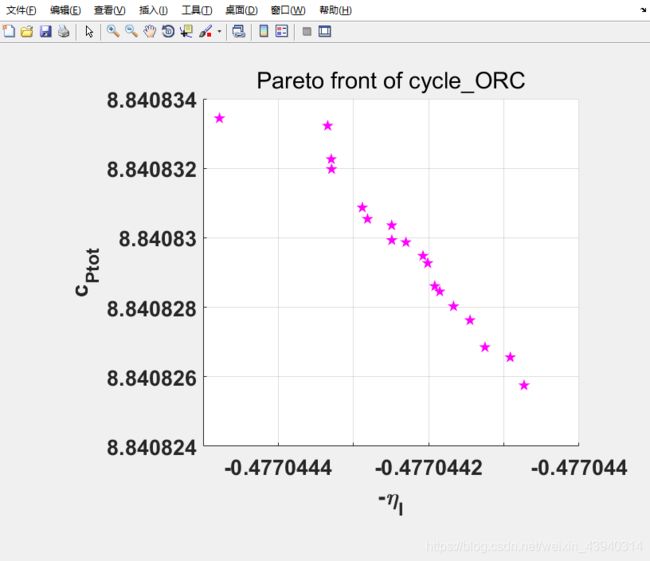
我在使用多目标遗传算法的时候,发现优化结果可能掉入一个“坑”里。也就是说本来应该有不同的结果供我选择,但是最后却越优化越集中,最后过度优化,帕累托前沿从一条线变成了一个点,如图:
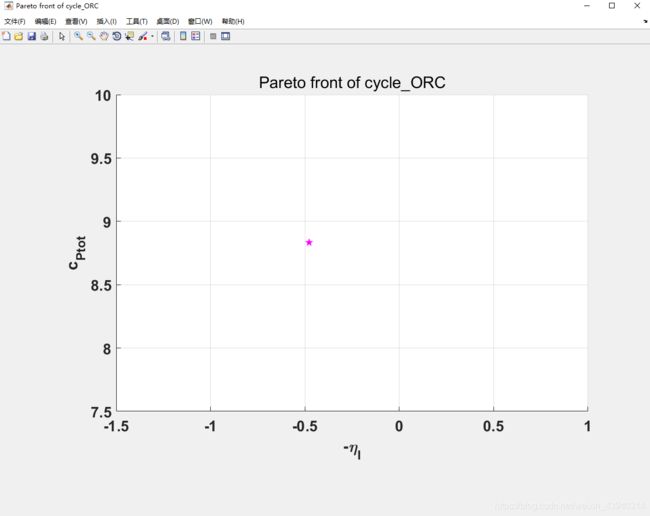
实际上上面这个图,虽然看上去是个线,但是仔细看的话会发现数值非常集中,如果坐标范围调大一些,其实还是缩成了一个点。
操作方法
1 调整种群数量
多目标遗传算法,按照官方帮助,默认种群数量是50,这显然是有点儿小了。所以我们把他调整为200
在optimoptions括号内加一个参数对
'PopulationSize',200
2 调整帕累托前沿中个体的距离
参考官方帮助
https://ww2.mathworks.cn/help/gads/gamultiobj-options-effects.html
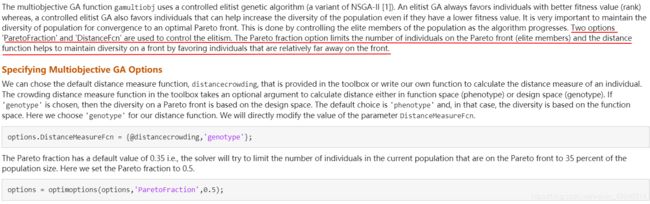
这里说两个选项是用来控制精英数量的(精英就是在帕累托前沿上的点),一个是DistanceFcn 一个是ParetoFraction
DistanceFcn:帕累托前沿上点的距离,这个可以自己定义,也可以用默认选项。
ParetoFraction:精英的在种群中的比例。就是帕累托前沿上点的数量除以种群数量。
下面给了两个选项的例子
‘DistanceMeasureFcn’,'distancecrowding'
或者
‘DistanceMeasureFcn’,{@distancecrowding,'genotype'}
这个就是利用默认的距离函数来计算的。默认计算的是表现型(生物上基因表现出来的性状叫表现型)phenotype(其实就是因变量)的距离。它注释里面的function space其实就是因变量空间。也可以改为genotype就是基因型。
3 控制精英比例(paretoFraction)
另一个就是控制帕累托前沿上的点占种群数量的比例
默认是0.35
这里改为0.5
'ParetoFraction',0.5
4 放宽收敛要求
gamultiobj默认的因变量收敛相对值是1e-4
这里改为1e-2
'FunctionTolerance',1e-3
默认最大代数是 nvar*200(自变量个数)
我们放宽为200
'MaxGenerations',200