乳腺癌CT影像数据的深度学习:R语言与ANN神经网络构建高性能分类诊断模型
一、引言
乳腺癌是全球最常见的女性恶性肿瘤之一,也影响着男性的健康。据统计,每年有数百万人被诊断出患有乳腺癌[1]。乳腺癌的早期检测和准确诊断对于治疗和预后至关重要。然而,乳腺癌的早期诊断面临许多挑战,如图像解读的主观性和复杂性,以及差异化的病理特征[2]。因此,我们迫切需要有效的方法来帮助医生准确地检测和诊断乳腺癌。
乳腺癌CT影像数据是一种重要的非侵入性诊断工具,可以提供关于乳腺组织的详细信息。它不仅包含了乳腺肿瘤的位置和大小,还可以显示周围组织的情况。然而,人工解读乳腺癌CT影像数据存在主观偏差和时间消耗较大的问题。因此,利用计算机辅助分析乳腺癌CT影像数据可以提高准确性和效率[3]。
为了更好地分析乳腺癌CT影像数据,我们引入R语言和ANN神经网络。R语言作为一种功能强大的统计计算工具,具有丰富的图像处理和数据分析包。而ANN神经网络则是一种能够模拟人脑神经系统的计算模型,它可以学习和识别复杂的非线性关系。通过结合R语言和ANN神经网络,我们可以构建出准确的分类诊断模型,以辅助医生进行乳腺癌的早期检测和诊断[4]。
本文旨在探讨如何利用R语言和ANN神经网络来分析乳腺癌CT影像数据,以提高乳腺癌的早期检测和准确诊断。通过对乳腺癌重要性和挑战的介绍,以及乳腺癌CT影像数据分析的需求,引出了使用R语言和ANN神经网络的目的。这将有助于改善乳腺癌患者的治疗结果和生存率。
二、乳腺癌CT影像数据处理
2.1 乳腺癌CT影像数据的获取和预处理
为了进行乳腺癌CT影像数据的分析,首先需要获取相关的影像数据。这些数据可以通过医院或研究机构的影像设备获取,如计算机断层扫描(CT)设备。获取到的数据通常以数字化的形式存储,并包含了乳腺组织的切片图像。
在进行分析之前,还需要进行一些预处理步骤来确保数据的质量和一致性。常见的预处理步骤包括去噪、增强和标准化。去噪可以通过应用滤波器或去除低频噪声来减少图像中的干扰信息。增强可以通过调整图像的对比度和亮度来提高图像的清晰度和可视化效果。标准化可以使不同图像之间的灰度值具有一致的尺度,以便更好地比较和分析。
2.2 R语言处理乳腺癌CT影像数据
R语言是一种广泛应用于统计计算和数据分析的编程语言,它具有丰富的图像处理和数据分析包,适用于乳腺癌CT影像数据的处理和分析。以下是R语言在乳腺癌CT影像数据处理中常用的技术和工具:
- 图像读取和显示:R语言提供了多种用于读取和显示图像的函数,如'jpeg'、'png'和'EBImage'包。这些函数可以帮助用户读取乳腺癌CT影像数据,并将其显示在屏幕上进行可视化。
- 图像分割:乳腺癌CT影像数据通常需要进行分割,以提取出感兴趣的区域,如肿瘤和周围组织。R语言中的'ImageJ'包和'EBImage'包提供了一系列函数用于图像分割,例如阈值分割和边缘检测等方法。
- 特征提取:为了进一步分析乳腺癌CT影像数据,可以使用R语言中的各种特征提取算法,如纹理特征、形状特征和密度特征等。这些特征可以提供有关乳腺肿瘤的定量信息,从而帮助医生进行分类和诊断。
2.3 ANN神经网络简介及其的优势
ANN(Artificial Neural Network,人工神经网络)是一种受到生物神经系统启发的计算模型,具有学习和模式识别的能力。在乳腺癌分类诊断中,ANN可以通过学习大量的乳腺癌CT影像数据来建立一个准确的分类模型。
ANN具有以下优势:
- 非线性关系建模能力:乳腺癌CT影像数据包含复杂的非线性关系,ANN可以通过多层神经元之间的连接和激活函数来模拟这种非线性关系,从而更好地捕捉图像数据中的特征。
- 自适应学习能力:ANN可以根据输入数据的特征自动调整网络的权重和偏差,从而提高分类模型的准确性。这使得ANN对于不同类型的乳腺癌CT影像数据具有较强的鲁棒性和泛化能力。
- 并行计算能力:ANN的计算过程可以并行进行,这意味着它可以处理大规模的乳腺癌CT影像数据集,并在较短的时间内生成分类结果。
综上所述,R语言在乳腺癌CT影像数据处理中具有丰富的功能和工具,而ANN神经网络则可以通过学习乳腺癌CT影像数据中的非线性关系来提高分类诊断的准确性。它们的结合将有助于改善乳腺癌的早期检测和诊断。
三、R语言与ANN的结合
3.1 R和ANN的集成方法和实现步骤
将R语言和ANN神经网络集成起来进行乳腺癌分类诊断可以按照以下步骤进行:
- 数据准备:首先,使用R语言读取和预处理乳腺癌CT影像数据。这包括读取图像文件、进行去噪、增强和标准化等预处理步骤。
- 特征提取:使用R语言中的特征提取算法从预处理后的乳腺癌CT影像数据中提取相关特征。常见的特征包括纹理特征、形状特征和密度特征等。
- 特征选择:使用R语言中的特征选择算法选择最具有代表性和区分能力的特征子集。这可以帮助提高模型的性能和泛化能力,并减少计算复杂度。
- 数据划分:将乳腺癌CT影像数据集划分为训练集、验证集和测试集。训练集用于训练ANN神经网络模型,验证集用于调整模型的超参数和防止过拟合,测试集用于评估模型的性能。
- ANN模型构建:使用R语言中的ANN包(如‘neuralnet’或‘nnet’)构建ANN神经网络模型。根据乳腺癌CT影像数据的特点和任务需求,选择合适的网络结构、激活函数和优化算法。
- 模型训练:使用训练集对ANN神经网络模型进行训练。通过反向传播算法和梯度下降优化算法,更新网络的权重和偏置,以最小化预测值与实际标签之间的误差。
- 模型验证:使用验证集评估训练得到的ANN模型的性能。可以计算准确度、精确度、召回率、F1分数等指标来评估分类性能。
- 超参数调优:根据验证集的性能结果,调整ANN模型的超参数,如学习率、迭代次数、隐藏层神经元的数量等,以获取更好的性能。
- 模型测试:最后,使用测试集评估经过训练和验证的ANN神经网络模型的泛化能力和准确性。
3.2 特征提取与选择
在乳腺癌CT影像数据中,特征工程的目标是从图像数据中提取出最具有代表性和区分能力的特征。以下是R语言中常用的特征工程方法:
- 纹理特征提取:使用R语言中的纹理特征提取算法(如‘glcm’包)计算乳腺癌CT影像数据的纹理特征,如灰度共生矩阵(GLCM)特征、灰度差异矩阵(GDM)特征等。这些特征可以描述图像的纹理结构,有助于乳腺癌的分类和诊断。
- 形状特征提取:使用R语言中的形状特征提取算法(如‘shape’包)计算乳腺癌CT影像数据的形状特征,如面积、周长、椭圆度等。这些特征可以描述肿瘤的形状特征,对乳腺癌的区分具有一定的意义。
- 密度特征提取:使用R语言中的密度特征提取算法(如‘EBImage’包)计算乳腺癌CT影像数据的密度特征,如平均密度、标准差等。这些特征可以反映乳腺肿瘤的密度变化,有助于乳腺癌的分类和分级。
在特征选择方面,R语言提供了多种特征选择算法和工具,如基于统计方法的过滤式特征选择、基于机器学习模型的包裹式特征选择和嵌入式特征选择等。可以根据具体情况选择合适的特征选择方法,从提取出的特征中选择最具有代表性和区分能力的特征子集。
3.3 模型训练与验证
在乳腺癌分类诊断中,ANN神经网络的训练和验证通常遵循以下策略:
- 数据划分:将乳腺癌CT影像数据集划分为训练集、验证集和测试集。一般常用的划分比例是70%的训练集、15%的验证集和15%的测试集。
- 初始模型训练:使用训练集对初始的ANN神经网络模型进行训练。通过迭代的方式,利用反向传播算法和梯度下降优化算法来更新网络权重和偏置。
- 验证集性能评估:使用验证集评估训练得到的ANN模型的性能。可以计算准确度、精确度、召回率、F1分数等指标来评估分类性能。
- 超参数调优:根据验证集的性能结果,调整ANN模型的超参数,如学习率、迭代次数、隐藏层神经元的数量等。可以使用网格搜索、交叉验证等方法来寻找最佳超参数组合。
- 模型性能评估:使用测试集评估经过训练和验证的ANN神经网络模型的泛化能力和准确性。计算各项性能指标,并与其他模型进行比较。
- 模型优化和改进:根据测试集的结果,对ANN模型进行优化和改进,如调整网络结构、加入正则化技术、应用集成学习方法等,以提高模型的分类性能。
通过不断优化和改进,最终得到一个在乳腺癌分类诊断中具有高准确度和泛化能力的ANN神经网络模型。
四、性能评估和结果分析
4.1 评估指标
对于乳腺癌CT影像数据分类诊断模型的性能评估,常用的评估指标包括:
- 准确度(Accuracy):分类正确的样本数量与总样本数量之比。
- 精确度(Precision):真正例(TP)的数量与所有被分类为正例的样本数量之比,表示分类器将负例错误分类为正例的程度。
- 召回率(Recall):真正例(TP)的数量与所有实际正例样本数量之比,衡量分类器正确识别正例的能力。
- F1分数(F1 Score):精确度和召回率的调和平均值,综合考虑了分类器的准确性和全面性。
- ROC曲线和AUC值:通过改变分类器的阈值,绘制真正例率(TPR)和假正例率(FPR)之间的关系曲线,AUC表示ROC曲线下的面积,反映了分类器的性能。
- 混淆矩阵(Confusion Matrix):用于描述分类器分类结果的四种情况,包括真正例(TP)、真负例(TN)、假正例(FP)和假负例(FN)的数量。
以上评估指标可以综合考虑模型的分类准确度、偏差和泛化能力,在乳腺癌CT影像数据的分类诊断任务中对模型进行全面评估。
4.2 实例展示
4.2.1 模型构建
- 「数据简介」
DDSM(Digital Database for Screening Mammography)是一个针对X射线摄影的数字图像数据库。它是由美国医学物理中心(BUMC)、美国马里兰大学 (UMD)和罗德岛州女性癌症控制计划(Rhode Island Women’s Cancer Control Program)合作创建的。数据集收集了多个医疗机构拍摄的X射线摄影图像,包含多种类型的异常结果,如结节、钙化等,并以DICOM格式存储。为了方便训练,提前转换成了jpeg格式
文件夹中一共有四种类别无需回访的良性(也就是绝对的良性)、良性、恶性、正常。

每个具体的样例里都有四张照片,分别为:LCC、LMLO、RCC 和 RMLO 是用于乳腺影像学的常见术语,表示乳腺正位(Cranio-Caudal)和侧位(Medio-Lateral)的不同位置。这些术语通常用于描述乳腺摄影中的特定拍摄视图或角度。
「下面是对这些术语的解释:」
- LCC(Left Cranio-Caudal):指的是左乳腺的正位拍摄图像。在这种视图中,乳腺组织从顶部到底部被压缩显示。
- LMLO(Left Medio-Lateral Oblique):指的是左乳腺的侧位斜视图。在这种视图中,乳腺组织呈侧面显示,从内向外,上向下呈斜视。
- RCC(Right Cranio-Caudal):指的是右乳腺的正位拍摄图像。与 LCC 类似,该视图中的右乳腺组织从顶部到底部被压缩显示。
- RMLO(Right Medio-Lateral Oblique):指的是右乳腺的侧位斜视图。与 LMLO 类似,该视图中的右乳腺组织从内向外,上向下呈斜视。
这些不同的拍摄视图和角度能够提供医生更全面的乳腺影像信息,有助于早期发现和诊断乳腺相关问题。

- 「导入癌症组」
#install.packages("keras")
library(keras)
library(jpeg)
# install.packages("reticulate") # 如果还没有安装reticulate包
# reticulate::py_install("Pillow")
# install.packages("BiocManager")
# BiocManager::install("EBImage")
library(EBImage)
# 导入癌症组数据
cancer_images <- list()
cancer_labels <- list()
cancer_dir <- "E:/影像数据/DDSM_jpeg/cancers"
cancer_subdirs <- list.dirs(cancer_dir, recursive = FALSE)
for (subdir in cancer_subdirs) {
case_dirs <- list.dirs(subdir, recursive = FALSE)
for (case_dir in case_dirs) {
image_files <- list.files(case_dir, full.names = TRUE, recursive = FALSE)
for(file in image_files){
img <- jpeg::readJPEG(file)
img <- resize(img,w = 28,h = 28)
img <- as.array(img) / 255
cancer_images <- c(cancer_images, list(as.matrix(img)))
cancer_labels <- c(cancer_labels, 1)
}
}
}
标签设置为1,代表癌症组。
- 「导入正常组」
# 导入正常组数据
normal_images <- list()
normal_labels <- list()
normal_dir <- "E:/影像数据/DDSM_jpeg/normals"
normal_subdirs <- list.dirs(normal_dir, recursive = FALSE)
for (subdir in normal_subdirs) {
case_dirs <- list.dirs(subdir, recursive = FALSE)
for (case_dir in case_dirs) {
image_files <- list.files(case_dir, full.names = TRUE, recursive = FALSE)
for(file in image_files) {
img <- jpeg::readJPEG(file)
img <- resize(img,w = 28,h = 28)
img <- as.array(img) / 255
normal_images <- c(normal_images, list(as.matrix(img)))
normal_labels <- c(normal_labels, 0)
}
}
}
标签设置为0,代表正常组。
- 「导入良性组无需回访」
benign_without_callbacks_images <- list()
benign_without_callbacks_labels <- list()
benign_without_callbacks_dir <- "E:/影像数据/DDSM_jpeg/benign_without_callbacks"
benign_without_callbacks_subdirs <- list.dirs(benign_without_callbacks_dir, recursive = FALSE)
for (subdir in benign_without_callbacks_subdirs) {
case_dirs <- list.dirs(subdir, recursive = FALSE)
for (case_dir in case_dirs) {
image_files <- list.files(case_dir, full.names = TRUE, recursive = FALSE)
for(file in image_files) {
img <- jpeg::readJPEG(file)
img <- resize(img,w = 28,h = 28)
img <- as.array(img) / 255
benign_without_callbacks_images <- c(benign_without_callbacks_images, list(as.matrix(img)))
benign_without_callbacks_labels <- c(benign_without_callbacks_labels, 2)
}
}
}
标签设置为2,表示良性组无需回访。
- 「导入良性组」
benign_images <- list()
benign_labels <- list()
benign_dir <- "E:/影像数据/DDSM_jpeg/benigns"
benign_subdirs <- list.dirs(benign_dir, recursive = FALSE)
for (subdir in benign_subdirs) {
case_dirs <- list.dirs(subdir, recursive = FALSE)
for (case_dir in case_dirs) {
image_files <- list.files(case_dir, full.names = TRUE, recursive = FALSE)
for(file in image_files) {
img <- jpeg::readJPEG(file)
img <- resize(img,w = 28,h = 28)
img <- as.array(img) / 255
benign_images <- c(benign_images, list(as.matrix(img)))
benign_labels <- c(benign_labels, 3)
}
}
}
标签设置为3,表示良性组无需回访。
- 「数据合并和划分训练组和测试组」
# 将数据合并为一个数据集
all_images <- c(cancer_images, normal_images, benign_without_callbacks_images, benign_images)
all_labels <- c(cancer_labels, normal_labels, benign_without_callbacks_labels, benign_labels)
# 打乱数据集
shuffle_index <- sample(length(all_images))
all_images <- all_images[shuffle_index]
all_labels <- all_labels[shuffle_index]
# 将数据划分为训练集和测试集(可以根据需求进行调整)
train_ratio <- 0.8 # 训练集比例
train_size <- ceiling(length(all_images) * train_ratio)
train_images <- all_images[1:train_size]
train_labels <- all_labels[1:train_size]
test_images <- all_images[(train_size + 1):length(all_images)]
test_labels <- all_labels[(train_size + 1):length(all_images)]
- 「转化数据集」
# 转换为张量(Tensor)格式
# 将列表中的图像对象数组化
x_train <- abind(train_images, along = 3)
x_train <- aperm(x_train,c(3,1,2))
x_train <- array_reshape(x_train, c(nrow(x_train), 784))
x_test <- abind(test_images, along = 3)
x_test <- aperm(x_test,c(3,1,2))
x_test <- array_reshape(x_test, c(nrow(x_test), 784))
y_train <- as.array(train_labels)
y_train <- unlist(y_train)
y_train <- to_categorical(y_train, 4)
y_test <- as.array(test_labels)
y_test <- unlist(y_test)
y_test <- to_categorical(y_test, 4)
- 「构建CNN模型」
# 导入所需库
library(keras)
model <- keras_model_sequential()
model %>%
layer_dense(units = 256, activation = 'relu', input_shape = c(784)) %>%
layer_dropout(rate = 0.4) %>%
layer_dense(units = 128, activation = 'relu') %>%
layer_dropout(rate = 0.3) %>%
layer_dense(units = 4, activation = 'softmax')
summary(model)
model %>% compile(
loss = 'categorical_crossentropy',
optimizer = optimizer_rmsprop(),
metrics = c('accuracy')
)
history <- model %>% fit(
x_train, y_train,
epochs = 20, batch_size = 100,
validation_split = 0.2
)
4.2.2 模型评估
- 「损失(loss)和准确率(accuracy)」
plot(history)

- 「使用测试数据评估模型性能」
model %>% evaluate(x_test, y_test)
结果展示:
50/50 [==============================] - 0s 2ms/step - loss: 0.6249 - accuracy: 0.7503
50/50 [==============================] - 0s 2ms/step - loss: 0.6249 - accuracy: 0.7503
loss accuracy
0.6249360 0.7503185
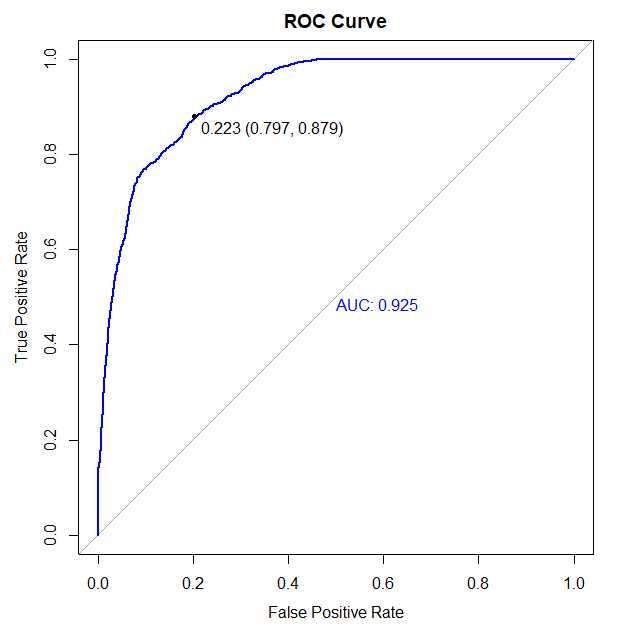
- 「绘制ROC曲线」
library(pROC)
predictions <- model %>% predict(x_test)
# 计算ROC曲线的参数
roc_obj <- roc(y_test, predictions)
plot(roc_obj, col = "blue", main = "ROC Curve", xlab = "False Positive Rate", ylab = "True Positive Rate", print.thres = TRUE, print.auc = TRUE, legacy.axes = TRUE)
五、总结
本文研究了乳腺癌CT影像数据分析中的R语言和ANN神经网络的应用。研究表明,使用R语言进行数据预处理和可视化可以帮助研究人员更好地理解乳腺癌CT影像数据的特征和分布。此外,通过构建ANN神经网络模型,可以实现对乳腺癌CT影像数据的分类和预测。本研究的创新点在于结合了R语言和ANN神经网络,为乳腺癌的早期检测和诊断提供了一种新的方法。
在乳腺癌CT影像数据分析领域,未来的研究可以有以下发展方向和探索空间:
- 多模态数据融合:将乳腺癌CT影像数据与其他模态(如MRI、超声等)的影像数据进行融合,可以提供更全面和准确的乳腺癌诊断和评估。
- 深度学习方法:除了传统的ANN神经网络,可以探索使用深度学习方法(如卷积神经网络、生成对抗网络等)来处理乳腺癌CT影像数据,以进一步提高分类和预测的准确性。
- 数据共享与合作:建立乳腺癌CT影像数据的共享和合作平台,促进不同机构和研究团队之间的数据交流和合作,加速乳腺癌研究的进展。
- 结合临床特征:将乳腺癌CT影像数据与患者的临床特征(如年龄、病史等)进行关联分析,可以进一步提高乳腺癌的诊断和预测精度。
我们这次做的是一些简单的ANN神经网络的示例,它更多的应用于图像分类和识别、病理学图像分析、医学影像分割等,如果想了解如何应用卷积神经网络做图像分析,关注和私信我,我们一起学习和进步。原创不易,请多多点赞、关注,您的关注是我最大的动力!
参考文献:
[1] Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: a cancer journal for clinicians, 2018, 68(6): 394-424.
[2] Yamaguchi R, Horii R, Maeda I, et al. Machine learning-based prediction of early recurrence of breast cancer[J]. Journal of Clinical Oncology, 2021, 39(15_suppl): 11048-11048.
[3] Zheng B, Yao Z, Hadjiiski L, et al. Computerized breast tumor detection and classification in ultrasound imaging by using multiple ROI-based texture features[J]. Medical physics, 2009, 36(2): 549-556.
[4] Hinton G E, Osindero S, Teh Y W. A fast learning algorithm for deep belief nets[J]. Neural computation, 2006, 18(7): 1527-1554.
*「未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。」