ggrisk | 高分文章必备的三图联动就这样轻松搞定哦!~(附完整教程)
1写在前面
终于有时间更新了,忙碌的一周也是告一段落了。
下午去单位的时候简直要晒费了,有一种生而为人,我很抱歉的感觉。
各位小伙伴的暑假是怎么度过的呀,欢迎分享图片给我,也让我看下浪浪山外面的世界。
今天写写美丽的三图联动吧,在高分paper中还是比较多见的,可以相对直观的观察variable对结局事件的风险影响。
2用到的包
rm(list = ls())
library(tidyverse)
library(ggrisk)
library(survival)
library(rms)
library(patchwork)
3示例数据
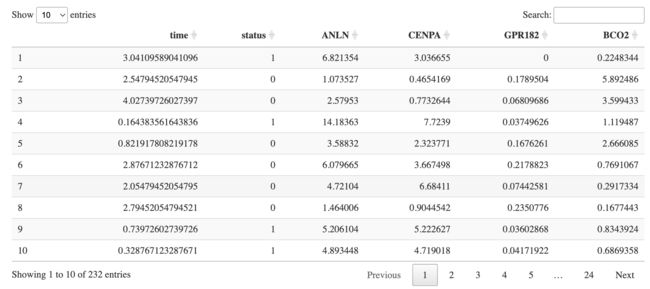
我们今天就用包内自带的示例数据了哦。
dat <- LIRI
DT::datatable(dat)
4构建模型
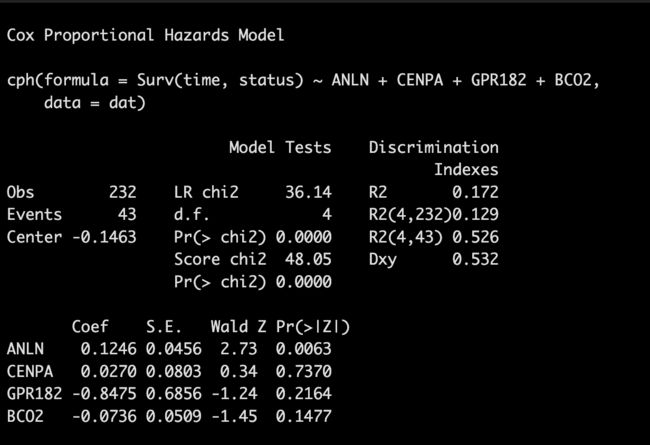
用cph或者coxph构建模型都可以,看大家的喜好吧。
fit1 <- cph(Surv(time,status)~ANLN+CENPA+GPR182+BCO2,dat)
fit1
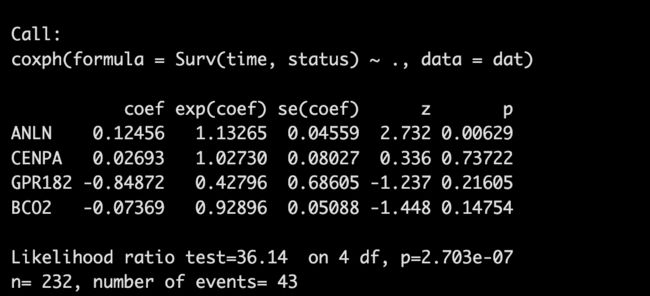
fit2 <- coxph(Surv(time, status)~., dat)
fit2
5初步可视化
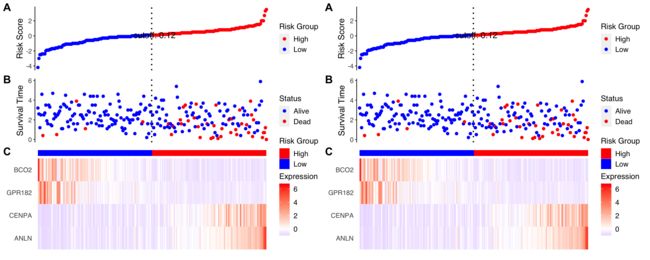
我们把2个模型都可视化一下,然后拼在一起。
p1 <- ggrisk(fit1)
p2 <- ggrisk(fit2)
wrap_plots(p1, p2)
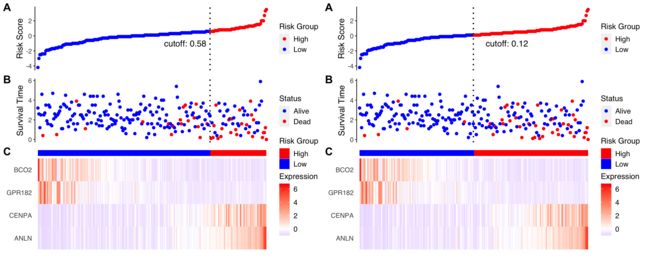
6更换cutoff的选取方式
cutoff. value可选:
1️⃣ median; 2️⃣ roc; 3️⃣ cutoff;
p1 <- ggrisk(fit1,
cutoff.value='cutoff',
cutoff.x = 150,
cutoff.y = -1
)
p2 <- ggrisk(fit2,
cutoff.value='median',
cutoff.x = 150,
cutoff.y = -1
)
wrap_plots(p1, p2)
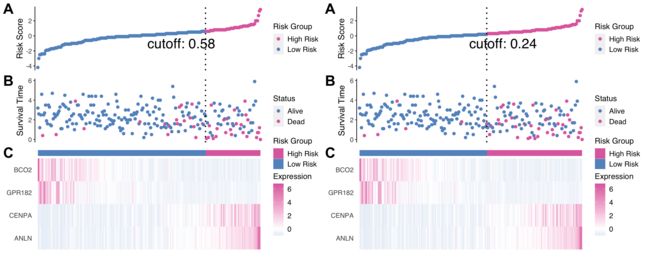
7修改细节
我在后面都做了备注,大家对着修改吧!~
p1 <- ggrisk(fit1,
heatmap.genes = NULL, # 热图中显示的基因
cutoff.value='cutoff',
cutoff.x = 150, # cutoff的位置
cutoff.y = -1,
size.cutoff = 8, # cutoff文字的大小
#============================================
code.highrisk = 'High Risk',# 高风险的label
code.lowrisk = 'Low Risk', # 低风险的lanbel
title.A.ylab='Risk Score', # 图A中y轴的title
title.B.ylab='Survival Time', # 图B中y轴的title
title.A.legend='Risk Group', # 图A的legend的title
title.B.legend='Status', # 图B的legend的title
title.C.legend='Expression', # 图C的legend的title
#============================================
size.ABC = 2, # 标识ABC的大小
color.A = c(low = '#6395C7',high = '#E06EAD'),# 图A的颜色
color.B = c(code.0 ='#6395C7',code.1 = '#E06EAD'), # 图B的颜色
color.C = c(low = "#6395C7",median = 'white',high = '#E06EAD'), # 图C的颜色
vjust.A.ylab=1, # 图A中y轴label到y坐标轴的距离
vjust.B.ylab=2, #图B中y轴label到y坐标轴的距离
relative_heights = c(0.1,0.1,0.01,0.15)# A.B.C三图的相对高度
)
p2 <- ggrisk(fit2,
heatmap.genes = NULL, # 热图中显示的基因
cutoff.value='roc',
cutoff.x = 150, # cutoff的位置
cutoff.y = -1,
size.cutoff = 8, # cutoff文字的大小
#============================================
code.highrisk = 'High Risk',# 高风险的label
code.lowrisk = 'Low Risk', # 低风险的lanbel
title.A.ylab='Risk Score', # 图A中y轴的title
title.B.ylab='Survival Time', # 图B中y轴的title
title.A.legend='Risk Group', # 图A的legend的title
title.B.legend='Status', # 图B的legend的title
title.C.legend='Expression', # 图C的legend的title
#============================================
size.ABC = 2, # 标识ABC的大小
color.A = c(low = '#6395C7',high = '#E06EAD'),# 图A的颜色
color.B = c(code.0 ='#6395C7',code.1 = '#E06EAD'), # 图B的颜色
color.C = c(low = "#6395C7",median = 'white',high = '#E06EAD'), # 图C的颜色
vjust.A.ylab=1, # 图A中y轴label到y坐标轴的距离
vjust.B.ylab=2, #图B中y轴label到y坐标轴的距离
relative_heights = c(0.1,0.1,0.01,0.15)# A.B.C三图的相对高度
)
wrap_plots(p1, p2)
点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~
WGCNA | 值得你深入学习的生信分析方法!~
ComplexHeatmap | 颜狗写的高颜值热图代码!
ComplexHeatmap | 你的热图注释还挤在一起看不清吗!?
Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
scRNA-seq | 吐血整理的单细胞入门教程
NetworkD3 | 让我们一起画个动态的桑基图吧~
RColorBrewer | 再多的配色也能轻松搞定!~
rms | 批量完成你的线性回归
CMplot | 完美复刻Nature上的曼哈顿图
Network | 高颜值动态网络可视化工具
boxjitter | 完美复刻Nature上的高颜值统计图
linkET | 完美解决ggcor安装失败方案(附教程)
......
本文由 mdnice 多平台发布