机器学习实战:Python基于KDE核密度估计进行分布估计(十六)
文章目录
-
- 1. 前言
-
- 1.1 KDE简介
- 1.2 KDE应用领域
- 2. diy数据集实战演示
-
- 2.1 导入函数
- 2.2 自定义数据
- 2.3 可视化数据
- 2.4 KDE建模
- 3. 参数探讨
-
- 3.1 带宽
- 3.2 选择最佳带宽
- 3.2 核函数
- 3.4 挑选合适核函数
- 4. 讨论
1. 前言
1.1 KDE简介
核密度估计(Kernel Density Estimation,简称KDE)是用于估计连续随机变量概率密度函数的非参数方法。它的工作原理是在每个数据点周围放置一个“核”(通常是某种平滑的、对称的函数),然后将这些核加起来,形成一个整体的估计。这可以被视为对直方图的平滑,使得得到的密度函数更连续、更平滑。
KDE的主要组件是核函数和带宽。核函数确定了每个数据点对估计的贡献的形状,而带宽决定了核的宽度,影响估计的平滑程度。正确选择这两个组件对于获得有意义的估计至关重要。
优点:
-
非参数性:KDE不假设数据遵循任何特定的概率分布,使其具有很高的灵活性,能够适应多种不同的数据形态。
-
平滑性:与传统的直方图相比,KDE提供的概率密度估计是平滑的,它可以揭示数据中的细微特征而不是粗糙的组块。
-
数据探索:KDE对于初步的数据探索和可视化非常有用,可以帮助我们直观地了解数据的分布和形状。
-
核的选择:KDE允许使用不同的核,例如高斯核、三角核等,这为不同的应用和数据特点提供了选择的灵活性。
缺点:
-
计算复杂性:KDE的计算复杂性可以高,特别是当数据集很大时,因为它需要考虑每个数据点与其他所有数据点的关系。
-
带宽选择:带宽是KDE中最关键的参数。不合适的带宽选择可能导致估计过于嘈杂(过小的带宽)或过于平滑(过大的带宽)。尽管有带宽选择的方法,但它们通常需要额外的计算和调优。
-
边界问题:在数据分布的边界附近,KDE可能不会提供良好的估计。这是因为在边界附近,核函数可能会延伸到数据的实际范围之外,导致边界处的估计偏低。
-
高维数据:随着数据维度的增加,KDE面临“维度灾难”。在高维空间中,数据点之间的距离变得相对更远,这使得密度估计变得更加困难。
1.2 KDE应用领域
核密度估计(KDE)是一种广泛应用的统计方法,它在多个领域都有其应用。以下是一些KDE部分应用领域的具体实例:
- 数据探索和可视化:
考虑一个电商网站,其中的产品有各种价格。为了了解大多数产品的价格范围,数据分析师可以使用KDE来估计价格分布。这不仅能够揭示价格的中心趋势,还可以揭示价格的多模态分布(例如,低端、中端和高端产品可能有明显的价格聚类)。 - 异常检测:
在信用卡欺诈检测中,可以使用KDE来估计正常的交易行为分布,例如交易金额和交易时间。然后,任何明显偏离这个估计的交易都可能被标记为可疑,然后进一步进行调查。 - 地理空间分析:
在城市治安管理中,警察部门可以利用KDE来分析报告的犯罪事件的地理位置,以确定犯罪的“热点”地区。这可以帮助他们更有效地部署警力和资源。 - 图像处理:
在医学图像分析中,KDE可以用于估计某一区域(如肿瘤)的像素强度分布。这有助于区分正常组织和异常组织,并可以用于自动检测和标记潜在的问题区域。 - 生物信息学:
考虑一项研究,旨在分析不同类型的细胞在特定环境下的基因表达。KDE可以用于估计特定基因在各种条件下的表达水平分布,从而帮助研究人员了解基因的激活和抑制模式。 - 市场研究:
一个汽车制造商想要了解其竞争对手车型的价格分布。使用KDE,他们可以揭示市场上的价格段,并据此确定他们自己车型的定价策略。
2. diy数据集实战演示
2.1 导入函数
import numpy as np
import matplotlib.pyplot as plt
from sklearn.neighbors import KernelDensity
from sklearn.model_selection import GridSearchCV
2.2 自定义数据
生成一个数据集,其中数据来自两个不同的分布:一个对数正态分布和一个正态分布。两个分布各生成1000个数据点,并将这些数据点连接在一起:
- 对数正态分布使用均值0和标准差0.3。
- 正态分布使用均值3和标准差1。
def generate_data(rand=None):
if rand is None:
rand = np.random.RandomState()
dat1 = rand.lognormal(0, 0.3, 1000)
dat2 = rand.normal(3, 1, 1000)
x = np.concatenate((dat1, dat2))
return x
data = generate_data(np.random.RandomState(17))
data[:10]
# array([1.08641118, 0.57327576, 1.20583266, 1.41000519, 1.3650037 ,
# 1.76119346, 0.96704574, 0.89706191, 1.04561216, 0.876924 ])
2.3 可视化数据
- 展示数据点的顺序和相对位置
- 展示数据的分布情况
x_train = generate_data()[:, np.newaxis]
fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(10, 5))
ax[0].scatter(np.arange(len(x_train)), x_train, c='red')
ax[0].set_xlabel('Sample no.')
ax[0].set_ylabel('Value')
ax[0].set_title('Scatter plot')
ax[1].hist(x_train, bins=50)
ax[1].set_title('Histogram')
fig.subplots_adjust(wspace=.3)
plt.show()
2.4 KDE建模
x_test = np.linspace(-1, 7, 2000)[:, np.newaxis]
model = KernelDensity().fit(x_train)
log_dens = model.score_samples(x_test)
plt.fill_between(x_test.ravel(), np.exp(log_dens), color='pink')
plt.show()
3. 参数探讨
3.1 带宽
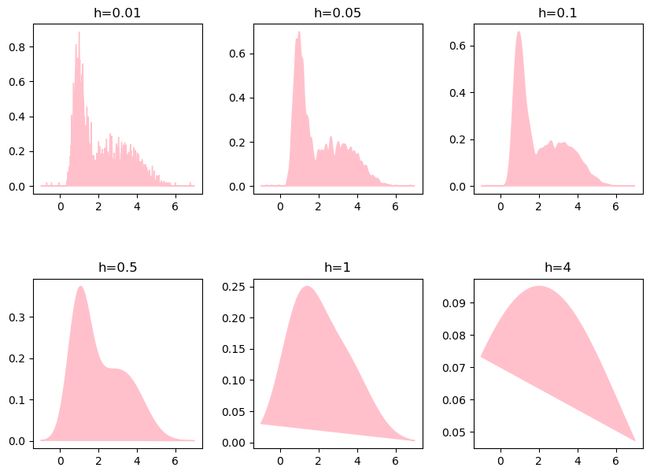
带宽(bandwidth)是核密度估计(KDE)中的一个关键参数,它决定了估计的平滑程度。选择合适的带宽对于获得有意义的密度估计至关重要。
bandwidths = [0.01, 0.05, 0.1, 0.5, 1, 4]
fig, axes = plt.subplots(nrows=2, ncols=3, figsize=(10, 7))
for ax, b in zip(axes.ravel(), bandwidths):
kde_model = KernelDensity(kernel='gaussian', bandwidth=b).fit(x_train)
score = kde_model.score_samples(x_test)
ax.fill(x_test, np.exp(score), c='pink')
ax.set_title(f"h={b}")
fig.subplots_adjust(hspace=0.5, wspace=.3)
plt.show()
3.2 选择最佳带宽
常见的就这四种方法:
-
经验法则:一些常见的方法,如Silverman’s rule,可以为一般情况提供一个好的起点。但这些方法可能不适用于所有数据集。
-
交叉验证:使用交叉验证选择带宽是一种常见方法。这涉及到将数据分成训练集和验证集,然后对于不同的带宽值,使用训练集进行拟合,并使用验证集计算对数似然评分。选择使得评分最大化的带宽值。
-
可视化:像您之前的代码那样,通过为不同的带宽值绘制KDE,可以帮助直观地选择一个合适的带宽。
-
数据的范围和分辨率:带宽的绝对值通常与数据的范围和单位有关。例如,如果数据是以米为单位的距离测量值,那么0.01米的带宽与100米的带宽意味着完全不同的事情。
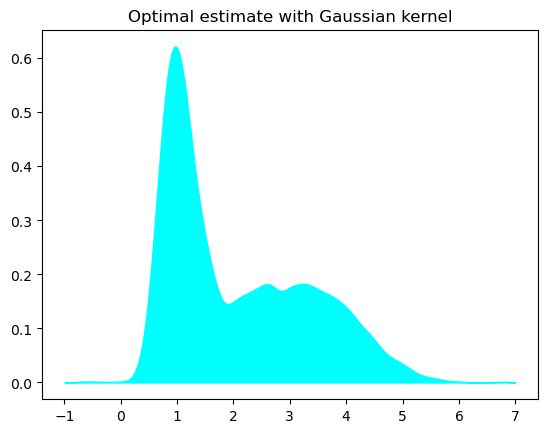
这里用的是交叉验证:
bandwidth = np.arange(0.05, 2, .05)
grid = GridSearchCV(KernelDensity(kernel='gaussian'), {'bandwidth': bandwidth})
grid.fit(x_train)
kde = grid.best_estimator_
log_dens = kde.score_samples(x_test)
plt.fill_between(x_test.ravel(), np.exp(log_dens), color='cyan')
plt.title('Optimal estimate with Gaussian kernel')
plt.show()
print(f"optimal bandwidth: {kde.bandwidth:.2f}")
# optimal bandwidth: 0.15
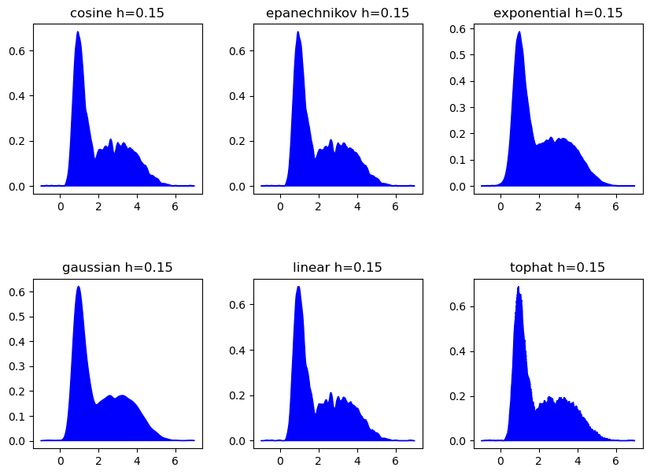
3.2 核函数
| Kernel | Name | Description |
|---|---|---|
| cosine | 余弦 | 形状类似于余弦曲线的一部分,平滑地从中心下降到零。 |
| epanechnikov | Epanechnikov | 这是一个凸的、平滑的、有界的核,常被用作默认核,因为它在某些理论性质上优于其他核。 |
| exponential | 指数 | 这是一个从中心点开始快速下降的核,类似于高斯核,但下降得更快。 |
| gaussian | 高斯或正态 | 这是最常用的核,其形状是标准的正态分布曲线。它为数据点提供了平滑的、无界的权重。 |
| linear | 线性 | 这是一个三角形的核,从中心线性下降到零。 |
| tophat | 平顶帽 | 这是一个矩形的核,它在一个固定的范围内为数据点提供均匀的权重,然后突然下降到零。 |
吐槽一点,“Epanechnikov” 核函数并没有一个广泛接受的中文名字。它是以俄罗斯统计学家Urii Epanechnikov的名字命名的。文献常见被翻译成 “埃潘尼克尼科夫” 核。
kernels = ['cosine', 'epanechnikov', 'exponential', 'gaussian', 'linear', 'tophat']
fig, axes = plt.subplots(nrows=2, ncols=3, figsize=(10, 7))
x_range = np.arange(-2, 2, 0.1)[:, None]
for ax, k in zip(axes.ravel(), kernels):
kde_model = KernelDensity(kernel=k).fit([[0]])
score = kde_model.score_samples(x_range)
ax.fill(x_range, np.exp(score), c='blue')
ax.set_title(k)
fig.subplots_adjust(hspace=0.5, wspace=.3)
plt.show()
3.4 挑选合适核函数
这里直接用刚刚的最佳带宽0.15即可
def my_scores(estimator, X):
scores = estimator.score_samples(X)
scores = scores[scores != float('-inf')]
return np.mean(scores)
kernels = ['cosine', 'epanechnikov', 'exponential', 'gaussian', 'linear', 'tophat']
h_vals = np.arange(0.05, 1, .1)
fig, axes = plt.subplots(nrows=2, ncols=3, figsize=(10, 7))
for ax, k in zip(axes.ravel(), kernels):
grid = GridSearchCV(KernelDensity(kernel=k), {'bandwidth': h_vals}, scoring=my_scores)
grid.fit(x_train)
kde = grid.best_estimator_
log_dens = kde.score_samples(x_test)
ax.fill(x_test.ravel(), np.exp(log_dens), c='cyan')
ax.set_title(f"{k} h={kde.bandwidth:.2f}")
fig.subplots_adjust(hspace=.5, wspace=.3)
plt.show()
还是用交叉验证:
grid = GridSearchCV(KernelDensity(),
{'bandwidth': h_vals, 'kernel': kernels},
scoring=my_scores)
grid.fit(x_train)
best_kde = grid.best_estimator_
log_dens = best_kde.score_samples(x_test)
plt.fill_between(x_test.ravel(), np.exp(log_dens), color='cyan')
plt.title(f"Best Kernel: {best_kde.kernel} h={best_kde.bandwidth:.2f}")
plt.show()
4. 讨论
核密度估计(KDE)是一种非参数方法,用于估计随机变量的概率密度函数。通过在每个观测数据点处放置一个核函数并将它们相加,KDE提供了数据的平滑估计,可以捕捉数据的复杂分布特性,而不受特定参数分布的限制。