RNA结合蛋白研究技术:RIP-seq实验分析流程及案例分享
RNA免疫共沉淀—RIP-seq(RNA Immunoprecipititation)是研究细胞内RNA与蛋白结合情况的技术,RIP利用目标蛋白的抗体将相应的RNA-蛋白复合物(RBP)沉淀下来,分离纯化捕获的RNA,结合高通量测序技术对目标RNA进行测序分析。RIP可以看成是普遍使用的染色质免疫沉淀ChIP技术的类似应用,蛋白结合对象为RNA,RIP-seq即对富集得到的RNA片段进行高通量测序,是了解转录后调控网络动态过程的有力工具,能够有效探究在基因组范围内发现与蛋白或蛋白复合物相结合的RNA。
01 应用方向:
1. 研究细胞内RNA与蛋白结合情况
2. 发现RBP与非编码RNA(LncRNA、miRNA等)的相互作用
3. 绘制全基因组范围的RNA与RBP相互作用图谱
02 实验流程:
1.前期准备
· 需要提供成熟的基因组信息(包括基因组fa文件、gff 文件、pep.fa 文件)。
· 需要提供抗体(对应的WB检测图,若无法提供,可委托爱基百客进行WB检测)。
· 样本:动物组织、植物组织、肿瘤组织、细胞、真菌样品均可以。
· 对照:一般选用Input作为阳性对照;此外,可以设置样本对照(例如,处理前vs处理后)。
· 抗体!抗体!抗体!:首选ChIP级别抗体,至少IP级别;若没有IP级别抗体,也可选择标签抗体(要确保标签和蛋白成功融合表达)。
2.具体步骤
主要步骤包括紫外交联、准备裂解产物、准备免疫沉淀的磁珠、免疫沉淀 RNA连接的蛋白-RNA 复合物、文库构建、上机测序、生信分析等。具体流程如下图所示:
研究对象RNA主要包括mRNA、LncRNA、circRNA、miRNA,针对不同种类RNA选择不同的建库方式,其中mRNA、LncRNA、circRNA一般选择去除rRNA后构建链特异性文库,miRNA一般构建Small RNA文库。
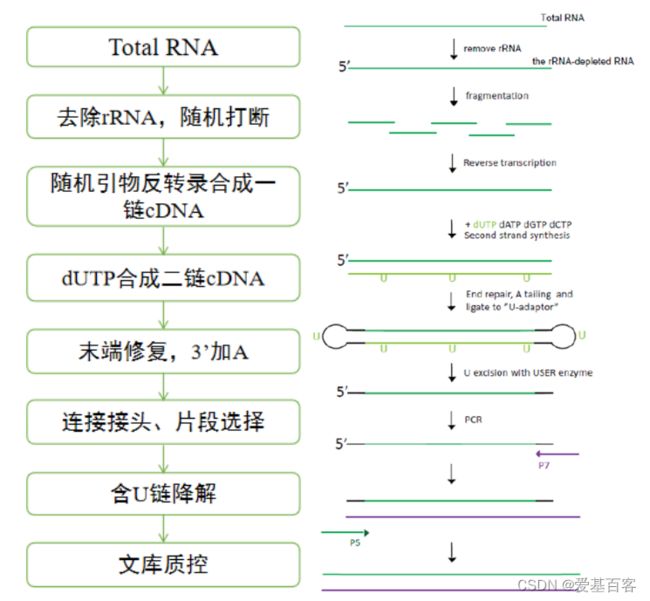
链特异性文库构建流程如下图所示:
链特异性文库实验步骤
链特异性文库:可以保留转录组测序时转录本的方向信息,即可以确定转录本是来源于基因组的正链或负链。
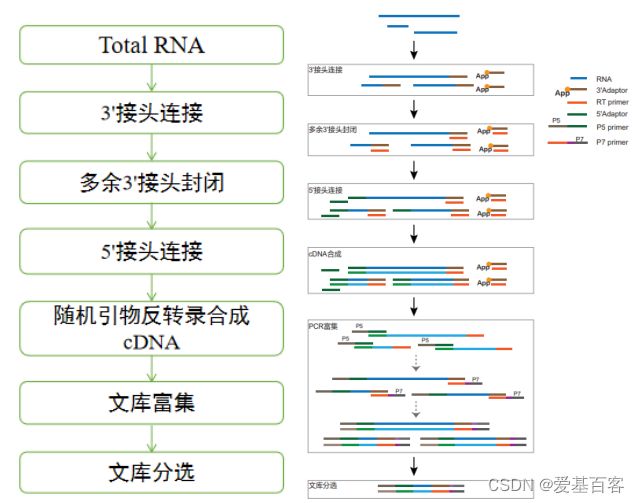
Small RNA文库构建流程如下图所示:
Small RNA文库实验步骤
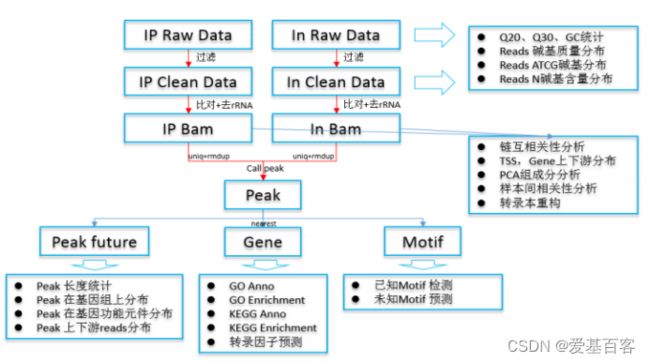
03分析流程
分析结果主要包括基因组比对分析、Reads在基因功能元件上分布、PCA分析、基因组Peak分析、Peak关联基因的GO/KEGG分析、富集区间Motif分析等。(感兴趣的老师可以联系爱基百客相关人员获取结题报告demo)
04多组学联合分析和案例分享
在研究中大多数技术都不是单一使用,RIP-seq也不例外,它会搭配其他组学一起对研究的深度和广度进行扩充。RIP-seq可以与多种技术联合分析,例如RNA-seq、 RNA pull down、m6A-seq等。本次精选几篇联合分析的文章,帮助大家开拓思路。
案例分享一
题目:RIP-Seq of EZH2 Identifies TCONS-00036665 as a Regulator of Myogenesis in Pigs E2H2的RIP-seq将TCONS-OOO36665鉴定为猪肌细胞生成的关键因子
发表单位:华中农业大学
期刊:Frontiers in Cell and Developmental Biology(IF2022=6.081)
相关技术:RIP-seq、RNA pull down、lncRNA-seq、ChIP-seq等(爱基都可提供)
研究内容:EZH2是PCR2的催化亚基,它包含一个SET结构域,可催化赖氨酸 27 (H3K27me3) 上的组蛋白H3三甲基化以产生表观遗传沉默标记。EZH2通过与转录因子或RNA转录本相互作用来实现其功能。在本研究中,作者采用RIP-seq和lncRNA-seq来鉴定EZH2结合的lncRNAs,通过生物信息学分析鉴定出356 个可以结合EZH2的lncRNA,并对其中的TCONS-00036665进行了表征。
TCONS-00036665能促进猪骨骼肌卫星细胞增殖但抑制细胞分化,这种功能在猪和小鼠之间是保守的。进一步的机制研究表明,TCONS-00036665可以与EZH2结合,并将EZH2招募到靶基因 p21、MyoG和Myh4的启动子上,从而富集H3K27me3以及抑制靶基因表达和猪肌细胞生成。
总之,lncRNA TCONS-00036665通过与 EZH2的相互作用调节猪肌细胞的生成。
结合EZH2的LncRNA鉴定
案例分享二
题目:The m6A reader MhYTP2 regulates MdMLO19 mRNA stability and antioxidant genes translation efficiency conferring powdery mildew resistance in apple
m6A阅读蛋白MhYTP2调节MdMLO19 mRNA稳定性和抗氧化基因翻译效率,从而赋予苹果白粉病抗性
发表单位:西北农林科技大学园艺学院
期刊:Plant Biotechnology Journal(IF2022=13.263)
相关技术:RIP-seq、EMSA、m6A-seq、RNA-seq等(爱基都可提供)
研究内容:
研究人员前期获得了RNA结合蛋白MhYTP2超表达转基因苹果植株,并发现转基因植株具有明显的白粉病抗性。作者首先证明了苹果的MhYTP2同样具有m6A结合功能;其次,通过检测转基因材料中m6A甲基化酶和去甲基化酶相关基因的表达水平,发现MhYTP2的超表达会改变m6A甲基化酶和去甲基化酶相关基因的表达。
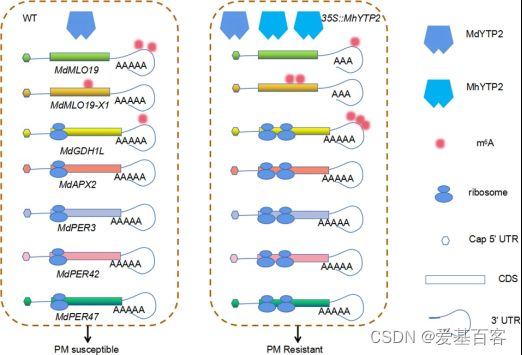
借助于m6A-seq、RNA-seq和Ribo-seq等技术,研究人员发现MhYTP2过表达增加了一批mRNA的m6A修饰水平和翻译效率。同时,外显子区域的m6A修饰水平可能与mRNA的稳定性呈负相关,而非翻译区(UTR)的m6A变化与相关mRNA丰度呈正相关。此外,MhYTP2能够与含m6A修饰白粉病感病基因MdMLO19和MdMLO19-X1的mRNAs结合,并加速其降解;MhYTP2还能够结合谷氨酸脱氢酶1基因MdGDH1L的mRNA,并提高其翻译效率,增加蛋白积累,提高转基因植株的抗氧化能力。
综上所述,该研究结果揭示了苹果m6A基因谱、MhYTP2对m6A基因谱的影响以及m6A在调控苹果白粉病感病基因MdMLO19 mRNA稳定性和抗氧化相关基因翻译效率中的作用。
m6A阅读蛋白调控苹果白粉病抗性分子机制
总 结
本期主要为大家介绍了RIP-seq的技术原理、应用方向、实验流程、案例分享,那么做完RIP-seq之后该如何解读报告呢?这部分内容后期再为大家详细介绍,敬请期待。