逻辑回归L1和L2正则化
正则化
正则化是用来防止模型过拟合的过程,常用的有L1正则化和L2正则化两种选项,分别通过在损失函数后加上参数向 量 的L1范式和L2范式的倍数来实现。这个增加的范式,被称为“正则项”,也被称为"惩罚项"。损失函数改变,基 于损失函数的最优化来求解的参数取值必然改变,我们以此来调节模型拟合的程度。
L1范式表现为参数向量中 的每个参数的绝对值之和,
L2范数表现为参数向量中的每个参数的平方和的开方值。
重要参数penalty & C
| 参数 | 说明 |
| penalty | 可以输入"l1"或"l2"来指定使用哪一种正则化方式,不填写默认"l2"。 注意,若选择"l1"正则化,参数solver仅能够使用求解方式”liblinear"和"saga“,若使用“l2”正则 化,参数solver中所有的求解方式都可以使用 |
| C | C正则化强度的倒数,必须是一个大于0的浮点数,不填写默认1.0,即默认正则项与损失函数的 比值是1:1。C越小,损失函数会越小,模型对损失函数的惩罚越重,正则化的效力越强,参数 会逐渐被压缩得越来越小 |
L1正则化和L2正则化虽然都可以控制过拟合,但它们的效果并不相同。当正则化强度逐渐增大(即C逐渐变小), 参数 的取值会逐渐变小,但L1正则化会将参数压缩为0,L2正则化只会让参数尽量小,不会取到0。
通过建立两个逻辑回归来看L1和L2的正则化差别
1.导入所需库
from sklearn.linear_model import LogisticRegression as LR
from sklearn.datasets import load_breast_cancer
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score2.读取数据并且查看数据维度
data = load_breast_cancer()
X = data.data
y = data.target
data.data.shape3.创建L1,L2并查看每个特征对应参数
lrl1 = LR(penalty="l1",solver="liblinear",C=0.5,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=0.5,max_iter=1000)
#逻辑回归的重要属性coef_,查看每个特征所对应的参数
lrl1 = lrl1.fit(X,y)
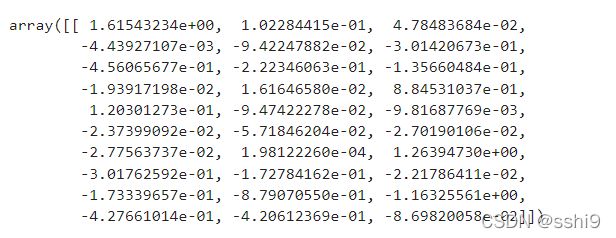
lrl1.coef_
(lrl1.coef_ != 0).sum(axis=1)
lrl2 = lrl2.fit(X,y)
lrl2.coef_l1特征对应参数,很多为0
l2特征对应参数
4.可视化
l1 = []
l2 = []
l1test = []
l2test = []
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420)
for i in np.linspace(0.05,1,19):
lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000)
lrl1 = lrl1.fit(Xtrain,Ytrain)
l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain))
l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest))
lrl2 = lrl2.fit(Xtrain,Ytrain)
l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain))
l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest))
graph = [l1,l2,l1test,l2test]
color = ["green","black","lightgreen","gray"]
label = ["L1","L2","L1test","L2test"]
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.05,1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4) #图例的位置在哪里?4表示,右下角
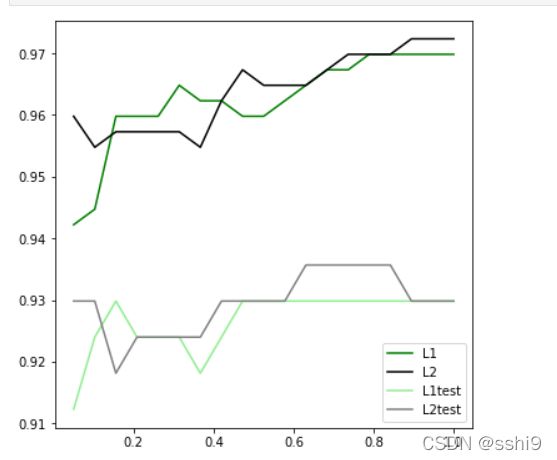
plt.show()结果
结论: 可见,至少在我们的乳腺癌数据集下,两种正则化的结果区别不大。但随着C的逐渐变大,正则化的强度越来越 小,模型在训练集和测试集上的表现都呈上升趋势,直到C=0.8左右,训练集上的表现依然在走高,但模型在未知 数据集上的表现开始下跌,这时候就是出现了过拟合。我们可以认为,C设定为0.8会比较好。在实际使用时,基本 就默认使用l2正则化,如果感觉到模型的效果不好,那就换L1试试看