五、核支持向量机算法(NuSVC,Nu-Support Vector Classification)(有监督学习)
和支持向量分类(Nu-Support Vector Classification),与 SVC 类似,但使用一个参数来控制支持向量的数量,其实现基于libsvm
一、算法思路
本质都是SVM中的一种优化,原理都类似,详细算法思路可以参考博文:三、支持向量机算法(SVC,Support Vector Classification)(有监督学习)
二、官网API
官网API
class sklearn.svm.NuSVC(*, nu=0.5, kernel='rbf', degree=3, gamma='scale', coef0=0.0, shrinking=True, probability=False, tol=0.001, cache_size=200, class_weight=None, verbose=False, max_iter=-1, decision_function_shape='ovr', break_ties=False, random_state=None)
导包:from sklearn.svm import NuSVC
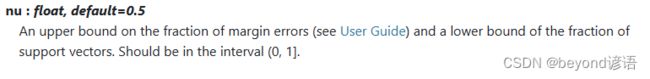
①边际误差分数nu
边际误差分数的上限和支持向量分数的下限,用来控制支持向量的数目和边际误差;nu范围应为(0,1],默认值为0.5
使用方法
NuSVC(nu=0.5)
②核函数kernel
‘linear’:线性核函数,速度快;只能处理数据集样本线性可分,不能处理线性不可分。
‘poly’:多项式核函数,可将数据集样本升维,从低维空间映射到高维空间;参数较多,计算量大
‘rbf’:高斯核函数,和多项式核函数一样,可将样本升维;相较于多项式核函数来说,参数较少;默认值
'sigmoid’:sigmoid 核函数;当选用 sigmoid 核函数时,SVM 可实现的是多层神经网络
‘precomputed’:核矩阵;使用用户给定的核函数矩阵(n*n)
也可以自定义自己的核函数,然后进行调用即可
使用方法
NuSVC(kernel='sigmoid')
③多项式核函数的阶数degree
多项式核函数的阶数;该参数只对多项式核函数(poly)有用;若是其他的核函数,系统会自动忽略该参数
使用方式
NuSVC(kernel='poly',degree=2)
④核系数gamma
rbf、poly 和 sigmoid核函数的核系数,该参数只针对这三个核函数,需要注意
‘scale’:默认值,具体的计算公式看下面的详细官网详情
‘auto’:具体的计算公式看下面的详细官网详情
或者是其他的浮点数均可
使用方式
NuSVC(gamma='auto')
⑤随机种子random_state
如果要是为了对比,需要控制变量的话,这里的随机种子最好设置为同一个整型数
使用方式
NuSVC(random_state=42)
⑥最终构建模型
NuSVC(nu=0.5,kernel=‘rbf’,gamma=‘auto’,random_state=42)
三、代码实现
①导包
这里需要评估、训练、保存和加载模型,以下是一些必要的包,若导入过程报错,pip安装即可
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import joblib
%matplotlib inline
import seaborn as sns
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.svm import NuSVC
from sklearn.metrics import confusion_matrix, classification_report, accuracy_score
②加载数据集
数据集可以自己简单整个,csv格式即可,我这里使用的是6个自变量X和1个因变量Y
![]()
fiber = pd.read_csv("./fiber.csv")
fiber.head(5) #展示下头5条数据信息
![]()
③划分数据集
前六列是自变量X,最后一列是因变量Y
常用的划分数据集函数官网API:train_test_split
![]()
test_size:测试集数据所占比例
train_size:训练集数据所占比例
random_state:随机种子
shuffle:是否将数据进行打乱
因为我这里的数据集共48个,训练集0.75,测试集0.25,即训练集36个,测试集12个
X = fiber.drop(['Grade'], axis=1)
Y = fiber['Grade']
X_train, X_test, y_train, y_test = train_test_split(X,Y,train_size=0.75,test_size=0.25,random_state=42,shuffle=True)
print(X_train.shape) #(36,6)
print(y_train.shape) #(36,)
print(X_test.shape) #(12,6)
print(y_test.shape) #(12,)
④构建NuSVC模型
参数可以自己去尝试设置调整
nusvc = NuSVC(nu=0.5,kernel='rbf',gamma='auto',random_state=42)
⑤模型训练
就这么简单,一个fit函数就可以实现模型训练
nusvc.fit(X_train,y_train)
⑥模型评估
把测试集扔进去,得到预测的测试结果
y_pred = nusvc.predict(X_test)
看看预测结果和实际测试集结果是否一致,一致为1否则为0,取个平均值就是准确率
accuracy = np.mean(y_pred==y_test)
print(accuracy)
也可以通过score得分进行评估,计算的结果和思路都是一样的,都是看所有的数据集中模型猜对的概率,只不过这个score函数已经封装好了,当然传入的参数也不一样,需要导入accuracy_score才行,from sklearn.metrics import accuracy_score
score = nusvc.score(X_test,y_test)#得分
print(score)
⑦模型测试
拿到一条数据,使用训练好的模型进行评估
这里是六个自变量,我这里随机整个test = np.array([[16,18312.5,6614.5,2842.31,25.23,1147430.19]])
扔到模型里面得到预测结果,prediction = nusvc.predict(test)
看下预测结果是多少,是否和正确结果相同,print(prediction)
test = np.array([[16,18312.5,6614.5,2842.31,25.23,1147430.19]])
prediction = nusvc.predict(test)
print(prediction) #[2]
⑧保存模型
nusvc是模型名称,需要对应一致
后面的参数是保存模型的路径
joblib.dump(nusvc, './nusvc.model')#保存模型
⑨加载和使用模型
nusvc_yy = joblib.load('./nusvc.model')
test = np.array([[11,99498,5369,9045.27,28.47,3827588.56]])#随便找的一条数据
prediction = nusvc_yy.predict(test)#带入数据,预测一下
print(prediction) #[4]
完整代码
模型训练和评估,不包含⑧⑨。
from sklearn.svm import NuSVC
import pandas as pd
import numpy as np
from sklearn.model_selection import train_test_split
fiber = pd.read_csv("./fiber.csv")
# 划分自变量和因变量
X = fiber.drop(['Grade'], axis=1)
Y = fiber['Grade']
#划分数据集
X_train, X_test, y_train, y_test = train_test_split(X, Y, random_state=0)
nusvc = NuSVC(nu=0.5,kernel='rbf',gamma='auto',random_state=42)
nusvc.fit(X_train,y_train)#模型拟合
y_pred = nusvc.predict(X_test)#模型预测结果
accuracy = np.mean(y_pred==y_test)#准确度
score = nusvc.score(X_test,y_test)#得分
print(accuracy)
print(score)
test = np.array([[23,97215.5,22795.5,2613.09,29.72,1786141.62]])#随便找的一条数据
prediction = nusvc.predict(test)#带入数据,预测一下
print(prediction)