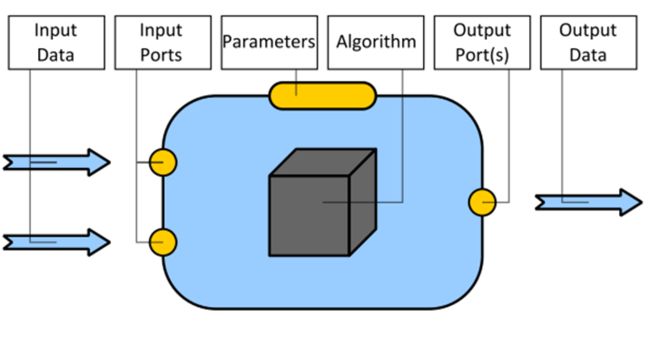
生信工作流框架搭建 | 从零开始入门指南 - 00工作流之华山论剑
本篇为biodoge《生信工作流框架搭建》系列笔记的开篇作,该系列将持续更新。
导语
小白生信工程师一枚,写这样的系列其实是个大工程,出发点很简单,工作是宏基因组相关,我司长期使用的流程pipeline是用perl脚本封装的,流程监控、代码维护不是非常方便。偶然了解到snakemake和nextflow,便开始入坑工作流框架(语言),尝试使用工作流语言改写流程,并在AWS上成功实践。此过程中发现互联网上相关信息较少,而坑又甚多,便将自己的经验教训总结成文,分享给在生信之海遨(zheng)游(zha)的你们。
在正文之前,安利一篇幽默的nature文章:
你用Python我用AWE,工作流程工具能让我们做回朋友吗?
一个实例解释我们为什么需要框架
都说生信人员有那么几个阶段:用生信程序、跑生信流程、写生信算法。
我对此感同身受。学生时代我更多是在用生信的程序,甚至还是在linux的图形化界面上。跑个实验,我可能会选择直接一个个执行命令行。别的不说,每次跑相同的步骤,都要人工操作一遍吗?完全可以自动化。于是便有了“工作流”一说。将标准化的流程写为pipeline,用脚本封装,再多的步骤,一个命令行+参数搞定。输入输出也标准化了,效率大大提高。
举例来说,假如我们开始分析一组包含多个样本的宏病毒组测序数据
前两个步骤包含,
-
读取两对illumina双端数据(fastq.gz格式),QC、去宿主
-
用SPAdes组装
对单个样本,bash代码如下(多个样本,可以写个脚本,每个样本都各自生成一个脚本)
mkdir -p /data/output/01.QC/A1/
java -jar trimmomatic.jar PE -threads 16 -phred33 /data/raw_data/A1_1.fq.gz /data/raw_data/A1_2.fq.gz /data/output/01.QC/A1/A1_trim_1.fq /data/output/01.QC/A1/A1_trim_single.fq_1 /data/output/01.QC/A1/A1_trim_2.fq /data/output/01.QC/A1/A1_trim_single.fq_2 ILLUMINACLIP:/db/trimmomatic-adapters/TruSeq3-PE.fa:2:30:10 LEADING:5 TRAILING:5 SLIDINGWINDOW:4:15 MINLEN:50
cat /data/output/01.QC/A1/A1_trim_single.fq_1 /data/output/01.QC/A1/A1_trim_single.fq_2 > /data/output/01.QC/A1/A1_trim_single.fq
rm -rf /data/output/01.QC/A1/*.pe /data/output/01.QC/A1/*.se /data/output/01.QC/A1/*.single /data/output/01.QC/A1/*.fq_1 /data/output/01.QC/A1/*.fq_2 /data/output/01.QC/A1/*.log
mkdir -p /data/output/01.QC/A1/
soap2 -a /data/output/01.QC/A1/A1_trim_1.fq -b /data/output/01.QC/A1A1_trim_2.fq -D /db/hg38_2bwt_index/hg38.fa.index -o /data/output/01.QC/A1/A1_trim.pe -M 4 -l 32 -r 1 -m 400 -x 600 -2 /data/output/01.QC/A1/A1_trim.se -v 8 -c 0.9 -S -p 16 2> /data/output/01.QC/A1/A1_trimsoap2.pe.log
soap2 -a /data/output/01.QC/A1/A1_trim_single.fq -D /db/hg38_2bwt_index/hg38.fa.index -o /data/output/01.QC/A1/A1_trim.single -M 4 -l 32 -r 1 -v 8 -c 0.9 -S -p 16 2> /data/output/01.QC/A1/A1_trimsoap2.single.log
mkdir -p /data/output/02.assembly/A1/
python spades.py -o /data/output/02.assembly/A1/ --meta -1 /data/input/01.QC/A1/A1_trim_rmhost_1.fq.gz -2 /data/input/01.QC/A1/A1_trim_rmhost_2.fq.gz -t 32
可以想见,仅仅两个步骤这样简单的流程,样本一多,操作起来也是比较麻烦的。
然后我们继续往上,接触到上云、批量计算、HPC,项目一大周期一长,各种问题又接踵而至。不同人员写的脚本不同语言不同怎么维护?是否能够在各种平台上稳定运行?中间报错任务中断了怎么办?任务之间怎么去调度?
生信工作流框架(即工作流管理系统)也就应运而生。
什么是工作流框架
首先我们要清楚工作流的概念。有一篇文章很好地道出了科学工作流的核心:科学工作流是连续的和并发的数据处理任务的组合,其顺序由数据相互依赖性决定;科学工作流程通常以有向无环图(DAG)的形式来描述[1]。
Scientific workflows are compositions of sequential and concurrent data processing tasks, whose order is determined by data interdependencies. A task is the basic data processing component of a scientific workflow, consuming data from input files or previous tasks and producing data for followup tasks or output files (see Figure 1).
A scientific workflow is usually specified in the form of a directed, acyclic graph (DAG), in which individual tasks are represented as nodes.
如果只用一个词来形容工作流,那就是DAG。
各类工作流,无非就是构建一个DAG;而各类工作流框架,本质也就是实现DAG的调度。
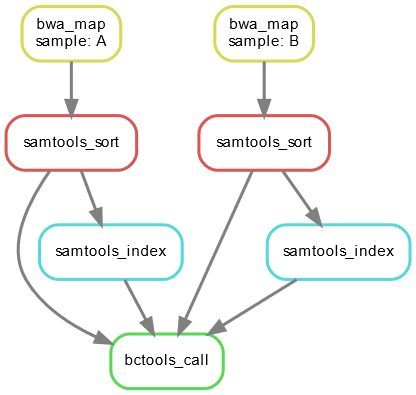
以防万一你不知道工作流的DAG长什么样,没错,这就是任务之间的关系↑
因此,关于工作流框架(也有叫工作流管理系统),一目了然:
constitute four phases of the workflow lifecycle:
(1)the design and composition of concrete workflows;
(2)the mapping of concrete workflows to the underlying physical resources (scheduling);
(3)the execution of physical workflows;
(4)the recording of metadata and provenance information at all stages of the workflow lifecycle.
Scientific workflow management systems (SWfMS) address this lifecycle or subsets thereof by providing capabilities for modeling, executing, monitoring and storing scientific workflows. Most SWfMS operate on concrete workflows by requesting users to specify compositions of concrete processing tasks.
构成工作流生命周期的四个阶段:
(1) 具体工作流的设计和组成;
(2) 将具体工作流映射到底层物理资源(调度);
(3) 物理工作流的执行;
(4) 在工作流生命周期的所有阶段记录元数据和来源信息。
科学工作流管理系统 (SWfMS) 通过提供建模,执行,监视和存储科学工作流的功能来解决此生命周期或其子集
为什么要用工作流框架
工作流之五大流派
一篇大佬综述这样写道:
生物信息学框架应该能够适应由串行和并行步骤、复杂依赖关系、各种软件和数据文件类型、固定和用户定义的参数和可交付成果组成的生产流程。许多现代管道框架提供高级功能,例如用于实时可视化进度的显示、实例化可在任何地方运行的容器化工具的能力、支持在分布式集群或云中执行工作以及允许构建工作流的图形用户界面用户无需编写代码。
框架之间的区别不是功能,而是设计理念。
而根据这篇综述[3],还有snakemake的论文[4],以及网络资源[5],我将现有工作流分为五大流派:

下面将对五大流派做一简介。
1、图形界面派
该流派非常强调易用性。它们提供了用于工作流设计和执行监视的图形用户界面,以及一系列通用的任务库。尽管其中一些系统使用脚本工作流语言进行内部表达,但该语言并非旨在并设计为用户访问。明显的优势是学习曲线较浅,使每个人都可以使用此类系统,而无需编程技能。

![]()
2、脚本语言派
工作流是使用一套通用编程语言(如Python、Scala)的类和函数来指定的。 此类系统的优势在于它们可以在没有图形界面的情况下使用(例如在服务器环境中),并且可以使用 Git 等版本控制系统直接管理工作流。

示例:Anduril数据分析和集成框架的更新版本,许多常见的预处理和下游分析步骤已封装在以各种支持语言(R,Matlab,Python,Java)编码的组件中。组件被组织成专用于特定数据集的捆绑包,并在Anduril引擎运行的Scala程序中组合成管道。Anduril构建一个图并并行处理管道任务的执行,同时跟踪不同步骤的更改和状态(通过/失败),以确保可重入性
3、DSL派(领域特定语言)
工作流是使用领域特定语言(DSL)指定的。DSL提供了专门为工作流管理的核心组件建模的语句和声明,从而避免了多余的运算符或样板代码。对于Nextflow和Snakemake,由于DSL是作为通用编程语言(Groovy和Python)的扩展来实现的,因此可以访问底层编程语言的全部功能(例如,用于实现条件执行和处理配置)

示例:snakemake
4、配置文件派
工作流规范通过像YAML这样的配置文件格式,以纯粹的声明方式进行。 工作流规范对于非开发人员来说特别易读。 缺点是,由于不允许命令式或函数式编程,这些工作流系统可能在可以表达的过程中受到更多限制。
5、通用语言派
独立于系统的工作流规范语言,如 CWL 和 WDL。 这些定义了用于指定工作流的声明性语法,可以由执行器解析和执行,例如 cromwell、Toil 和 Tibanna。主要优点是可以在各种专门的执行后端上执行相同的工作流定义,从而保证几乎可以扩展到任何计算平台。缺点是命令式或函数式编程没有或很少集成到规范语言中,从而限制了表达能力。
另一个重要用途是它们促进了其他工作流定义语言之间的互操作性。 例如,Snakemake工作流程可以(在限制范围内)自动导出到 CWL,并且 Snakemake可以使用 CWL 工具定义。

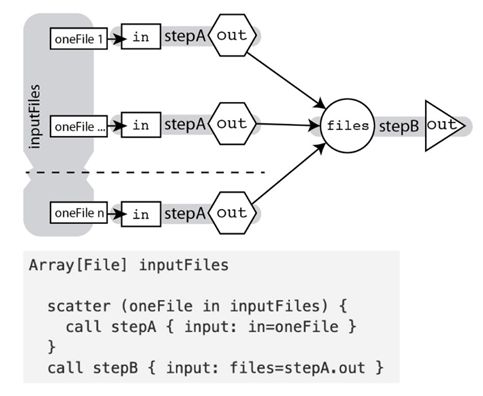
示例:WDL
近年来一些小门派也有声名鹊起之势,如Jupyter notebook和R markdown流,但篇幅有限,不再一一赘述。
工作流之华山论剑
这五大流派、几百种语言,孰胜孰负?争端由来已久。
Biostar开了几十层楼的帖子:Which Bioinformatic Friendly Pipeline Building Framework?
一些综述也给出了他们的答案:

实际上,选择什么样的流程要取决于你的目的,
作者的建议是:
- 如果是完全湿实验且没有时间去学习编程语言的生物研究者,那么可以使用Galaxy这类纯图形界面操作的框架,在完成分析的逻辑构建后就可以高效地进行分析了;
- 如果实验室做的是探索性的概念证明类工作(exploratory proofs-of-concept),那么需要的是 DSL-based pipeline,如:Snakemake、Nextflow等
- 如果是需要进行高性能流程开发,致力于解决特定的生物学问题,且有一定的计算机编程基础的话,那么可以使用Class-based流程。
而对于产业界跑pipeline来说,我们的需求是稳定、易维护、支持云计算和HPC。而已有标准化流程,只需要将脚本流程迁移到框架中。
根据实践经验,归纳出了工作流框架的评估指标:
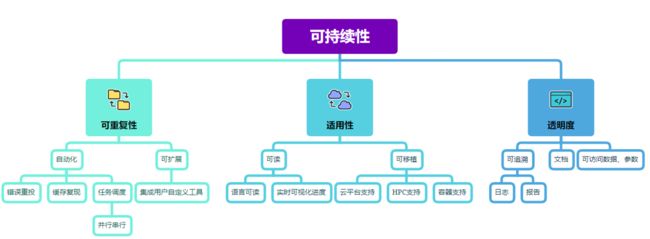
下一篇,将为大家展现我们的实测评估结果(snakemake、nextflow、WDL)。
欢迎关注本专栏。如果有更好的建议和意见,也欢迎评论区反馈^ ^
参考资料
[1] Bux, M. & Leser, U. Parallelization in scientific workflow management systems. Preprint at https://arxiv.org/abs/1303.7195 (2013).
[2] 标准流程描述语言 WDL 阿里云最佳实践
[3] Jeremy Leipzig, A review of bioinformatic pipeline frameworks, Briefings in Bioinformatics, Volume 18, Issue 3, May 2017, Pages 530–536, https://doi.org/10.1093/bib/bbw020citations:203
[4] Mölder F, Jablonski KP, Letcher B et al. Sustainable data analysis with Snakemake [version 2; peer review: 2 approved]. F1000Research 2021, 10:33 (https://doi.org/10.12688/f1000research.29032.2)
[5] 生信分析流程构建的几大流派