增强负样本提高CPI表现
确定CPI对药物发现至关重要。由于实验验证CPI通常是耗时和昂贵的,计算方法有望促进这一过程。可用CPI数据库的快速增长加速了许多用于CPI预测的机器学习方法发展。然而,它们的性能,特别是它们对外部数据的泛化性,经常受到数据不平衡的影响,这是由于缺乏经过实验验证的负样本。该研究开发了一种自训练方法,用于增加更可信和信息丰富的负样本,以提高受数据失衡影响的模型性能。所构建的模型在解决数据不平衡问题上表现出了比其他传统方法更高的性能,并且对于外部数据集的提高尤为突出。此外,对自训练过程中伪标记的预测分数阈值的检验表明,增加具有模糊预测分数的样本有利于构建具有高泛化性的模型。该研究为改进CPI对真实世界数据的预测提供了指导,从而促进药物发现。
来自:Improving Compound-Protein Interaction Prediction by Self-Training with Augmenting Negative Samples
项目:https://github.com/clinfo/kMoL-ST
目录
- 背景概述
- 数据集
- 方法
-
- 自训练方法
- CPI模型
- 评估
背景概述
CPI的鉴定对药物发现具有重要意义(特别是在药物开发的早期阶段),实验性的方法通常既费时又昂贵,因此计算方法有望加速药物发现中CPI的识别。计算方法大致分为两类:基于结构和无结构(基于化学-基因组学,知识图谱,也包括2D分子图-蛋白质序列交互)的方法。基于结构的方法通过从分子对接模拟生成的3D模型中计算基于物理化学的分数来评估CPI。当target蛋白的可靠3D结构可用时,无需已知相互作用的先验信息即可应用对接方法。它们的性能在很大程度上取决于评分函数的准确性和预测的对接姿态。无结构方法主要是化学信息学方法,利用已知CPI的先验信息来预测未知的相互作用。当先验信息覆盖足够的药理学空间时,它们可以以相对较低的计算成本准确地预测CPI。
近年来,公开可用的CPI数据库,如ChEMBL、BindingDB、PubChem、DrugBank和PDBbind迅速增长,使用深度学习技术的CPI预测模型,如CNN、GCN和Transformer,已经显示出显著提高的预测性能和可解释性。这些模型可以在端到端相互作用的过程中提取化合物和蛋白质的特征表示。
使用ML技术的无结构方法的性能经常受到CPI数据库中可用的已知交互产生的训练数据质量的阻碍(因为需要学习训练数据中的知识,缺少负样本会影响学习)。在许多情况下,公共数据库中缺乏经过实验验证的负样本,这导致可用CPI数据中的类别极度不平衡。为弥补负样本的不足,random pairing方法受到关注(也称为random negative)。random pairing方法在以前的研究中被使用过,这种方法随机地从没有实验证实相互作用的化合物-蛋白质pair中产生负样本。然而,提取的样本可能含有潜在的活性(即正样本),导致可信度低。为了解决这个问题,Liu等人提出了一种生成高可信度负样本的方法,使用系统筛选框架来选择远离阳性CPI的样本。然而,最近的一项研究表明,与正样本距离过远的负样本在模型训练中"容易"学习,并且模型在外部数据集上的泛化性能明显低于random negative模型。
为了克服这些限制,提出了一种新的自训练方法来有效地增加负样本。将该方法应用于基于graph的CPI预测模型,成功地提高了模型的性能,包括泛化性。具体来说,用此方法训练的CPI预测模型在外部数据集上比其他解决类别不平衡的方法表现出更高的鲁棒性。此外,对此方法中参数的检验表明,用near-boundary分数预测的伪标记样本比容易归类为阴性的样本更有利于模型的泛化。
数据集
用于构建二元分类模型并评价其性能的数据集来源于ChEMBL。验证泛化性的外部数据集由BioPrint,Davis和BindingDB构建。阳性和阴性标签标注阈值设置为10μM(描述化合物的浓度单位)。在这个阈值上,ChEMBL和BindingDB数据集主要由阳性相互作用组成,而BioPrint和Davis数据集包含大量实验验证的阴性样本。本研究采用G蛋白偶联受体(GPCR,G protein-coupled receptor)和激酶家族(kinase families)的CPI数据。下面描述了这四个数据集的细节,表1总结了它们的统计数据。

- 表1:ChEMBL,BioPrint,Davis和BindingDB统计数据。
ChEMBL
针对人体蛋白的活性数据收集自ChEMBL。该研究用IC50、Ki、EC50、Kd作为standard type,这些标准类型用于描述化合物与蛋白质之间的相互作用,例如药物的亲和力、抑制效应等:
- “IC50”(半数最大抑制浓度):表示在一定浓度下,化合物能够抑制蛋白质活性的程度。
- “Ki”(抑制常数):表示化合物与蛋白质结合的亲和力。
- “EC50”(半数最大效应浓度):表示在一定浓度下,化合物能够引起蛋白质活性的半数最大效应。
- “Kd”(解离常数):表示化合物与蛋白质结合后的解离速率常数。
另外,protein type描述了与 CPI 相关的蛋白质的类型或分类,而assay type描述了用于测定 CPI 数据的实验方法或测定类型。在该研究中,数据集中的蛋白质是单一类型的,测定类型为 “B”。
- “protein type”(蛋白质类型):指的是与 CPI 相关的蛋白质的类型或分类。在该研究中,“single protein” 表示所提取的 CPI 数据集中的蛋白质是单一类型的,即每个相互作用与一个特定类型的蛋白质有关。这可能意味着这些蛋白质属于同一家族、同一类别或具有相似的性质。
- “assay type”(测定类型):指的是用于测定 CPI 数据的实验方法或测定类型。在这里,“B” 被用作描述所提取的 CPI 数据集中的测定类型的标识符。
从预处理的ChEMBL数据集中提取GPCR和激酶数据集,这些数据集基于从Swiss-Prot获得的家族名称。pChEMBL 值≥ 5(活性≤ 10 μM)和<5(活性> 10 μM)的数据点分别标记为阳性和阴性。其中,pChEMBL被定义为-log(molar IC50, XC50, EC50, AC50, Ki, Kd, 或 Potency)。
BioPrint
针对GPCR家族的真实筛选数据来自BiopPrint,其中包含10 μM剂量下抑制活性的实验数据。如果在10 μM的抑制率为50%或更高,则认为CPIs呈阳性:
- 常情况下,如果一个化合物以一定浓度(比如 10 μM)能够抑制蛋白质的活性,使其活性降低到50%或更低的水平,那么这个化合物通常被认为是具有生物活性的,也就是对蛋白质呈现出一定程度的抑制作用。
- 这个标准是为了筛选具有潜在药物候选物特性的化合物而设定的。如果一个化合物以相对较低的浓度就能够显著抑制目标蛋白质的活性,那么它可能对治疗与该蛋白质相关的疾病具有潜在的价值。
Davis
针对激酶家族的真实筛选数据来自Davis数据集,该数据集由68种药物和442种激酶蛋白的结合亲和力信息和解离常数(Kd)值组成。这个版本与DeepDTA(https:// github.com/hkmztrk/DeepDTA/)的Github存储库中的版本相同。如果Kd < 10 μM,pair样本被标注为阳性,如果Kd = 10 μM,pair样本被标注为阴性(弱或无活性)。
BindingDB
正样本多于负样本的外部数据集来自BindingDB。针对人体蛋白的活性数据根据IC50值收集,用和ChEMBL相同的方法提取并标记为两类。
方法
自训练方法
自训练从训练一个有标记数据的teacher模型开始。教师模型通过预测未标记的数据来生成伪标签。将选择的伪标记数据添加到训练数据后,使用更新后的数据训练学生模型。这个过程将使用学生模型作为下一个教师模型进行迭代。图1说明了所提出的使用自训练方法的工作流程。
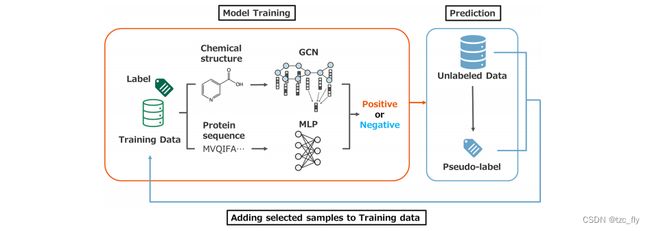
- 图1:自训练工作流程。
步骤1:建立Teacher
Teacher模型 f T ( x ) f_{T}(x) fT(x)通过在有标签数据 { ( x 1 , y 1 ) , . . . , ( x n , y n ) } \left\{(x_{1},y_{1}),...,(x_{n},y_{n})\right\} {(x1,y1),...,(xn,yn)}上最小化BCE loss训练: 1 n ∑ i n B C E ( y i , f T ( x i ) ) \frac{1}{n}\sum_{i}^{n}BCE(y_{i},f_{T}(x_{i})) n1i∑nBCE(yi,fT(xi))其中,BCE被定义为: B C E ( y i , y ^ i ) = y i ⋅ l o g ( y ^ i ) + ( 1 − y i ) ⋅ l o g ( 1 − y ^ i ) BCE(y_i,\widehat{y}_{i})=y_i\cdot log(\widehat{y}_{i})+(1-y_i)\cdot log(1-\widehat{y}_i) BCE(yi,y i)=yi⋅log(y i)+(1−yi)⋅log(1−y i)
步骤2:预测Unlabeled数据
Teacher模型 f T ( x ) f_{T}(x) fT(x)被用于为未标记数据 { x ~ 1 , . . . , x ~ n } \left\{\widetilde{x}_{1},...,\widetilde{x}_{n}\right\} {x 1,...,x n}生成伪标签。如果 x ~ i \widetilde{x}_{i} x i满足 ϕ ≤ f T ( x ~ i ) ≤ 0.5 \phi\leq f_{T}(\widetilde{x}_{i})\leq 0.5 ϕ≤fT(x i)≤0.5,则 x ~ i \widetilde{x}_{i} x i被视为伪负样本,其中阈值参数 ϕ ∈ [ 0 , 0.5 ) \phi\in[0,0.5) ϕ∈[0,0.5)。
步骤3:添加样本
伪标签的负样本被添加到训练数据中(只添加负样本)。
步骤4:建立Student模型
Student模型 f S ( x ) f_{S}(x) fS(x)通过在新的训练数据上最小化BCE loss训练: 1 n ∑ i n B C E ( y i , f S ( x i ) ) + 1 m ∑ i m B C E ( y ~ i , f S ( x ~ i ) ) \frac{1}{n}\sum_{i}^{n}BCE(y_{i},f_{S}(x_{i}))+\frac{1}{m}\sum_{i}^{m}BCE(\widetilde{y}_{i},f_{S}(\widetilde{x}_{i})) n1i∑nBCE(yi,fS(xi))+m1i∑mBCE(y i,fS(x i))由于只添加负样本,其中 y ~ i = 0 \widetilde{y}_i=0 y i=0。
步骤5:持续训练
用Student模型代替Teacher模型,返回步骤2。
在CPI预测中, x = ( c , p ) x=(c,p) x=(c,p)表示化合物-蛋白质pair(compound-protein)。未标记数据 { x ~ 1 , . . . , x ~ n } \left\{\widetilde{x}_{1},...,\widetilde{x}_{n}\right\} {x 1,...,x n}由化合物与靶蛋白随机配对生成。这些化合物来源于预处理的ChEMBL数据集(317,244个化合物)。在每次迭代中,从所有未标记的数据中随机抽取拟标记的数据子集。将GPCR和激酶数据集的样本大小分别设置为750和500 K。对于步骤2中的参数 ϕ \phi ϕ,设为0.2。如果每个靶蛋白的负样本量达到相同的正样本数或迭代次数达到预定最大值,则终止迭代。将最大迭代次数设置为9。
CPI模型
使用二元分类模型来预测CPI,以确定给定的化合物和蛋白质是否相互作用。利用kMoL (https://githubcom/elix-tech/kmol)构建了多模态神经网络模型,这是一个基于GNN的开源化学信息学库,可以使用多种输入特征组合各种架构。该研究主要采用分子图作为化合物的输入,Bag-of-Words作为蛋白质序列的输入。kMoL的输出由一个sigmoid函数激活,得到0到1的分数。关于模型架构和模型训练的其他详细信息请参见支持信息 Text S1,表S1−S3。
Text S1:CPI模型构建
CPI预测的kMoL管道分为三个部分:化合物模块、蛋白质模块和相互作用模块。
化合物模块:
一个化合物被表示成一个无向图,其中每个原子是一个节点,每个键是一条边。分子被转换成具有45-dim节点特征、12-dmi边特征和一个邻接矩阵的Graph表示,并通过三个图卷积网络GCN层。表S1和表S2给出了节点和边特征的描述。使用Local Extreme Convolution作为GCN架构,使用包含1024个unit的隐藏层,使用ReLU作为激活函数。此外,使用RDKit计算的17个属性通过MLP网络传递,输出层与GCN readout连接,并通过输出大小为128-dim的MLP网络传递。表S3显示了17种分子性质的总结。
蛋白质模块:
蛋白质序列被编码成n-gram词袋特征,其中最多n个氨基酸的出现被计数。由于有21种氨基酸,包括"Otherwise",因此可能的n-gram总数为 21 + 2 1 2 + ⋅ ⋅ ⋅ + 2 1 n 21 + 21^2 +\cdot\cdot\cdot+ 21^n 21+212+⋅⋅⋅+21n。选择 n = 3 n=3 n=3,并将每个序列编码为9723-dim向量,然后通过具有256-dmi隐藏层和128-dmi输出大小的MLP网络。
相互作用模块:
相互作用模块将化合物模块和蛋白质模块的输出组合成一个256-dim的向量。该层通过具有64-dim隐藏层和输出大小为1-dim的MLP网络传递。输出最终由一个sigmoid函数激活,产生一个从0到1的分数。
- 表S1:节点特征
- 表S2:边特征
- 表S3:RDKit计算的分子属性
评估
提出的方法通过使用ChEMBL数据集的5折交叉验证进行内部评估。ChEMBL数据集随机分为5个fold,以便根据每个靶蛋白的样本量对训练和测试分割进行分层。随机选择20%的训练数据作为验证数据。然后,在外部数据集Davis(激酶)、BiopPrint(GPCR)和BindingDB (GPCR和激酶)上评估了模型的性能。用ROC-AUC和PR-AUC进行评价。ROC曲线绘制了真阳性率和假阳性率的对比图,PR曲线绘制了准确率和召回率的对比图。
将提出的方法与其他四种处理类失衡的方法以及训练原始不平衡数据的基线模型的有效性进行了比较(Weighted loss,Random Undersampling,Random Negative,Similarity Controlled,没有优化的CPI模型即baseline)。


