运动想象 EEG 信号分析
基于运动想象的公开数据集:Data set IVa (BCI Competition III)1
数据描述参考前文:https://blog.csdn.net/qq_43811536/article/details/134224005?spm=1001.2014.3001.5501
本文使用公开数据集 Data set IVa 中的部分被试数据,数据已公开可以从网盘获取:
链接:https://pan.quark.cn/s/5425ee5918f4
提取码:hJFz
目录
- 1. 实验介绍
- 2. EEG 信号分析
-
- 2.1 EEG 的时域特征
- 2.2 EEG 的频域/时频特征
-
- 2.2.1 频域特征
- 2.2.2 时频特征
- 2.3 EEG 的空域(电极位置)特征
- 3. 分析代码
1. 实验介绍
本任务的实验数据来自一名健康受试者,代号al。受试者在视觉提示出现后3.5s内完成以下3个运动想象中的一个:(L)左手,(R)右手,(F)右脚。分类任务中的数据只包括了右手和右脚两类,共280个试次。实验过程中使用脑电帽记录了118个通道的EEG信号,电极位置如图1所示。采集到的EEG信号首先经过带通滤波(0.05-200Hz),再经过数字化和下采样,得到采样率为100Hz的信号。
2. EEG 信号分析
2.1 EEG 的时域特征
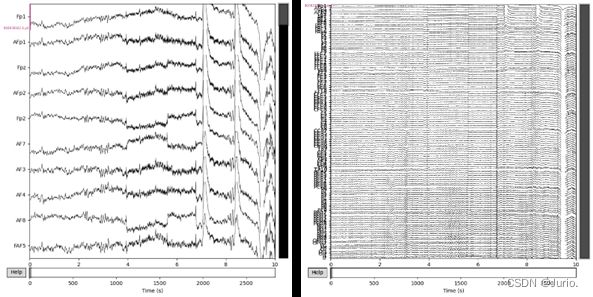
我们首先绘制EEG的时域图,图2(a)和图2(b)分别展示了10个通道和所有通道10s内的信号,可以观察到EEG信号的随机性较强。
2.2 EEG 的频域/时频特征
2.2.1 频域特征
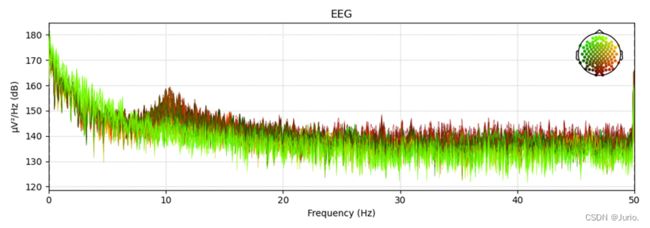
EEG信号是随机信号,通常使用功率谱描述其频域特征。我们截取了100s的EEG信号,使用welch算法估计其功率谱,结果如图3所示。图4则将功率谱按照各电极的位置进行了排列。可以看出:(1)10Hz左右具有明显的峰值,即alpah波;(2)相邻通道的功率谱具有较高的一致性;(3)不同脑区的功率谱具有较大的差异,Alpha波主要出现在枕叶和顶叶区域。
2.2.2 时频特征
EEG信号具有非平稳的特点,因此需要同时考虑时域和频域。这里,为了更加精细地观察,我们对提示出现后0.5s至3.5s的信号进行分析,并将频率范围限制在12Hz至28Hz。使用Morlet小波变换进行时频域分析,得到的结果如图5所示。可以看出,频率变化在13Hz左右较明显,且枕叶的变化尤为明显。
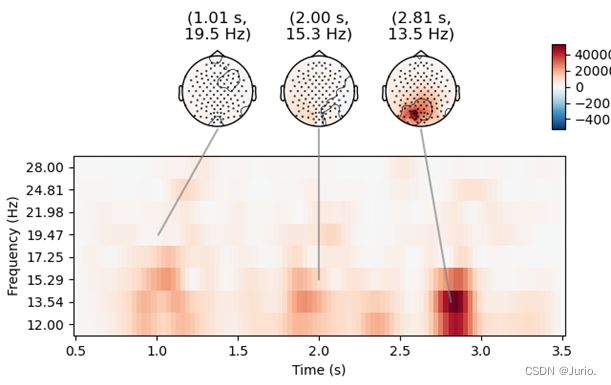
2.3 EEG 的空域(电极位置)特征
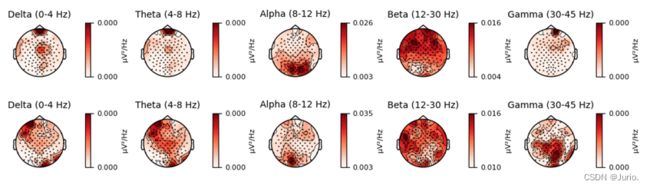
进一步,我们从空域的角度分析EEG,计算两次运动想象期间的功率谱并绘制了5个典型频段的功率谱密度拓扑图,如图6所示。
3. 分析代码
- 主要使用Python的MNE包进行分析
- MNE Tutorials 官网:https://mne.tools/stable/auto_tutorials/index.html
- 部分变量说明:
raw:由 mne.io.RawArray() 函数创建,代表原始EEG数据epochs:由 mne.Epochs() 函数创建,代表一个事件(event)对应的所有数据,在该数据集中一个事件即 “右手”或者“脚”的想象运动
# time domain
raw.crop(tmax=100)
# plot electrode position
plt.figure(figsize=(4,6))
raw.plot_sensors(title='Channel positions')
plt.savefig('Results\sensors.png')
plt.show()
# plot raw eeg data
plt.figure(figsize=(4,6))
raw.plot(n_channels=10, scalings='auto', title='Raw EEG Signals')
plt.show()
plt.figure(figsize=(4,6))
raw.plot(n_channels=118,scalings='auto', title='Raw EEG Signals')
plt.show()
================== plot frequency spectrum
raw.compute_psd().plot()
plt.tight_layout()
plt.savefig('Results\spectrum.png')
plt.show()
raw.compute_psd().plot_topo(color="k", fig_facecolor="w", axis_facecolor="w")
plt.tight_layout()
plt.savefig('Results\spectrum_all.png')
plt.show()
============================ plot time-frequency features
freqs = np.logspace(*np.log10([12, 28]), num=8)
n_cycles = freqs / 2.0 # different number of cycle per frequency
power, itc = mne.time_frequency.tfr_morlet(
epochs,
freqs=freqs,
n_cycles=n_cycles,
use_fft=True,
return_itc=True,
decim=3,
n_jobs=None,
)
power.plot_joint(
baseline=(0, 0.5), mode="mean", tmin=0, tmax=3.5,
timefreqs=[(0.5, 13), (1, 13),(2.8, 13)]
)
plt.show()
epochs[right_id].compute_psd().plot_topomap(ch_type="eeg", agg_fun=np.mean, normalize=True)
plt.savefig(r'Results\right_compute_psd_plot_topomap.png')
plt.show()
epochs[foot_id].compute_psd().plot_topomap(ch_type="eeg", agg_fun=np.mean, normalize=True)
plt.savefig(r'Results\foot_compute_psdplot_topomap.png')
plt.show()
https://bbci.de/competition/iii/desc_IVa.html ↩︎