逻辑回归-癌症病预测与不均衡样本评估
1.注册相关库(在命令行输入)
pip install scikit-learn
pip install pandas
pip install numpy2.导入相关库
import pandas as pd
import numpy as np
from sklearn.metrics import classification_report
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression3.读取数据文件,即癌症数据集
#库读取远程的 CSV 文件
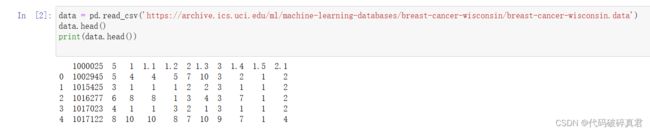
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data')
data.head()
print(data.head())
#给列名字
names = ['Sample code number', 'Clump Thickness', 'Uniformity of Cell Size', 'Uniformity of Cell Shape','Marginal Adhesion',
'Single Epithelial Cell Size', 'Bare Nuclei', 'Bland Chromatin','Normal Nucleoli', 'Mitoses', 'Class']
#给data增加一个names参数
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data',names = names)
data.Class#说明:2表示良性,4表示恶性
print(data['Class'])names列名依次为:
Sample code number:样本编号
Clump Thickness:肿块厚度
Uniformity of Cell Size:细胞大小的均匀性
Uniformity of Cell Shape:细胞形状的均匀性
Marginal Adhesion:边缘粘附
Single Epithelial Cell Size:单个上皮细胞大小
Bare Nuclei:裸核
Bland Chromatin:平淡的染色质
Normal Nucleoli:正常的核仁
Mitoses:有丝分裂
Class:肿瘤类型(良性或恶性)
4.数据清洗(替换空值)
#替换缺失值
data = data.replace(to_replace='?',value=np.nan)
## 删除缺失值的样本
data = data.dropna() #删除有np.nan的行5.进行训练
训练集(x_train,y_train):训练集是用于训练机器学习模型的数据集。通常,我们会利用训练集中的已知样本(包括特征值和目标值)来训练模型,并通过优化模型参数来使其适合数据。
测试集(x_test,y_test):测试集是用于评估机器学习模型性能的数据集。测试集通常由未出现在训练集中的样本组成,用于测试模型是否能够正确推断或预测样本的目标值。
#特征值
x = data.iloc[:,:-1]
x.head()
#目标值
y = data['Class']
y.head()
#表示将数据集中的 20% 作为测试集,剩下的 80% 作为训练集
#:x_train 是训练集的特征值,x_test 是测试集的特征值,y_train 是训练集的目标值,y_test 是测试集的目标值
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.2)
transform = StandardScaler()#实例化转换器
#标准化
#标准化的目的是将不同尺度和单位的特征值转换为具有统一标准的值,以保证模型能够更好地学习和预测
# 通过标准化,可以使特征值的均值为0,标准差为1,从而使得特征值在相同的尺度范围内,避免不同特征值之间的偏差对模型造成影响,使得不同特征之间可以进行可比较的比较。。
x_train = transform.fit_transform(x_train)
x_test = transform.fit_transform(x_test)
mode= LogisticRegression()#用默认的就行
mode.fit(x_train,y_train)#得到了模型
y_predict = mode.predict(x_test)6.模型评估:
#print("y_predict:\n", y_predict)
print("直接比对真实值和预测值:\n", y_test == y_predict)
# 计算准确率
accuracy = mode.score(x_test, y_test)
print("准确率为:\n", accuracy)
#输出混淆矩阵
cm = confusion_matrix(y_test, y_predict, labels=[2, 4])
print("混淆矩阵:\n",cm)
# 打印分类报告
res = classification_report(y_test, y_predict, labels=[2, 4], target_names=['良性', '恶性'])
print(res)
混淆矩阵:
当评估一个分类模型的性能时,准确率(precision)、召回率(recall)和F1值是常用的指标。这些指标可以帮助我们理解模型在不同类别上的预测质量。
准确率(Precision)是指模型在所有被分类为正例的样本中,正确预测为正例的比例。准确率告诉我们被模型预测为正例的样本有多少是真正的正例。它的计算公式如下:
Precision = TP / (TP + FP)
召回率(Recall)是指在所有实际为正例的样本中,模型正确预测为正例的比例。召回率告诉我们模型有多少能够捕捉到真正的正例。它的计算公式如下:
Recall = TP / (TP + FN)F1值是综合考虑了准确率和召回率的指标,它是准确率和召回率的加权调和平均值。F1值的计算公式如下:
F1 = 2 * (Precision * Recall) / (Precision + Recall)7.完整代码:
import pandas as pd
import numpy as np
from sklearn.metrics import classification_report
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression
#库读取远程的 CSV 文件
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data')
data.head()
print(data.head())
#给列名字
names = ['Sample code number', 'Clump Thickness', 'Uniformity of Cell Size', 'Uniformity of Cell Shape','Marginal Adhesion',
'Single Epithelial Cell Size', 'Bare Nuclei', 'Bland Chromatin','Normal Nucleoli', 'Mitoses', 'Class']
#给data增加一个names参数
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data',names = names)
#print(data.head())
data.Class#说明:2表示良性,4表示恶性
#print(data['Class'])
#替换缺失值
data = data.replace(to_replace='?',value=np.nan)
## 删除缺失值的样本
data = data.dropna() #删除有np.nan的行
#特征值
x = data.iloc[:,:-1]
x.head()
#目标值
y = data['Class']
y.head()
#表示将数据集中的 20% 作为测试集,剩下的 80% 作为训练集
#:x_train 是训练集的特征值,x_test 是测试集的特征值,y_train 是训练集的目标值,y_test 是测试集的目标值
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.2)
transform = StandardScaler()#实例化转换器
#标准化
#标准化的目的是将不同尺度和单位的特征值转换为具有统一标准的值,以保证模型能够更好地学习和预测
# 通过标准化,可以使特征值的均值为0,标准差为1,从而使得特征值在相同的尺度范围内,避免不同特征值之间的偏差对模型造成影响。
x_train = transform.fit_transform(x_train)
x_test = transform.fit_transform(x_test)
mode= LogisticRegression()#用默认的就行
mode.fit(x_train,y_train)#得到了模型
y_predict = mode.predict(x_test)
#print("y_predict:\n", y_predict)
print("直接比对真实值和预测值:\n", y_test == y_predict)
# 计算准确率
accuracy = mode.score(x_test, y_test)
print("准确率为:\n", accuracy)
# 打印分类报告
res = classification_report(y_test, y_predict, labels=[2, 4], target_names=['良性', '恶性'])
print(res)