python机器学习 | 聚类算法之K-Means算法介绍及实现
本篇博客具体学习参考:
K-means聚类算法原理及python实现
聚类算法之K-Means算法介绍及实现
- 1 K-Means算法介绍
-
- 1.1 聚类算法介绍
- 1.2 K-means算法思想和流程介绍
- 1.3 K-means算法的代码实现
-
- 1.3.1 简单例子
- 1.3.2 python代码实现
- 1.4 K-means评价
- 1.5 K-means优化
- 2 K-means的API
-
- 2.1 API介绍
- 2.2 API应用
-
- 2.2.1 国家的足球运动等级聚类
- 2.2.2 菊花图像的颜色分割
- 2.2.3微信界面的颜色分割
1 K-Means算法介绍
1.1 聚类算法介绍
- 对于"监督学习"(supervised learning),其训练样本是带有标记信息的,并且监督学习的目的是:对带有标记的数据集进行模型学习,从而便于对新的样本进行分类。
- 而在“无监督学习”(unsupervised learning)中,训练样本的标记信息是未知的,目标是通过对无标记训练样本的学习来揭示数据的内在性质及规律,为进一步的数据分析提供基础。对于无监督学习,应用最广的便是"聚类"(clustering)。
- 聚类算法试图将数据集中的样本划分为若干个通常是不相交的子集,每个子集称为一个“簇”(cluster),通过这样的划分,每个簇可能对应于一些潜在的概念或类别。
- 简单来说:聚类算法的样本集都是没有标签的,那我们就需要根据样本的特征,给样本数据集进行聚类。并且无标签的算法也叫做无监督学习。
1.2 K-means算法思想和流程介绍
kmeans算法,又称为k均值算法。K-means算法中的k表示的是聚类为k个簇,means代表取每一个聚类中数据值的均值作为该簇的中心,或者称为质心,即用每一个的类的质心对该簇进行描述。
-
即,K-Means算法接受参数K;然后将样本数据集划分为K个聚类。获得的聚类需要满足:同一个聚类中的样本数据集相似度较高;而不同聚类中的样本数据集相似度较小。
-
算法思想为:以空间中K个点为中心进行聚类(即先从样本集中随机选取 k个样本作为簇中心),对最靠近他们的对象归类(所有样本与这 k个“簇中心”的距离,对于每一个样本,将其划分到与其距离最近的“簇中心”所在的簇中)。通过迭代的方法,逐次更新各聚类中心的值,直至得到最好的聚类结果。
-
算法流程:
- .先从没有标签的元素集合A中随机取K个元素,作为K个子集各自的质心。
- 分别计算剩下的元素到K个子集质心的距离,根据距离将元素分别划分到最近的子集。
- 根据聚类结果,重新计算质心(计算方法为子集中所有元素各个维度的算术平均数)
- 将集合A中全部元素按照新的质心然后再重新聚类。
- 重复第4步,直到聚类结果不再发生变化。
1.3 K-means算法的代码实现
1.3.1 简单例子
先简单举个例子,来串一下流程,再封装函数
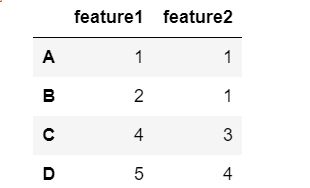
例子:我们现在我们有A、B、C、D四个数据。其中,每个数据都有两个特征。现在我们需要给它们进行聚类。

聚类实现代码为:
导入库+读取数据
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
df = pd.read_excel("test.xlsx",index_col=0)
- .先从没有标签的元素集合A中随机取K个元素,作为K个子集各自的质心。
# 初始化质心
c1 = (1,1)
c2 = (2,1)
- 分别计算剩下的元素到K个子集质心的距离,根据距离将元素分别划分到最近的子集。
# 每个点分别到c1与c2的距离 (广播机制)
# 点到点的距离公式为:np.sqrt((f1-x1)**2+(f2-x2)**2)
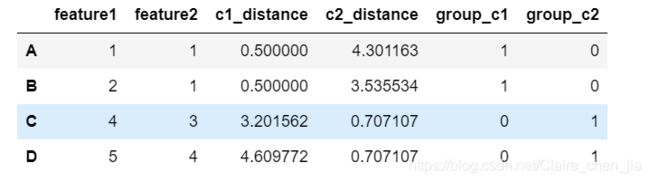
df["c1_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c1)**2,axis=1))
df["c2_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c2)**2,axis=1))
df
"""
需要比较 c1_distance 与 c2_distance 大小
- c1_dis小:1 否则赋值为0
- c2_dis小:1 否则赋值为0
"""
def test(s):
if s[0]<s[1]:
return 1
else:
return 0
def test2(s):
if s[0]>s[1]:
return 1
else:
return 0
df["group_c1"] = df.loc[:,"c1_distance":"c2_distance"].apply(test,axis=1)
df["group_c2"] = df.loc[:,"c1_distance":"c2_distance"].apply(test2,axis=1)
df

3. 根据聚类结果,重新计算质心(计算方法为子集中所有元素各个维度的算术平均数)
4. 将集合A中全部元素按照新的质心然后再重新聚类。
"""
此时并不平衡,需要去调整质心。
- c1-->1
- c2-->3
"""
c2 = df[df.group_c2==1].iloc[:,[0,1]].mean(axis=0).values #根据聚类结果,重新计算质心(计算方法为子集中所有元素各个维度的算术平均数)
c2
# 每个点分别到c1与c2的距离
# 点到点的距离公式为:np.sqrt((f1-x1)**2+(f2-x2)**2)
df["c1_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c1)**2,axis=1))
df["c2_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c2)**2,axis=1))
df
def test(s):
if s[0]<s[1]:
return 1
else:
return 0
def test2(s):
if s[0]>s[1]:
return 1
else:
return 0
df["group_c1"] = df.loc[:,"c1_distance":"c2_distance"].apply(test,axis=1)
df["group_c2"] = df.loc[:,"c1_distance":"c2_distance"].apply(test2,axis=1)
df
plt.scatter(df.feature1.values,df.feature2.values,marker="*",s=200)
plt.scatter(c1[0],c1[1],color="r")
plt.scatter(c2[0],c2[1],color="r")
plt.show()
# 两个点的质心都需要调整
c1 = df[df.group_c1==1].iloc[:,[0,1]].mean(axis=0).values
c2 = df[df.group_c2==1].iloc[:,[0,1]].mean(axis=0).values
# 每个点分别到c1与c2的距离
# 点到点的距离公式为:np.sqrt((f1-x1)**2+(f2-x2)**2)
df["c1_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c1)**2,axis=1))
df["c2_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c2)**2,axis=1))
df
def test(s):
if s[0]<s[1]:
return 1
else:
return 0
def test2(s):
if s[0]>s[1]:
return 1
else:
return 0
df["group_c1"] = df.loc[:,"c1_distance":"c2_distance"].apply(test,axis=1)
df["group_c2"] = df.loc[:,"c1_distance":"c2_distance"].apply(test2,axis=1)
df
# 每个点分别到c1与c2的距离
# 点到点的距离公式为:np.sqrt((f1-x1)**2+(f2-x2)**2)
df["c1_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c1)**2,axis=1))
df["c2_distance"] = np.sqrt(np.sum((df.iloc[:,[0,1]].values-c2)**2,axis=1))
df
def test(s):
if s[0]<s[1]:
return 1
else:
return 0
def test2(s):
if s[0]>s[1]:
return 1
else:
return 0
df["group_c1"] = df.loc[:,"c1_distance":"c2_distance"].apply(test,axis=1)
df["group_c2"] = df.loc[:,"c1_distance":"c2_distance"].apply(test2,axis=1)
plt.scatter(df.feature1.values,df.feature2.values,marker="*",s=200)
plt.scatter(c1[0],c1[1],color="r")
plt.scatter(c2[0],c2[1],color="r")
plt.show() # 质心分布合适

质心不再发生改变,并且质心与点的距离,和质心都不再改变,聚类完成。
1.3.2 python代码实现
import random
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
" 计算质心到点的距离"
def calcDis(dataSet, centroids, k):
clalist=[]
for data in dataSet:
diff = np.tile(data, (k, 1)) - centroids #相减 (np.tile(a,(2,1))就是把a先沿x轴复制1倍,即没有复制,仍然是 [0,1,2]。 再把结果沿y方向复制2倍得到array([[0,1,2],[0,1,2]]))
squaredDiff = diff ** 2 #平方
squaredDist = np.sum(squaredDiff, axis=1) #和 (axis=1表示行)
distance = squaredDist ** 0.5 #开根号
clalist.append(distance)
clalist = np.array(clalist) #返回一个每个点到质点的距离len(dateSet)*k的数组
return clalist
""" 计算质心,求出样本属于哪一个簇 """
def classify(dataSet, centroids, k):
# 计算样本到质心的距离
clalist = calcDis(dataSet, centroids, k)
# 分组并计算新的质心
minDistIndices = np.argmin(clalist, axis=1) #axis=1 表示求出每行的最小值的下标
newCentroids = pd.DataFrame(dataSet).groupby(minDistIndices).mean() #DataFramte(dataSet)对DataSet分组,groupby(min)按照min进行统计分类,mean()对分类结果求均值
newCentroids = newCentroids.values
# 计算变化量
changed = newCentroids - centroids
return changed, newCentroids
""" 点的簇分布 --> 展示出来"""
def showCluster(cluster):
"""
绘制样本点
"""
# 用不同颜色形状来表示各个类别
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', ', 'pr']
for i,points in enumerate(cluster): # 取出簇
# 因为同一个簇的需要绘制同样的颜色,所以需要得到同一个簇的索引
markIndex = int(i)
for point in points : # 取出属于簇中的每个样本点
plt.plot(point[0],point[1],mark[markIndex])
"""
绘制质心点
"""
# 用不同颜色形状来表示各个类别
mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', ', 'pb']
# 绘制质心点
for j in range(len(centroids)):
plt.plot(centroids[j][0],centroids[j][1],mark[j], markersize = 20)
plt.show
"""封装并 使用k-means分类"""
def kmeans(dataSet, k):
# 随机取质心
centroids = random.sample(dataSet, k)
# 更新质心 直到变化量全为0
changed, newCentroids = classify(dataSet, centroids, k)
while np.any(changed != 0):
changed, newCentroids = classify(dataSet, newCentroids, k)
centroids = sorted(newCentroids.tolist()) #tolist()将矩阵转换成列表 sorted()排序
# 根据质心计算每个集群
cluster = []
clalist = calcDis(dataSet, centroids, k) #调用欧拉距离
minDistIndices = np.argmin(clalist, axis=1)
for i in range(k):
cluster.append([])
for i, j in enumerate(minDistIndices): #enumerate()可同时遍历索引和遍历元素 i对应索引,j对应元素(0,...,k-1),而元素对应的刚好是k
cluster[j].append(dataSet[i])
# 展示点的簇分布
showCluster(cluster)
# 计算平均损失,判断不同k的分类效果
minDistance = np.min(clalist, axis=1) # axis=1 表示求出每行的最小值
loss = np.mean(minDistance) # # 计算距离变化值(损失) 样本跟它所属簇的误差求和/样本个数-->平均损失
return centroids, cluster,loss
"""创建数据集 """
def createDataSet():
data = pd.read_csv("data.csv").values
plt.scatter(data[:,0],data[:,1])
plt.show()
data = np.array(data)
data = data.tolist()
return data
if __name__=='__main__':
dataset = createDataSet()
# 存储每个k值下的损失
loss_list = []
for k in range(2,10): # k的范围在2,9
centroids, cluster,loss = kmeans(dataset, k)
# print('质心为:%s' % centroids)
# print('集群为:%s' % cluster)
loss_list.append(loss)
# 观察k值与损失的关系
plt.figure(figsize=(8,6))
plt.plot(range(2,9),loss_list) # 绘制不同k下的效果
plt.xlabel('k')
plt.ylabel("loss")
plt.show()
1.4 K-means评价
优点:容易实现
缺点:1)对K个初始质心的选择比较敏感,容易陷入局部最小值;2)K值是用户指定的,而用户很难去选择一个合适的完美的K值。又因为没有标签,很难进行评估;3)对于一些复杂的数据分布就无法进行正确的聚类,如下图。

1.5 K-means优化
解决对初始质心敏感问题:使用多次的随机初始化,计算每一次建模得到的代价函数的值,选取代价函数最小结果作为聚类结果。
解决k值选择困难问题——肘部法则:肘部法则实际上就是来观察代价函数与k之间的关系。找出拐点位置(肘部位置)的点也就是理想的K值取值。
2 K-means的API
2.1 API介绍
from sklearn.cluster import KMeans
KMeans(
n_clusters=8,
*,
init='k-means++',
n_init=10,
max_iter=300,
tol=0.0001,
precompute_distances='deprecated',
verbose=0,
random_state=None,
copy_x=True,
n_jobs='deprecated',
algorithm='auto',
)
- n_clusters: 即 K 值,一般需要多试一些 K 值来保证更好的聚类效果。你可以随机设置一些 K 值,然后选择聚类效果最好的作为最终的 K 值;
- max_iter: 最大迭代次数,如果聚类很难收敛的话,设置最大迭代次数可以让我们及时得到反馈结果,否则程序运行时间会非常长;
- n_init:初始化中心点的运算次数,默认是 10。程序是否能快速收敛和中心点的选择关系非常大,所以在中心点选择上多花一些时间,来争取整体时间上的快速收敛还是非常值得的。由于每一次中心点都是随机生成的,这样得到的结果就有好有坏,非常不确定,所以要运行 n_init 次, 取其中最好的作为初始的中心点。如果 K 值比较大的时候,你可以适当增大 n_init 这个值;
- init: 即初始值选择的方式,默认是采用优化过的 k-means++ 方式,你也可以自己指定中心点,或者采用 random 完全随机的方式。自己设置中心点一般是对于个性化的数据进行设置,很少采用。random 的方式则是完全随机的方式,一般推荐采用优化过的 k-means++ 方式;
- algorithm:k-means 的实现算法,有“auto” “full”“elkan”三种。一般来说建议直接用默认的"auto"。简单说下这三个取值的区别,如果你选择"full"采用的是传统的 K-Means 算法,“auto”会根据数据的特点自动选择是选择“full”还是“elkan”。我们一般选择默认的取值,即“auto” 。
2.2 API应用
2.2.1 国家的足球运动等级聚类
读取以下数据,根据2019年国际排名、2018世界杯、2015亚洲杯获奖情况,对国家足球进行聚类。

import numpy as np
import pandas as pd
"""
得出信息
- 20行样本数据
- 3个特征
- 有无缺失值:无
"""
df = pd.read_csv("athlates.csv",encoding="gbk")
df.info()
df.head()
# 获取三列特征值
train_x = df[["2019年国际排名","2018世界杯","2015亚洲杯"]]
df_02 = pd.DataFrame(train_x)
导入模块
from sklearn.cluster import KMeans
from sklearn import preprocessing
# 建立模型 设置为3个簇
k_model = KMeans(n_clusters=3)
k_model
# 数据归一化
min_max_scaler = preprocessing.MinMaxScaler()
train_x = min_max_scaler.fit_transform(train_x)
# 训练模型
k_model.fit(train_x)
predict_y = k_model.predict(train_x)
predict_y
df["聚类"] = predict_y
df
2.2.2 菊花图像的颜色分割
from matplotlib.image import imread
from matplotlib import pyplot as plt
from sklearn.cluster import KMeans
image = imread("flowers.png")
# 三维:(行,列,颜色RGB)
image.shape # (533, 800, 3)
# 转为像素点 --> 也就是 行*列 个像素点
# 重塑形状 指定为-1 也就是 行*列*颜色/颜色
X = image.reshape(-1,3)
# 像素点的个数,颜色
X.shape # (426400, 3)
kmeans = KMeans(n_clusters=2).fit(X)
# 获取最终每个簇的质心位置
kmeans.cluster_centers_
# 以质心来表示同一个簇的值
print(kmeans.labels_)
print(kmeans.cluster_centers_)
# 将labels_传到cluster_centers_取到对应簇的值
seg_img = kmeans.cluster_centers_[kmeans.labels_] # 像素点对应的簇的值
print(seg_img.shape) # (426400, 3)
seg_img
# 重塑图片形状 将像素点重置为图片形状
seg_img = seg_img.reshape(533,800,3)
seg_img
plt.figure(figsize=(14,8))
plt.imshow(seg_img)
2.2.3微信界面的颜色分割
f = open("./weixin.jpg",'rb')
"""
获取每个像素的颜色特征,存储二维数组
- 获取图片每个像素点
- 获取图片大小(行,列)
- 嵌套循环来获取每个像素点
- 获取每个像素点的颜色特征
- 存储为二维数组
"""
import PIL.Image as image
data = []
img = image.open(f)
width,height = img.size
# 获取每一个像素点
for x in range(width):
for y in range(height):
# 获取每个像素点的RGB颜色特征
c1,c2,c3 = img.getpixel((x,y))
data.append([c1,c2,c3])
f.close()
data
"""
数据的归一化
"""
from sklearn.preprocessing import MinMaxScaler
# 构建归一化模型
min_max = MinMaxScaler()
# 训练
data = min_max.fit_transform(data)
data
"""
聚类
"""
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=2) # 2类
kmeans
kmeans.fit(data)
label = kmeans.predict(data)
label
"""
聚类的标识就为0或者1
除此之外,得到的聚类的结果是一维的向量
需要将它转化为图像尺寸的矩阵
"""
label = label.reshape([width,height])
label
"""
想要将聚类后分割的结果呈现出来,我们可以构建新的图片对象
"""
# 构建新的对象
pic_mark = image.new("L",(width,height))
pic_mark
"""
怎么将聚类好的颜色呈现上去
所以,我们需要将0,1转为颜色特征(灰度值)0-255
实现:
- 取出每个点,添加颜色值
- 标签类别:1-->设置灰度值为255
- 标签类别:0-->设置灰度值为127
"""
for x in range(width):
for y in range(height):
# 每个聚类的标签:为0,1
# print(label[x,y])
# print(int(256/(label[x,y]+1))-1)
# 给每个像素点设置颜色值
if label[x,y] == 1:
pic_mark.putpixel((x,y),255)
else:
pic_mark.putpixel((x,y),127)
# 保存新的图片
pic_mark.save("wechat_mark.jpg","JPEG")