详细解答T-SNE程序中from sklearn.manifold import TSNE的数据设置,包括输入数据,绘制颜色的参数设置,代码复制可用!!
文章目录
- 前言——TSNE是t-Distributed Stochastic Neighbor Embedding的缩写
- 1、可运行的T-SNE程序
- 2. 实验结果
- 3、针对上述程序我们详细分析T-SNE的使用方法
-
- 3.1 加载数据
- 3.2 TSNE降维
- 3.3 绘制点
- 3.4 关于颜色设置,颜色使用的标签数据的说明c=y
- 总结
前言——TSNE是t-Distributed Stochastic Neighbor Embedding的缩写
TSNE是t-Distributed Stochastic Neighbor Embedding的缩写,它是一个非线性降维算法。
TSNE的主要作用和优点如下:
-
将
高维数据投影到低维空间,如二维或三维,实现高维数据的可视化。 -
相比其他降维方法如PCA,TSNE
在保留局部结构信息上的效果更好,尤其适用于高维稠密数据。 -
它可以很好地区分数据中的簇结构,
有利于观察不同类别或类型的数据分布情况。
1、可运行的T-SNE程序
from sklearn.datasets import load_iris
from sklearn.manifold import TSNE
import matplotlib.pyplot as plt
import matplotlib
matplotlib.use('TkAgg')
# 加载数据
iris = load_iris()
X = iris.data
y = iris.target
# TSNE降维
tsne = TSNE(n_components=2, random_state=0)
X_tsne = tsne.fit_transform(X)
# 绘制点
plt.scatter(X_tsne[:,0], X_tsne[:,1], c=y, marker='o', s=5)
# 添加图例
plt.legend(iris.target_names)
# 添加标题
plt.title("TSNE projection of the Iris dataset")
plt.show()
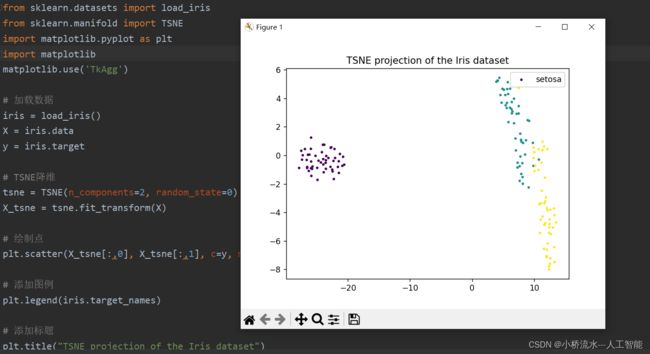
2. 实验结果
3、针对上述程序我们详细分析T-SNE的使用方法
3.1 加载数据
- load_iris()函数从sklearn.datasets模块加载鸢尾花数据集,
- iris包含数据集的特征
数据X和标签数据y。
3.2 TSNE降维
-
TSNE是一种非线性降维算法,用于高维数据的可视化。它可以将高维数据投影到二维或三维空间。
-
TSNE(n_components=2)实例化一个TSNE模型,
降维后的维度数设为2。 -
random_state=0
固定随机数种子,使得结果可重复。 -
fit_transform(X)对
特征数据X进行降维,返回降维后的新特征X_tsne。
3.3 绘制点
-
X_tsne包含每个样本的
二维坐标。 -
plt.scatter以(x,y)坐标方式绘制每个点,
c=y指定点的颜色。 -
marker='o’设置点的形
状为圆形。 -
s=5控制点的大小。
通过TSNE降维,高维数据X被投影到二维空间,得到低维表示X_tsne。然后根据X_tsne和y进行散点图绘制,就可以实现TSNE降维结果的可视化。这是TSNE的标准流程。
3.4 关于颜色设置,颜色使用的标签数据的说明c=y
c=y这行代码的含义和作用是:
-
c参数用于设置散点图中每个点的颜色。 -
y变量包含了样本的类别标签信息。对于鸢尾花数据集来说,y取值为0、1或2,分别表示三种花的类别。 -
当我们设置c=y时,就是根据每个样本在
y中的类别标签值,来动态设置这个样本点在散点图中的颜色。 -
具体来说:
-
如果一个样本的
y值为0,那么这个点的颜色就会取颜色映射中的第一个颜色。 -
如果
y值为1,点颜色取第二个颜色。 -
如果
y值为2,点颜色取第三个颜色。
-
-
这样每个类别的样本点就会使用不同的颜色来绘制,从而在可视化结果中清晰区分开各个类别。
总结
-
在科研中,
TSNE广泛应用于图像分类、自然语言处理等领域的数据降维和可视化。 -
比如对神经网络分类结果进行TSNE降维,可以
观察不同类别样本在特征空间中的分布,有助于分析模型表现。 -
对文本语料进行TSNE降维,可以观察词汇在语义空间中的分布
,帮助理解语义结构。 -
对单细胞RNA-seq数据进行TSNE降维,可以
观察不同类型细胞在表达空间中的分布,有助于发现新型细胞亚群。
所以总体来说,TSNE通过高效的降维和保留局部结构,有助于科研人员直观观察高维数据的内在结构,分析模型效果,发现数据中的新知识,从而推动科研工作的进展。它为数据可视化和理解提供了重要的工具支持。