佛罗里达大学利用神经网络,解密 GPCR-G 蛋白偶联选择性
内容一览:G 蛋白偶联受体 (GPCRs) 是一种将细胞膜外的刺激,传递到细胞膜内的跨膜蛋白,广泛参与到人体生理活动当中。近日,佛罗里达大学的研究者测定了 GPCRs 和 G 蛋白的结合选择性,并开发了预测二者选择性的算法,对这一选择性的结构基础进行了研究。
关键词:GPCR 神经网络 药物研发
作者 | 雪菜
编辑 | 三羊
G 蛋白偶联受体 (GPCRs) 是一种将细胞膜外的刺激传递到细胞膜内的跨膜蛋白。通过激活细胞膜内的 G 蛋白及其下游信号通路,GPCRs 能够广泛参与到发育、免疫、激素调节和神经活动等重要生理活动中。
G 蛋白由 Gα、Gβ 和 Gγ 亚基组成,其多样性决定了 GPCRs 信号反应的多样性。人类基因组共编码了 16 个 Gα 亚基,分为 4 个亚家族 Gαi/o、Gαq、Gαs 和 Gα12/13。G 蛋白决定了下游的信号通路,进而决定了细胞反应。因此,GPCRs 和 G 蛋白的选择性结合是理解生物体信号系统的关键。
学界一度认为 GPCRs 只会与单个 G 蛋白偶联,进而从功能上将 GPCRs 对应划分为 4 种。然而近期研究者发现,大多数 GPCRs 会与多个 G 蛋白偶联,以激活复杂细胞反应。一对一的分类模式已经不足以描述 GPCRs 和 G 蛋白的偶联关系,然而 GPCRs-G 蛋白的选择性机制又暂不明晰。
为此,佛罗里达大学的研究者使用动力学测量和生物发光共振能量转移 (BRET) 技术,测定了 GPCRs 对 G 蛋白的鸟苷酸交换因子,以分析二者的选择性结合。据此,研究人员以 GPCRs 对 G 蛋白的偏好对其分类,建立了包括不同哺乳动物的 124 种 GPCRs 的粗粒度模型。随后,开发了一个预测 GPCRs-G 蛋白选择性的算法,研究了选择性的结构基础。相关成果已发表在 「Cell Reports」。
这一成果已发表于「Cell Reports」
论文链接:
https://doi.org/10.1016/j.celrep.2023.113173
01 BRET:GPCRs-G 蛋白选择性量化
为了量化检测 GPCRs-G 蛋白选择性,研究人员使用 BRET 技术在活体细胞中测量了 G 蛋白的活性。
实时检测 G 蛋白活性的 BRET 技术
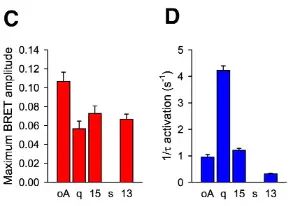
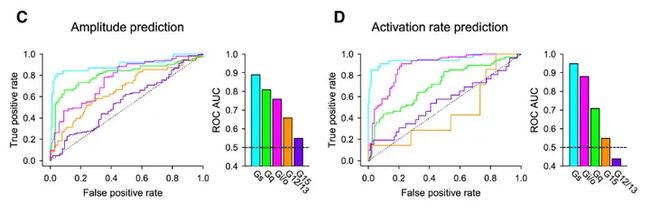
随后,研究人员在胆囊收缩素 Ⅱ 型受体 (CCKBR) 上进行了验证。响应振幅 (Amplitude) 结果表明,CCKBR 可以激活 Gαi/o、Gαq、Gα15 和 Gα12/13 家族的 G 蛋白,且激活水平差距不大,但无法激活 Gαs 家族的蛋白。
而基于激活率 (Activation Rate) 的结果清晰表明,CCKBR 对 Gαq 家族激活效果最好,随后才是 Gαi/o、Gα15 和 Gα12/13,这说明基于激活率的 BRET 技术可以捕获到不同 G 蛋白活性之间的细微差异。
基于振幅的 BRET 结果 (C) 及基于激活率的 BRET 结果 (D)
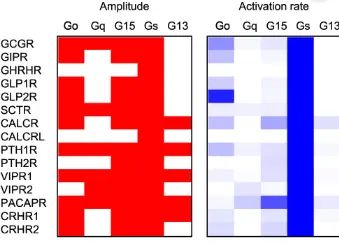
据此,研究人员测量了 124 种 GPCRs 和 G 蛋白的选择性,作为本研究的数据集。
B 类 GPCRs 与 G 蛋白的选择性测量结果
02 模型构建:二元分类神经网络
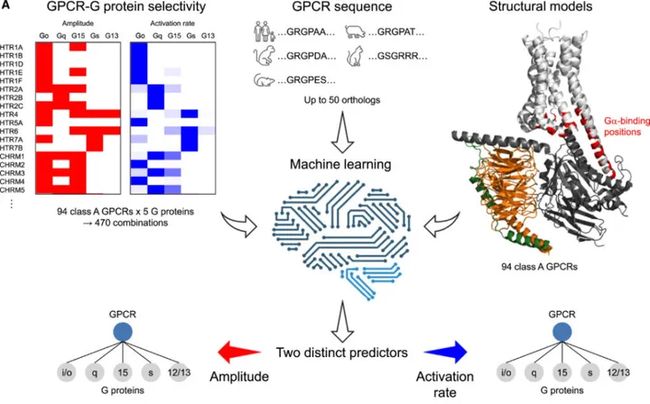
上述结果表明,基于激活率的 BRET 技术能够对上百种 GPCRs 与 G 蛋白的选择性进行区分。基于这一结果,研究人员开发了一种基于机器学习的 A 类 GPCRs-G 蛋白选择性预测算法。
算法的任务包括两个:
1、针对 GPCRs 的偶联性,判断某种 GPCR 能否与 G 蛋白偶联,即振幅 >0%;
2、针对 GPCRs 的选择性,判断某个 GPCR-G 蛋白偶联体能够迅速激活,即激活率 >30%。
机器学习算法的概念图
每个 GPCR 和不同家族的 G 蛋白的偶联都是一个分类问题,因此每个任务可以设计为 5 个二元分类。据此,研究人员设计了 10 个神经网络分类器处理这些任务。神经网络由两个全连接层(分别为 128 个和 16 个神经元)、一个压平层 (Flattening Layer)、三个全连接层(分别为 128 个、32 个和 4 个神经元)以及一个输出层(1 个神经元)组成,内层由校正线性单元 (ReLU) 激活,最后进行批量归一化。输出层通过 sigmoid 函数激活。
由于数据量有限,在假设决定 G 蛋白选择性的序列在进化过程中相对保守的前提下,为每种 GPCR 增加了 50 个同源序列,进行数据扩充。在模型中部署序列嵌入协议,使用无监督深度学习模型描述蛋白质残基在特定环境中的属性。
神经网络的输入是大小为 B*30*1024 的张量。其中第一维度是 batch size (B=32),第二维度是残基数量 (30),第三维度为每个氨基酸残基预训练序列嵌入的大小 (1024)。
模型对振幅和激活率预测的平均 AUROC 均为 0.85,说明模型对两项指标的预测均有较好的性能。其中,对 Gs 家族蛋白预测最佳,AUROC 分别为 0.89 和 0.95。然而,对于 Gα15 和 Gα12/13 家族的蛋白,模型没有展现出明显的学习能力。
振幅 (C) 和激活率 (D) 预测的 ROC 曲线
03 GPCRs-Gα 蛋白选择性的机制解密
BRET 实验和机器学习为破解 GPCRs-G 蛋白选择性的结构基础提供了解决方案。基于此,研究人员调查了可用的 GPCRs-G 蛋白复合物,并对 33 个 A 类受体进行了分析,找到了决定 A 类 GPCRs-Gα 蛋白选择性的结构。
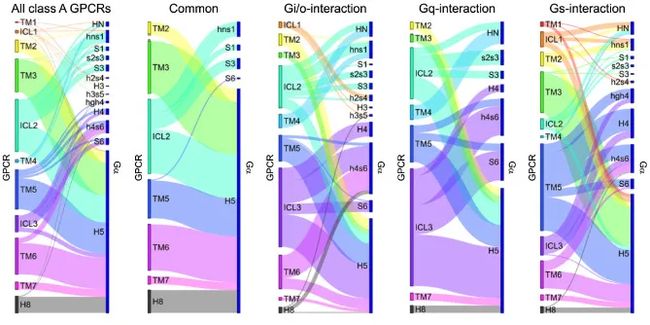
研究人员调查了 GPCRs-Gα 蛋白的残基网络,结果显示 GPCRs 面向细胞质的所有结构均不同程度地参与到了与 Gα 蛋白的结合当中。同样的,Gα 蛋白中也有 13 个结构与 GPCRs-Gα 蛋白的结合相关,其中 C 端的 α-螺旋 (H5) 参与程度最高。
GPCRs 和 Gα 蛋白不同结构元素的交互
对于普遍的 GPCRs-Gα 偶联,GPCRs 使用 ICL2、H8 和大部分 TM 残基与 Gα 蛋白相连。其中,绝大多数结构主要与 H5 相连,而 ICL2 的连接更为广泛。
GPCRs 与 Gαi/o 和 Gαq 家族的蛋白偶联模式类似,唯一的区别在于 GPCRs 与前者的连接严重依赖 TM6,而与后者的连接则不需要。GPCRs 与 Gαs 的连接当中,ICL2 和 ICL3 的份额大幅减少,而更依赖 TM3 和 TM5。上述结果说明,对于不同家族的 Gα 蛋白而言,其与 GPCRs 的连接依赖于不同的结构。
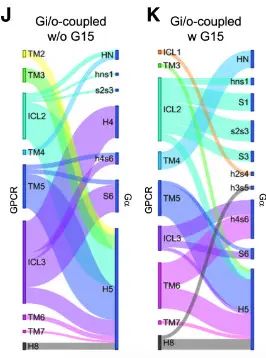
进一步的,结合 GPCRs-G 蛋白的选择性顺序,调查特定结构对不同家族 Gα 蛋白的影响。首先,判断与 Gαi/o 结合的 GPCRs 能否与 Gα15 结合,并对比二者残基网络的差异。与前者相比,GPCRs 与后者的连接切断了 ICL3 和 H4 的联系,弱化了 ICL2-H5 的交互,强化了 TM4-HN 和 ICL2-s2s3 的连接。这说明 ICL2 与其他残基的联系可能是与 Gαi/o 或 Gα15 相连的 GPCRs 的主要区别。
GPCRs 只与 Gαi/o 连接的残基网络 (J) 和 GPCRs 与 Gα15/Gαi/o 相连的残基网络 (K)
同样的,对比了 GPCRs 与 Gαs 和 Gαi/o 结合的残基网络之后,结果显示 ICL1 和 TM5 之间的联系是二者的主要区别。
上述结果说明,BRET 和机器学习能够对 GPCRs-G 蛋白结合的蛋白残基网络进行分析,进而找到二者选择性的结构基础,为 GPCRs 的研究提供了新方法。
04 AI-GPCR:96.4% 的未探索区域
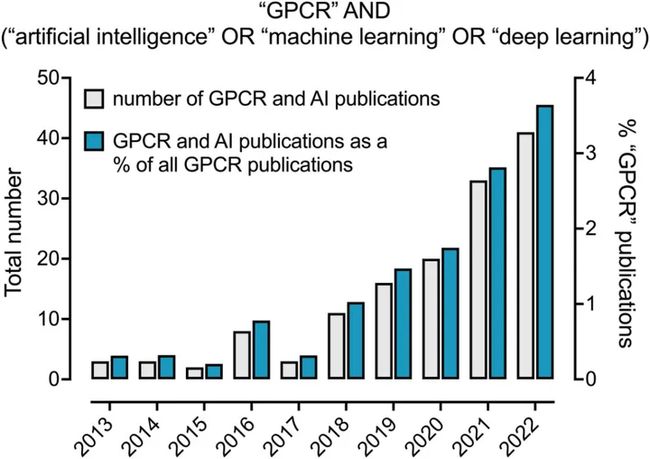
过去十年间,AI 和机器学习在 GPCR 领域的应用比例稳步提升。2022 年,有 3.6% 的 GPCR 相关论文中提到了 AI 相关方法。
GPCR 相关论文中提及 AI 的比例
鉴于 AI 在 GPCR 药物研究中的应用的不断增加,对应的算法也不断被开发。对于分类问题,最常用的算法是传统机器学习领域的常用算法,如 scikit-learn 库中的算法,包括支持向量机 (SVM)、决策树、梯度提升机和 k-近邻算法等。
对于数值结果,如蛋白-配体结合的亲和力等,常用回归算法求解,如多元线性回归、支持向量机和深度学习网络等。
最近的成果多使用多层感知器、卷积神经网络 (CNN) 等深度学习算法进行预测。随着深度学习生成算法的发展,蛋白质配体和结构设计愈发高效和准确。生成对抗性网络 (Generative Adversarial Network)、循环神经网络 (Recurrent Neural Network)、强化学习等算法可以利用向量空间的自动构造和自适应度量来探索更大的生成空间。
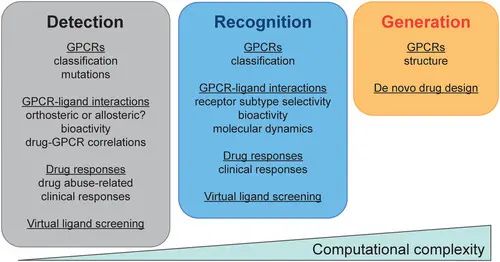
AI 在 GPCR 药物研发各阶段的作用
因此,这些算法可以产生更多具有所需功能的配体,或是更准确地预测未知蛋白质的结构,如 AlphaFold2。虽然 AlphaFold2 等模型并不专用于预测 GPCR 的结构,但仍可以高效准确地预测 GPCR 的结构。此外,无监督或自监督深度学习也在药物发现重崭露头角。
可见,AI-GPCR 可能是未来药物研发的新方向,但同时也为我们留下了 96.4% 的未知区域。在高效分类和精准预测的算法帮助下,人们能够对 GPCR 的偶联机制有更清晰的了解,为生物医学的发展注入新动能。