毕业论文知识点记录(六)——基于R语言优化maxent模型
毕业论文知识点记录(六)——基于R语言优化maxent模型
第一步:R安装
这个网上都有很多详细的步骤,就不再详细介绍了。
第二步:R安装包
因为优化maxent模型需要用到kuenm程序包,但是官网上没有这个程序包,是因为不是所有的R包都提交上传到CRAN,如Github。所以需要从Github安装程序包(GitHub上的kuenm程序包)。总结了一下网上一共有两种方式:第一种,在线安装;第二种离线安装。我参考下面这位博主的方法进行离线安装。
从Github安装R安装包方法
其中安装RTools的时候也遇到了一点点问题,主要是配置环境比较麻烦,但是根据这个步骤去做会成功安装的。Rtools环境配置
OK啊,果然安装过程没有那么简单。
总结起来大概有两种错误:
①“package ‘XX’ is not available for this version of R”
解决办法:将repos值设置为官网地址:https://mran.microsoft.com/snapshot/2019-02-01/
install.packages("hier.part",repos=‘https://mran.microsoft.com/snapshot/2019-02-01/’)
②: lazy loading failed for package ‘kuenm’ * removing ‘D:/R/R-4.2.1/library/kuenm’
这种一般是有个包没有安装,或者包的版本不对。比如我的原因是“Rcpp”这个包现有的版本是1.0.9,但要求是1.0.10,所以手动更新一下就行。
第三步:就是认真学习kuenm程序包
刚才GitHub上的程序包网址链接上有详细的内容介绍,接下来我把我学习的内容总结出来。
一、准备
1、需要准备的数据目录
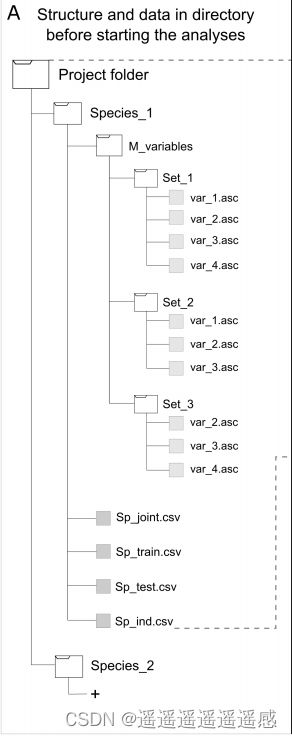
1、包含要使用的不同环境变量集
即图中的M_variables
2、包含训练和测试发生数据的 csv 文件
该数据集由三个字段组成:物种名称、经度和纬度。请参见图中的Sp_joint.csv
3、包含训练模型的发生数据的 csv 文件
需要训练和测试数据的一定程度的独立性。请参阅图中的Sp_train.csv。
4、包含用于测试模型的物种出现数据的 csv 文件
作为校准过程的一部分(即图中的Sp_test.csv)。
5、包含完全独立的发生数据子集的 csv 文件(训练和测试数据外部)
用于最终的正式模型评估。该数据集(即用于最终模型评估)如图中的Sp_ind.csv
我的理解是:你准备的所有发生记录=2+5=3+4+5
2、安装软件包
这个刚才已经进行过了。
3、设置工作目录
setwd("YOUR/DIRECTORY/ku.enm_example_data/A_americanum") # set the working directory
dir() # check what is in your working directory
这一步很重要,一开始我直接从下面的工作流记录开始,结果一直提醒我工作目录里没有我需要的文件,纳闷了半天发现上面还有一段代码!
然后原网址的前半段代码我没有使用,因为我直接下载了数据,所以不需要下载-解压-删掉压缩文件这个步骤。当然,当你跑自己的数据的时候也是不需要的,设置好工作目录就行了。
二、对单个物种项目进行分析
1、工作流记录
数据目录准备好之后,利用函数kuenm_start生成 R Markdown (.Rmd) 文件,作为执行此软件包包含的所有分析的指南。通过记录过程中使用的所有代码块,此文件还有助于使分析更具可重复性。此文件将写入工作目录中。此功能的使用是可选的,但如果需要记录每个物种的单独工作流程,建议使用此功能。
# Preparing variables to be used in arguments
file_name <- "sf_enm_process"
kuenm_start(file.name = file_name)
2、模型校准
(1)创建候选模型
函数kuenm_cal创建并执行一个批处理文件,用于生成Maxent候选模型,该文件将写入输出目录内的子目录,命名为所选参数化。将使用正则化乘数、要素类和环境预测变量集的多种组合创建校准模型。对于每个组合,此函数创建一个包含完整发生次数集的 Maxent 模型,另一个仅包含训练发生次数的 Maxent 模型。
#创建候选模型
# Variables with information to be used as arguments. Change "YOUR/DIRECTORY" by your actual directory.
occ_joint <- "sf_joint.csv"
occ_tra <- "sf_train.csv"
M_var_dir <- "M_variables"
batch_cal <- "Candidate_models"
out_dir <- "Candidate_Models"
reg_mult <- c(seq(0.1, 1, 0.1), seq(2, 6, 1), 8, 10)#这行代码就是调整正则化参数的
f_clas <- "all"#这行代码就是调整函数组合的
args <- NULL # e.g., "maximumbackground=20000" for increasing the number of pixels in the bacground or
# note that some arguments are fixed in the function and should not be changed
maxent_path <- "YOUR/DIRECTORY/ku.enm_example_data/A_americanum"
wait <- FALSE
run <- TRUE
kuenm_cal(occ.joint = occ_joint, occ.tra = occ_tra, M.var.dir = M_var_dir, batch = batch_cal,
out.dir = out_dir, reg.mult = reg_mult, f.clas = f_clas, args = args,
maxent.path = maxent_path, wait = wait, run = run)
代码块中进行标注的两行就是使用者最需要关注的地方,每个人可能会有不同的组合。在这里我的正则化参数是(0.1,5,0.5),也就是从0.1到5,步长变化为0.5。
(2)评估和选择最佳模型
函数kuenm_ceval根据统计显著性(部分ROC)、遗漏率(E =用户选择的可能出现有意义误差的出现数据比例)和模型复杂性(AICc)来评估模型性能,并根据不同的用户设置标准选择最佳模型)。部分ROC和遗漏率是根据使用训练事件创建的模型进行评估的,而AICc值是根据使用完整事件集创建的模型计算的。输出存储在一个文件夹中,该文件夹将包含一个.csv文件,其中包含满足每个评估标准的模型的统计信息,另一个文件仅包含根据用户指定的标准选择的模型,第三个包含所有候选模型的性能指标,模型性能图,以及一个 HTML 文件,报告模型评估和选择过程的所有结果,旨在指导进一步的解释。
#评估和选择最佳模型
occ_test <- "sf_test.csv"
out_eval <- "Calibration_results"
threshold <- 5#遗漏率
rand_percent <- 30#测试集占比
iterations <- 100
kept <- TRUE
selection <- "OR_AICc"
paral_proc <- FALSE # make this true to perform pROC calculations in parallel, recommended
# only if a powerfull computer is used (see function's help)
# Note, some of the variables used here as arguments were already created for previous function
cal_eval <- kuenm_ceval(path = out_dir, occ.joint = occ_joint, occ.tra = occ_tra, occ.test = occ_test, batch = batch_cal,
out.eval = out_eval, threshold = threshold, rand.percent = rand_percent, iterations = iterations,
kept = kept, selection = selection, parallel.proc = paral_proc)
(3)最终模型创建
选择生成最佳模型的参数化后,下一步是创建最终模型,并在需要时将其转移到其他区域或场景。kuenm_mod函数从模型选择过程中获取具有最佳模型的.csv文件,并编写并执行批处理文件,以创建具有所选参数化的最终模型。模型和投影存储在输出文件夹内的子目录中;这些子目录将按候选模型命名。通过允许投影(即,project = TRUE)并定义保存传输数据的文件夹(即G.var.dir参数中的文件夹名称),此函数会自动执行这些传输。但是,创建最终模型的过程可能需要相当长的时间,尤其是在转移到其他区域或方案时。
#最终模型创建
batch_fin <- "Final_models"
mod_dir <- "Final_Models"
rep_n <- 10#模型重复计算的次数(最后取平均值)
rep_type <- "Bootstrap"
jackknife <- FALSE
out_format <- "logistic"
project <- TRUE
G_var_dir <- "G_variables"
ext_type <- "all"
write_mess <- FALSE
write_clamp <- FALSE
wait1 <- FALSE
run1 <- TRUE
args <- NULL # e.g., "maximumbackground=20000" for increasing the number of pixels in the bacground or
# "outputgrids=false" which avoids writing grids of replicated models and only writes the
# summary of them (e.g., average, median, etc.) when rep.n > 1
# note that some arguments are fixed in the function and should not be changed
# Again, some of the variables used here as arguments were already created for previous functions
kuenm_mod(occ.joint = occ_joint, M.var.dir = M_var_dir, out.eval = out_eval, batch = batch_fin, rep.n = rep_n,
rep.type = rep_type, jackknife = jackknife, out.dir = mod_dir, out.format = out_format, project = project,
G.var.dir = G_var_dir, ext.type = ext_type, write.mess = write_mess, write.clamp = write_clamp,
maxent.path = maxent_path, args = args, wait = wait1, run = run1)
(4)最终模型评估
最终模型应使用独立的出现数据(即校准过程中未使用的数据,通常来自不同来源)进行评估。kuenm_feval函数根据统计显著性(部分ROC)和遗漏率(E)评估最终模型。此函数将返回一个文件夹,其中包含一个包含评估结果的.csv文件
#最终模型评估
occ_ind <- "sf_ind.csv"
replicates <- TRUE
out_feval <- "Final_Models_evaluation"
# Most of the variables used here as arguments were already created for previous functions
fin_eval <- kuenm_feval(path = mod_dir, occ.joint = occ_joint, occ.ind = occ_ind, replicates = replicates,
out.eval = out_feval, threshold = threshold, rand.percent = rand_percent,
iterations = iterations, parallel.proc = paral_proc)
由于还有一个植被指数的环境数据没有准备好,所以暂时还没有跑结果。等下一篇文章中将如何在GEE下载月均植被指数之后,在下下篇里面放一下运行的结果。
最后在处理数据的时候,还学习到了其他文献里处理数据的方法
补充两点:
①关于环境数据的去相关:发现很多文献里去相关的步骤是:先把所有变量导入,然后得出贡献率,之后再在SPSS中计算相关系数,如果都大于0.8,保留贡献率大的一个变量。此外还需要去除贡献率小于5%的变量。根据这个思路,重新进行变量筛选。
②草贪发生数据的筛选:还需要筛掉那些坐落在海洋上的点。
这个后来检查了一下,我下载的数据没有落在海洋上的。
