NLP论文阅读记录-EMNLP 2023 | 02 Improving Biomedical Abstractive Summarisation with Knowledge Aggregation
文章目录
- 前言
- 0、论文摘要
- 一、Introduction
-
- 1.1目标问题
- 1.2相关的工作
- 1.3本文贡献
- 三.本文方法
-
- 3.1 任务定义
- 3.2 来自引文的知识聚合
- 3.3摘要生成
- 3.4训练和推理
- 四 实验效果
-
- 4.1数据集
-
- 数据集的创建
-
- 创建过程
- 提取引文图
- 4.2 对比模型
- 4.3实施细节
- 4.4评估指标
- 4.5 实验结果
-
- 自动评估
- 人工评价
- 深入分析
- 五 总结
-
- 局限性
前言
通过引文论文的知识聚合改进生物医学抽象总结 (2310)
code
paper
0、论文摘要
来自生物医学文献的摘要具有明显的特定领域特征,包括专门的写作风格和生物医学术语,这需要对相关文献的深入理解。因此,由于缺乏特定领域的背景知识,现有的语言模型很难生成与生物医学专家生成的技术摘要相媲美的技术摘要。
本文旨在通过聚合源文章中引用的外部论文的知识来提高生物医学抽象概括中语言模型的性能。
我们提出了一种新颖的基于注意力的引文聚合模型,该模型集成了引文论文中的特定领域知识,允许神经网络通过利用论文内容和引文论文中的相关知识来生成摘要。
此外,我们构建并发布了一个大规模的生物医学总结数据集,作为我们研究的基础。
大量的实验表明,我们的模型优于最先进的方法,并在抽象生物医学文本摘要方面取得了实质性改进。
一、Introduction
生物医学文本摘要在促进理解庞大且不断扩展的生物医学文献方面发挥着关键作用(Xie et al., 2022),这对努力保持各自领域知识的临床医生和领域专家提出了重大挑战。字段。为了应对这一挑战,从广泛的生物医学文献中生成高质量的摘要在支持生物医学领域的研究和进步方面具有巨大的潜力(DeYoung et al., 2021)。
1.1目标问题
生物医学自然语言生成(NLG)的关键挑战之一在于有效地处理生物医学文本中流行的特定领域术语。因此,人们进行了大量的研究,主要重点是通过更好地整合生物医学领域的特定领域知识来提高语言质量(Sotudeh Gharebagh 等人,2020 年;Tangsali 等人,2022 年;An 等人, 2021;Tang 等人,2023b)
然而,大多数先前的作品主要尝试通过利用论文内容中的附加注释来整合知识。这些注释包括频繁项(Givchi et al., 2022)、命名实体(Schulze and Neves, 2016;Peng et al., 2021)、实体关系(Shang et al., 2011)以及外部知识系统,例如生物医学本体(Chandu et al., 2017)和外部术语搜索工具(Gigioli 等人,2018)。
令人惊讶的是,在以前的生物医学研究中很少探索包含来自引文论文的外部知识。
现有的生物医学文本摘要语料库通常以模型在生成摘要时仅依赖于源文章的方式构建。
然而,如图 1 所示,引文网络中具有共同研究背景、术语和摘要风格的论文之间存在很强的联系,这将成为改进生物医学抽象概括的有用知识来源,但现有数据集中未捕获。

1.2相关的工作
近年来,出现了多种大规模预训练模型(PLM),例如 BART(Lewis et al., 2019); T5(拉斐尔等人,2020); GPT-2(Radford 等人,2019)在各种任务中表现出了显着的性能提升(Loakman 等人,2023;Zhang 等人,2023;Zhao 等人,2023;Tang 等人,2022b)自然语言生成(NLG)领域。这些 PLM 也被广泛应用于生物医学文本摘要。这些模型,例如BioBERT (Lee et al., 2020) 和 BioBART (Yuan et al., 2022) 通过在 Pubmed4 和 MIMIC-III5 等广泛的生物医学文献语料库上进行训练,取得了显着的性能。然而,某些高级知识,例如对医学术语的理解,不能仅凭通过单词概率的隐式建模。为了解决这一限制,生物医学背景知识理解的提高需要整合额外的知识系统,例如概念本体。这些本体明确地模拟了神经网络学习到的特定领域知识的表示。最近的研究建议结合生物医学知识,包括术语(Tang 等人,2023b))和概念(Chandu 等人,2017),以增强这些语言模型的性能,并弥合语言理解和专业生物医学知识之间的差距。事实上,一些著名的著作专注于通过开放领域的引用来增强摘要,例如 An 等人。 (2021)和安永等人。 (2019)。然而,需要强调的是,语言模型在生物医学领域的进展受到数据集和资源有限可用性的阻碍。这种稀缺性阻碍了专为生物医学应用量身定制的预训练语言模型(PLM)的进一步发展和改进。在本研究中,我们需要一个包含可检索的引文论文的数据集,这使得 Pubmed 和 MIMIC-III 等传统原始数据语料库不够用。迄今为止,我们能找到的唯一公共数据集是文本分析会议 (TAC) 2014 年生物医学总结轨道(Cohan 等人,2014 年)。然而,该数据集的大小有限,仅包含 313 个实例,并且有些过时。因此,我们构建了一个新的数据集来研究生物医学引文增强摘要。
1.3本文贡献
为了解决现有生物医学摘要数据集中的这一差距,我们利用艾伦研究所提供的开源生物医学文献语料库构建了一个新颖的生物医学摘要数据集。
在数据集构建过程中,我们应用严格的过滤标准来消除低质量样本。具体来说,我们丢弃了引用数量不足(少于三个不同引用)的样本,以及在语料库中无法访问其唯一标识符(UID)或引文 UID 的不合格论文。
此外,我们设计了启发式规则来选择非结构化原始数据集并将其转换为 JsonL 格式的结构化数据集。最终数据集包含 10,000 多个实例,每个实例平均有 16 次引用。
据我们所知,这是最大的生物医学文献数据集2,专为引文论文增强生物医学文本摘要而定制。此外,我们还提供了相应的引文网络收集方法,包括被引论文及其关联。
在我们的生物医学摘要数据集的推动下,我们进一步提出了一种新的生物医学文档摘要方法,通过引用论文摘要的形式,我们利用外部特定领域知识来增强神经模型。因此,我们引入了一种基于注意力的网络(Vaswani et al., 2017),该网络动态将从引文摘要中提取的特征与主论文的编码内容特征进行聚合。这种聚合是通过将注意力机制应用于所有被引用论文的相关摘要来实现的,这为后续的摘要解码过程提供了附加功能
来自引文论文摘要的特征。在此框架内,基础语言模型可以有效地利用主论文的特征以及从引用论文中获得的附加特定领域知识。因此,这种集成可以提高文本摘要的性能。大量的实验表明,我们的模型在抽象生物医学文本摘要方面优于最先进的基线。我们还进行了深入的定量分析,以验证基于注意力的引文知识增强框架所获得的性能增益3。
总之,我们的贡献如下:
• 我们构建了一个大规模的生物医学文献数据集,可用于通过从引用论文中提取的外部知识来增强生物医学文本摘要。
• 我们提出了一种新颖的框架,可以有效地利用引文论文来增强大规模语言模型在生物医学文献抽象概括方面的性能。
• 我们进行了广泛的实验来评估我们提出的框架的有效性,包括与SOTA 模型的比较以及对通过聚合不同数量的引用所实现的性能增益的深入分析。
三.本文方法
如图 3 所示,我们提出的框架旨在通过利用一组引文论文中的集体知识来增强基本语言模型的性能。在我们的实验中,我们选择 BART (Lewis et al., 2019) 作为基础模型,这是一种广泛使用的摘要模型,在生物医学领域已经证明了有希望的结果 (Goldsack et al., 2022, 2023)。
在本研究中,我们采用一种策略,将引文论文的摘要与输入文档连接起来,形成模型的输入。这种方法的动机是使模型能够捕获和模拟相关论文中存在的写作风格。通过纳入这些附加信息,我们的目标是提高模型生成高质量摘要的能力,这些摘要与特定领域文献中观察到的惯例和模式保持一致。
3.1 任务定义
该任务的表述如下:给定一篇论文文档 di ∈ D 作为输入,其中 D 表示论文语料库,di 表示第 i 篇论文。此外,还提供引文论文 Dc = {dc1, dc2, …, dc k} 作为输入。 dc k ∈ Dc 的摘要表示为absck。 di 或 djc 由表示为 X = {x1, x2, …, xt} 的单词序列组成,其中 xt 表示 X 中的第 t 个单词。目标是生成摘要 Y = {y1, y2, …。 …, yt} 通过对条件概率分布 P (Y |X Î di, X Î Dc) 进行建模。
3.2 来自引文的知识聚合
输入 在初始阶段,输入文档di 及其检索到的N 个引文摘要absc 都通过语言模型连接和编码。字节对编码(Radford et al., 2019)是在从文本到固定词嵌入的转换中实现的:

其中 LMemb 表示负责将单词标记和转换为子词嵌入的模块。 T okCLS 是一个特殊的标记,表示输入文本中的全局上下文标记。 T okABS 是一个特殊的标记,用于指示输入文档和引用的摘要之间的区别。 EQj 表示为第 j 个 (j ∈ [1, N ]) 文档摘要对生成的嵌入。
Encoding
为了捕获每个引用摘要的相关性,我们采用注意机制来衡量 di 相对于 absjc 的重要性。注意力分数记为attnij,聚合知识的过程如下图所示:

其中 EQ 表示所有组合 Qj 的嵌入矩阵,由语言模型编码器编码以生成编码特征 Q。

在上面的等式中,First_Pool收集表示输入di和absjc对的全局上下文的特征。作为神经编码器的隐藏状态,Q 结合了文档和摘要的特征。因此,Q 中第一个位置的表示(表示为 QCLS)对应于全局上下文标记 T okCLS。通过将可训练参数 W Q ∈ RM×1 应用于 QCLS 的特征来获得注意力 logits 矩阵。应用soft tmax函数进行归一化后,Attn代表输入特征的重要性,并用于重新加权原始编码特征Q,得到最终特征F。
3.3摘要生成
与其他抽象摘要系统一致,我们采用自回归解码器以顺序方式生成摘要标记 yt 。流程描述如下:
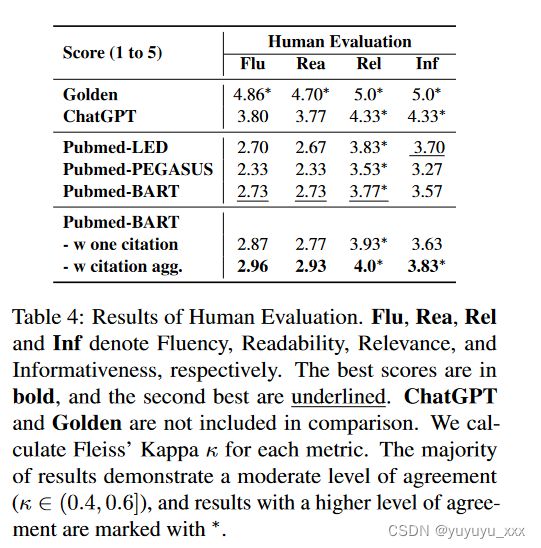
最后,如图 3 所示,通过以下目标函数训练神经模型以适应引文增强训练集: 为了创建包含生物医学文献及其相关引文的数据集,我们处理艾伦研究所发布的半结构化原始语料库 6。我们在整篇论文中将该数据集称为 BioCiteDB。数据集的构建过程如算法1所示,其中C代表原始语料库,D代表处理后的数据集。 科学论文通过引用关系错综复杂地连接起来,形成互连节点的网络。该引文图提供了有关论文相关性的宝贵见解。为了检索本引文中的相关论文和图,我们提出了算法2中概述的算法。hopmax定义了算法可以遍历的论文之间的最大跳数,N指定了每个跳检索到的论文的最大数量。作为输出,P 将论文表示为节点,而引用关系表示为网络中的边。由于处理长文档进行摘要的计算成本很高,考虑到可用计算资源的限制,我们将 hopmax 设置为 1,neighbormax 设置为 12。然而,值得注意的是,基于注意力的引文聚合模块可以扩展以合并图注意力网络(Zhou et al., 2020),该网络具有集成多层引文图的能力(Zhang et al., 2023)。 Baselines 使用的所有预训练模型都是从 Hugging Face9 上公开的检查点恢复的。我们选择的检查点包括:LED10、PEGASUS11、BART12、Pubmed-LED13、Pubmed-PEGASUS14 和 Pubmed-BART15。为了使比较公平,所有输入文本根据所选语言模型的最小输入大小限制进行分块。在我们的实验中,它是 BART(1024 个代币)。模型在具有 40 GB GPU 内存的 Tesla A40 机器上训练最多 10 个 epoch,并根据验证过程中生成的响应的复杂性保留最佳检查点 在文本摘要领域(Sun et al., 2021; Tang et al., 2022a; Xie et al., 2022),ROUGE(Lin, 2004)是评估生成摘要最常用的指标。为了评估生成的摘要的质量,我们使用 rouge_score 的 python 包实现了 ROUGE 指标。具体来说,我们报告生成的摘要和参考(黄金)摘要之间的一元和二元重叠(分别为 ROUGE-1 和 ROUGE-2)。此外,我们还包括最长公共子序列(ROUGE-L)指标来评估生成的摘要的流畅性。对于每个 ROUGE 指标,我们提供精确度、召回率和 F 值的细粒度测量,从而对摘要性能进行全面评估。除了 ROUGE 指标之外,我们还利用更广泛的评估指标进行广泛的自动评估。具体来说,我们使用 BERTScore (Zhang et al., 2019) (BeS) 和 BartScore (Yuan et al., 2021) (BaS) 来评估生成输出的质量。我们还引入了一些可读性指标,例如Flesch-Kincaid (FLK) 和 Coleman-Liau Index (CLI),用于评估生成文本的可读性。这种综合评估可以对多个维度的总结表现进行更稳健的评估。 所有实验的结果如表 2 所示。可以看出,我们提出的框架(-w 引用聚合)在所有 ROUGE 分数(F1 分数)上显着优于所有基线模型,表明生物医学论文的总结能力显着增强。更具体地说,引文知识的结合极大地提高了召回率,ROUGE-1 提高了 5.7%,ROUGE-2 提高了 8.1%。这表明引用知识的整合促进了从参考文献摘要中提取的更多相似表达的利用。 为了对生成的摘要进行更全面的评估,我们还结合了人的评价。本次评估重点关注四个关键方面:流畅性、可读性、相关性和信息性。流利度评估旨在衡量摘要中使用的语言的整体质量。可读性评估决定摘要容易被读者理解的程度。相关性评估检查摘要的内容是否相关并与输入文档的内容一致。信息性测量评估生成的摘要提供从给定输入得出的充分且有意义的信息的程度。通过结合人工评估,我们可以评估自动化指标可能无法完全捕获的摘要质量的主观方面。考虑到评估生成的摘要的难度,需要对源论文和摘要的内容有透彻的理解,因此人类评估者必须拥有强大的学术写作和生物医学知识背景。我们通过滚雪球抽样邀请 3 名合格的评估员对测试集中随机抽样的 30 个实例进行评分。为了最大限度地减少偏差并增加注释者之间的一致性,为评估者提供了相同的注释指南(参见附录 A.3)。人工评价的结果如表 4 所示。可以看出,-w 一次引用和 -w 引用总数。与其他基线模型相比,模型表现出优越的性能,从而证实了我们提出的框架的有效性。 为了进一步深入评估,相关性和信息性指标强调了从输入内容中提取相关信息并生成综合摘要的能力的提高。此外,流畅性和可读性指标评估了语言质量,表明语言模型生成的摘要更加连贯和自然。然而,值得注意的是,与 ChatGPT 的性能相比,测试的预训练语言模型 (PLM) 在语言质量方面表现出显着的差异。这种差异可归因于模型大小的巨大差异,ChatGPT 具有 1300 亿个参数,而测试的 PLM 具有不到 50 亿个参数。 为了进一步研究引文知识聚合模块的影响,我们进行了评估以评估生成的摘要的改进。该评估涉及使用 ROUGE 分数将我们提出的框架(表示为 -w 引用聚合)的性能与基本模型 Pubmed-BART 进行比较。结果如图 4 (a)、(b) 和 © 所示,说明了不同引用次数下 ROUGE 分数(F 值)的增加。事实证明,引用的包含对摘要生成过程有积极的影响。高斯核平滑的增长曲线如图 4(d)所示,表明了一个明显的趋势:随着更多引文摘要的引入,语言模型表现出更大的改进。结果凸显了利用引文信息提高生成摘要质量的潜力。 总之,我们提出了一种新颖的基于注意力的引文聚合模型,该模型结合了引文论文中的特定领域知识。通过整合这些附加信息,我们的模型使神经网络能够生成受益于论文内容和从引文论文中提取的相关知识的摘要。此外,我们引入了专门的生物医学总结数据集,它作为评估和推进我们的研究的宝贵资源。我们的方法的有效性通过大量的实验得到了证明,我们的模型在生物医学文本摘要方面始终优于最先进的方法。结果凸显了通过利用引文论文中的知识所取得的显着改进,以及我们的模型通过自然语言生成技术增强对生物医学文献的理解的潜力。 在文本摘要领域,通常采用两种主要方法:提取摘要和抽象摘要。提取式摘要是通过直接从输入内容中选择句子来撰写摘要,而抽象式摘要则是生成摘要不受输入内容的约束,提供了更大的灵活性,但给管理和控制带来了挑战。在这项工作中,由于资源和时间限制,我们专注于实现抽象摘要模型,并没有进一步进行实验来使用我们提出的算法开发提取摘要对应物。然而,值得注意的是,我们提出的方法已经显示出有希望的结果,强调了利用引文论文来增强语言模型在生成高质量生物医学摘要方面的性能的重要性。从理论上讲,来自引文论文的知识的聚合也有利于提取摘要方法。
其中 t 表示当前时间步长。 X 对应于输入,由提供给神经模型的 di 和 absc1、…、absjc 中的单词组成。 Ht 指的是解码器模块在时间步 t 的隐藏状态。该状态是由语言模型使用注入的特征 F 计算的,该特征封装了来自输入文档及其引用的摘要的信息,以及先前预测的标记 y3.4训练和推理

其中 L 是交叉熵损失,用于训练模型对令牌序列 P (yt|y四 实验效果
4.1数据集
数据集的创建
创建过程
为保证数据的质量和相关性,入选论文必须满足以下要求:
(1)论文必须包括“引言”部分,因为它被认为是生成摘要的最关键部分;
(2) 论文必须至少有3次不同的引用,以确保整理数据的质量;
(3) 论文的基本要素,包括 UID (Pubmed id)、标题、摘要、章节和引文,必须在原始语料库中可访问。
作为此构建过程的结果,数据集 D 包含 JsonL7 格式的结构化数据,每个样本代表一篇单独的论文。提取引文图
4.2 对比模型
我们将一系列具有竞争力的 PLM 模型作为我们的基准。我们还提供了两个基于规则的系统的结果,即 LEAD-3 和 ORACLE,作为代表模型性能上限和下限的基准。
LEAD-3从输入中提取前3个句子作为摘要,可以被视为性能的下限。
ORACLE从输入文档中选择句子并组成最高分的摘要8,这是提取式摘要系统的上限。 PLM 模型充当抽象生物医学总结的基线。
长文档 Transformer (LED) 是一种基于 Transformer 的模型,由于其自注意力操作,能够处理长序列(Beltagy 等人,2020)。
PEGASUS(Zhang 等人,2020)正在海量文本语料库上预训练基于 Transformer 的大型编码器-解码器模型,具有新的自我监督目标,该目标是为抽象文本摘要量身定制的。
BART(Lewis 等人,2019)是一种广泛使用的基于去噪自动编码器的 PLM 模型,已被证明对于长文本生成任务有效。
Pubmed-X 是指在大规模生物医学文献语料库 Pubmed (Cohan et al., 2018)(数据集大小为 215k)上预先训练的几个基于 PLM 的基线,其中 X 表示 PLM 的名称。
此外,我们还包括 ChatGPT 进行比较。然而,由于它是闭源的并且训练成本非常高,我们无法使用 ChatGPT 作为基础语言模型来对我们的数据集进行微调。相反,我们在零样本设置下比较 ChatGPT 的输出。4.3实施细节
测试集上的生成。批量大小设置为 16,学习率为 1e−4,选择 Adam 优化器进行训练。详细内容请参见附录A.1。4.4评估指标
4.5 实验结果
自动评估
此外,在我们的框架内,语言模型实现了显着较低的困惑度 (PPL) 和 ROUGE-L 分数,这意味着语言质量的提高并减少了摘要生成过程中的混乱。我们假设 PPL 和 ROUGE-L 的下降表明语言模型已经通过参考引用论文的摘要学习了写作风格和相关的生物医学术语。
关于消融研究,与基线模型 Pubmed-BART 相比,-w 一次引用产生了轻微的改进,但表现出更高的困惑度。这一观察结果表明,直接包含随机引用内容可能会引入一定的噪音。
相比之下,我们基于注意力的机制使神经网络能够动态选择和聚合来自多个引用的重要信息,有效解决与额外输入相关的混乱问题。在表 3 中,我们列出了其他评估指标的结果。 BertScore 和 BartScore 作为基于机器学习的指标,衡量生成的摘要和参考摘要之间的语义相似性。 Flesch-Kincaid 和 Coleman-Liau 指标评估词汇级别的文本可读性。在所有这些指标中,-w 引用聚合。优于所有基线模型,展示了使用我们的框架引入引文知识的优势。

“Pubmed-BART”模型中的进一步分析表明,使用单次引用会导致 BERTScore 和 BartScore 略有下降,同时 Flesch-Kincaid 分数 (14.78) 和 ColemanLiau 指数分数 (13.51) 略有上升。然而,采用引文聚合可以提高所有指标,包括 BERTScore (83.60)、BartScore (3.35)、Flesch-Kincaid 分数 (13.82) 和 ColemanLiau Index 分数 (13.20)。这一分析证实了我们最初的假设,即直接引入随机引用可能会引入影响模型性能的噪声,而我们的聚合模型综合考虑了所有引文论文,有效减少了单次引用引入的随机噪声。人工评价
深入分析
五 总结
局限性