【论文阅读笔记】PraNet: Parallel Reverse Attention Network for Polyp Segmentation
1. 论文介绍
PraNet: Parallel Reverse Attention Network for Polyp Segmentation
PraNet:用于息肉分割的并行反向注意力网络
2020年发表在MICCAI
Paper Code
2. 摘要
结肠镜检查是检测结直肠息肉的有效技术,结直肠息肉与结直肠癌高度相关。在临床实践中,从结肠镜图像中分割息肉是非常重要的,因为它为诊断和手术提供了有价值的信息。然而,由于两个主要原因,准确的息肉分割是一项具有挑战性的任务:(i)相同类型的息肉具有不同的大小、颜色和纹理;以及(ii)息肉与其周围粘膜之间的边界不清晰。为了解决这些挑战,我们提出了一种并行反向注意力网络(PraNet),用于结肠镜图像中的准确息肉分割。具体来说,我们首先使用并行部分解码器(PPD)聚合高级层中的特征。基于组合特征,我们然后生成全局地图作为以下组件的初始引导区域。此外,我们使用反向注意(RA)模块,它能够建立区域和边界线索之间的关系,挖掘边界线索。由于区域和边界之间的循环合作机制,我们的PraNet能够校准一些未对齐的预测,提高分割精度。针对五个具有挑战性的数据集进行的六项指标的定量和定性评估显示,我们的PraNet显著提高了分割准确性,并在可推广性和实时分割效率(每秒50帧)方面呈现出多项优势。
Keywords:反向注意力 部分解码器 息肉分割 实时分割 边界线索
3. Introduction
准确的息肉分割是一项具有挑战性的任务。因为1)息肉通常在外观上变化,例如,尺寸、颜色和质地,即使它们是同一类型。2)在结肠镜检查图像中,息肉及其周围粘膜之间的边界通常是模糊的,并且缺乏分割方法所需的强烈对比度。
在各种息肉分割方法中,基于早期学习的方法依赖于提取的手工特征,例如颜色,纹理,形状,外观或这些特征的组合。这些方法通常是训练分类器来区分息肉及其周围环境。然而,这些模型通常遭受高的误检率。主要原因是,当涉及到处理息肉的高类内变化和息肉与硬模仿物之间的低类间变化时,手工特征的表示能力非常有限。
定位息肉边界的一些工作:1.带有预训练模型的FCN来识别和分割息肉;2.用FCN的修改版本来提高息肉分割的准确性;3. U-Net,U-Net++ 和ResUNet++被用于息肉分割。
这些方法都集中在分割整个区域的息肉,但他们忽略了区域边界约束,这是非常关键的提高分割性能。1. Psi-Net 在息肉分割中同时利用面积和边界信息,但面积和边界之间的关系没有完全捕获。2. 一种具有区域和边界约束的三步选择性特征聚合网络用于息肉分割。该方法显式地考虑了区域和边界之间的依赖关系,并通过附加边缘监督获得了良好的结果;但是该方法耗时(>20小时),并且容易受到过拟合的破坏。
本文提出的一种新的深度神经网络,称为并行反向注意力网络(PraNet),用于实时准确的息肉分割任务。动机源于息肉注释,临床医生首先粗略定位息肉,然后准确地提取其轮廓掩模根据当地的特点。基于面积和边界是区分正常组织和息肉的两个关键特征。首先预测粗糙区域,然后通过反向注意隐式地对边界进行建模。该策略有三个优点,包括更好的学习能力,提高泛化能力和更高的训练效率。
通过使用并行部分解码器(PPD)聚合高级层中的特征,组合特征获取上下文信息并生成全局地图作为后续步骤的初始引导区域。为了进一步挖掘边界线索,利用一组经常性的反向注意(RA)模块,建立区域和边界线索之间的关系。由于区域和边界之间的这种经常性合作机制,该模型能够校准一些不一致的预测。
4.模型结构详解
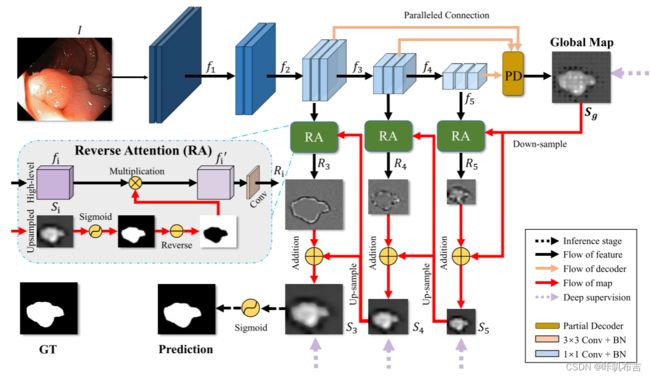
首先输入大小为 h ∗ w h*w h∗w的息肉图像I,经过基于Res2Net的骨干网络提取得到五层特征 f i , i = 1 , 2 , 3 , 4 , 5 ; [ h / 2 k − 1 , w / 2 k − 1 ] f_i,i=1,2,3,4,5;[h/2^k-1,w/2^k-1] fi,i=1,2,3,4,5;[h/2k−1,w/2k−1] ,其中1、2层为低层特征,3、4、5层为高层特征。高层特征通过部分解码器聚合,得到粗略的特征图 S g S_g Sg。 S g S_g Sg下采样与最高层特征 f 5 f_5 f5输入最高层特征的反向注意力模块,消除前景得到反向注意力特征 R 5 R_5 R5,即区域信息, R 5 R_5 R5再与 S g S_g Sg进行加运算得到 S 5 S_5 S5。 S 5 S_5 S5下采样与下一级特征反向注意力得到对应反向注意力特征,再与 S 5 S_5 S5下采样后的特征相加得到 S 4 S_4 S4,依次再得到 S 3 S_3 S3。取 S 3 S_3 S3特征进行Sigmoid激活函数激活得到预测结果。
PD部分解码器
部分解码器PD,以聚合(3,4,5)高级特征,最后得到全局映射 S g S_g Sg。 S g S_g Sg来自最深的CNN层,它只能捕获息肉组织的相对粗略的位置,而没有结构细节。参考Cascaded partial decoder for fast and accurate salient object detection 只整合三层高级特征。

这是参考文献的普通多级特征聚合网络如(a)所示,结构图如上(b),它整合高级特征(3、4、5)。
RA 反向注意力模块

高层特征不包含具体的边界信息,所以通过从高级侧输出特征中擦除现有的估计息肉区域来顺序地挖掘互补区域和细节,其中现有的估计是从更深层上采样的。
RA接收高层特征和更深层上采样的结果,将高级侧输出特征 f i , i = 3 , 4 , 5 {fi,i = 3,4,5} fi,i=3,4,5 乘以逐元素的反向注意力权重Ai来获得输出反向注意力特征Ri:

其中,反向注意力权重Ai:

其中,P(·) 表示上采样操作,σ(·) 是Sigmoid函数,而![]() (·)是从矩阵E中减去输入的逆操作,其中所有元素都是1。最终得到准确、完整的预测图。
(·)是从矩阵E中减去输入的逆操作,其中所有元素都是1。最终得到准确、完整的预测图。
损失函数
定义损失函数 L = L I o U w + L B C E w L = L^w_{IoU} +L^w_{BCE} L=LIoUw+LBCEw ,其中 L I o U w L^w_{IoU} LIoUw和 L B C E w L^w_{BCE} LBCEw表示全局限制和局部(像素级)限制的加权IoU损失和二进制交叉熵(BCE)损失。在这里,对三个侧输出(即,S3、S4和S4)和全局地图Sg 每个映射被上采样与地面实况图G的大小相同。因此,所提出的PraNet的总损失可以用公式表示为: L t o t a l = L ( G , S g u p ) + ∑ i = 3 i = 5 L ( G , S i u p ) L_{total} = L(G,S^{up}_g ) +\sum^{i=5}_{i=3} L(G,S^{up}_i ) Ltotal=L(G,Sgup)+∑i=3i=5L(G,Siup)。
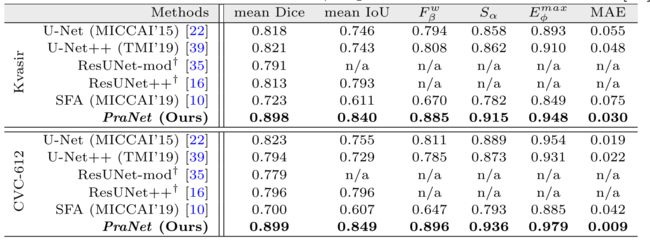
5. 实验与结果
实验基于PyTorch,使用NVIDIA TITAN RTX GPU加速。所有输入都统一调整为352×352,并采用多尺度训练策略{0.75,1,1.25}而不是数据增强。我们采用Adam优化算法来优化整体参数,学习率为 1 e − 4 1e−4 1e−4。整个网络以端到端的方式进行训练,需要32分钟才能收敛20个epoch,批量大小为16。我们的最终预测图 S p S_p Sp由 S 3 S_3 S3在S形运算之后生成。