HNU-数据挖掘-实验3-图深度学习
数据挖掘课程实验
实验3 图深度学习
文章目录
- 数据挖掘课程实验
实验3 图深度学习 -
- 实验背景
- 实验要求
- 数据集解析
- 实验内容
-
- (0)基础知识:基于图的深度学习方法
-
- 浅识:图卷积网络 (GCN)
- 浅识:图注意力网络 (GAT)
- 浅识:对抗生成网络 (GAN):
- 比较与选择
- 了解:图卷积网络(GCN)
- 了解:图注意力网络(GAT)
- (1)节点表征学习
-
- ①数据预处理与构建对象
- ②定义图自编码器模型
- ③模型训练
- ④绘制训练损失曲线
- ⑤绘制节点嵌入的散点图
- ⑥在绘制时加入节点密度计算
- (2)关联关系预测
-
- ①构建对象
- ②定义GAT模型
- ③模型训练
- ④预测效果分析
- ⑤可视化分析
-
- 散点图(Scatter Plot)
- **残差图(Residual Plot)**
- 综合分析
- (3)多关系图的联合学习
-
- 注意力头
- ①新增MultiHeadGATConv(nn.Module)类
- ②修改GraphAutoencoder(nn.Module)类
- ③初始化图自编码器模型
- ④测试:设置注意力头参数为1
- ⑤测试:设置注意力头参数为2
- 附录
-
- 节点表征学习代码 GAT1.py
- 关联关系预测代码 GAT2.py
- 多关系图的联合学习代码 GAT3.py
实验背景
深度学习(Deep Learning, DL),由Hinton等人于2006年提出,是机器学习(MachineLearning, ML)的一个新领域。深度学习是学习样本数据的内在规律和表示层次,这些学习过程中获得的信息对诸如文字、图像和声音等数据的解释有很大的帮助。它的最终目标是让机器能够像人一样具有分析学习能力,能够识别文字、图像和声音等数据。
实验要求
- 熟悉基于图的深度学习方法,比如图卷积网络、图注意力网络、对抗生成网络等。
- 熟练掌握图网络中节点的特征表示学习,并分析节点在某一任务下的表征差异,比如在SL_Human_FinalCheck关联关系中。
- 探索在联合多种关系图的特征表示学习,比如多通道卷积网络、多头注意力网络等,并分析其与单网络的特征表征差异,
- 实现以上数据分析的结果可视化。
数据集解析
共有4个数据集,其中
- List_Proteins_in_SL为基因列表,
- SL_Human_FinalCheck为基因关系,
- feature_ppi_128和feature_go_128为两种基因原始特征。
具体描述如下:
- List_Proteins_in_SL.txt:6375个基因,每一行为基因名称(英文表示)。
- SL_Human_FinalCheck.txt:总共有19667对基因关系,可以看作一种基因与基因之间的关联网络。共有19667行,每行第一列和第二列分别代表基因(英文表示),第三列代表置信分数。
- feature_ppi_128.txt:一种基因的原始特征,每一个基因有128维属性,每行为该基因的128个特征。
- feature_go_128.txt:一种基因的原始特征,每一个基因有128维属性,每行为该基因的128个特征。
可以较为简单地理解如下:
- 对于基因编号i,
List_Proteins_in_SL内保存了基因i对应的名称, - 对于基因编号i,j。
SL_Human_FinalCheck内保存了基因i和j的联系,该文件内的每一行都是某两个基因之间的联系以及该联系的置信分数, - 对于基因编号i,剩下两个以“feature”开头的文件的每一行有128列,每一列是刻画该基因的一个维度的特征值。可以理解为对于基因的刻画有两个角度(ppi和go),每个角度有128个维度的特征。这两个文件都各自有6375行,对应6375个基因。
实验内容
结合数据集提供的数据,本实验将按照如下所列出的点逐个完成:
- **节点表征学习:**使用图卷积网络 (GCN)、图注意力网络 (GAT)、对抗生成网络 (GAN) 等基于图的深度学习方法,通过学习基因关联网络中基因节点的表征。模型将尝试捕捉基因之间的关系,以便更好地理解和表示基因网络的结构。
- 关联关系预测: 通过学习基因节点的表征,模型将被应用于解决关联关系预测的问题。也就是说,给定两个基因,模型需要预测它们之间的关系,并输出一个置信分数。这可以帮助理解基因在生物学上的相互作用和功能。
- 多关系图的联合学习: 联合学习来自不同数据源的信息,包括基因关联网络 (SL_Human_FinalCheck) 和基因的原始特征图 (feature_ppi_128、feature_go_128)。这有助于更全面地理解基因的特征和相互关系。
同时还需要兼顾以下任务
- 结果可视化与解释性分析: 将学到的基因表征可视化,以便更好地理解模型在基因网络中的表现。此外,对模型进行解释性分析,理解模型是如何做出预测的,有助于增强对模型的信任和理解。
- 性能评估与对比分析: 对模型进行性能评估,比较不同模型和方法在关联关系预测任务上的效果。这可以帮助确定哪种方法更适合特定问题。
总体而言,构建一个深度学习模型,通过学习基因网络的结构和关联关系,以及联合学习不同数据源的信息,来解决基因关联关系预测的问题。通过实验和分析,更好地理解基因之间的关系,以及模型在这个生物学领域的应用潜力。
(0)基础知识:基于图的深度学习方法
根据实验提及,主要简要了解:图卷积网络、图注意力网络、对抗生成网络
浅识:图卷积网络 (GCN)
构建方式:
- 对于一个节点,GCN通过聚合其邻居的特征来更新节点的表示。这可以通过拉普拉斯矩阵的特征分解实现。
应用领域:
- 用于处理图结构数据,如社交网络、生物网络等。
- 典型任务包括节点分类、链接预测、社区发现等。
- GCN 能够在学习中考虑节点及其邻居之间的关系,适用于保留图的局部结构。
浅识:图注意力网络 (GAT)
构建方式:
- GAT 引入了注意力机制,允许节点关注邻居节点的不同程度,而不是平等对待所有邻居。
- 通过学习每个邻居节点的权重,GAT能够更灵活地捕捉图中节点之间的关系。
应用领域:
- GAT 适用于处理异构图或者图中节点之间的关系强度不同的情况。
- 能够更灵活地捕捉图中的局部结构,适用于节点分类、图分类等任务。
浅识:对抗生成网络 (GAN):
构建:
- GAN 由生成器(Generator)和判别器(Discriminator)组成。
- 生成器试图生成与真实数据相似的数据,而判别器则试图区分生成的数据和真实数据。
- GAN 通过对抗训练来优化生成器和判别器的参数。
应用领域:
- GAN 主要用于生成新的数据样本,如图像、文本等。
- 典型应用包括图像生成、风格迁移、超分辨率等。
- GAN 的生成器能够学习数据的分布,生成具有真实样本特征的新样本。
比较与选择
可以从输入与输出对这三种算法进行比较
输入:
- GCN 和 GAT 的输入包括图的邻接矩阵和节点特征矩阵。
- GAN 的输入是噪声向量,通过生成器产生虚构的数据。
输出:
- GCN 和 GAT 的输出是更新后的节点表示,可以用于节点分类、图分类等任务。
- GAN 的输出是生成器生成的数据,目标是尽量接近真实数据分布。
总体来说,这三种网络分别解决了不同类型的问题。GCN 和 GAT 处理图结构数据,用于节点级和图级的任务;GAN 专注于生成与真实数据相似的新数据。
结合实验题目来说,使用GCN与GAT来构建似乎更加合适。而实际上,GAT可以看作是在GCN上引入注意力机制进行改进,在以下的几个方面会有不同。
- 注意力机制:
- 主要的创新在于 GAT 引入了注意力机制,这使得每个节点能够对其邻居节点分配不同的权重。这意味着在信息传递的过程中,模型可以更灵活地关注那些对当前节点更重要的邻居节点。
- 权重的计算:
- 在 GCN 中,所有邻居节点的权重是相同的,通过图卷积操作对邻居节点进行平均聚合。
- 在 GAT 中,权重是通过学习到的注意力系数动态计算的,每个邻居节点都有一个相关的权重,使得模型可以更细致地处理不同节点之间的关系。
- 灵活性:
- GAT 更加灵活,能够适应异构图或者图中不同节点之间关系强度不同的情况。
- 在 GCN 中,所有邻居节点被视为等权重的,这可能在一些场景下不够灵活。
- 参数量:
- GAT 中引入了注意力机制,因此其参数量相对较大。每个节点对应的注意力权重需要学习,而在 GCN 中每个节点的权重是相同的,参数量相对较少。
综上比较,我们选择引入了注意力机制的GAT来进行下面实验的探索。
了解:图卷积网络(GCN)
详细可看该网站,深入浅出地讲解了GCN大概是什么。
https://zhuanlan.zhihu.com/p/71200936
简要来说,GCN能很好地描述不同点各自的特征和它与它们邻居的关系。
了解:图注意力网络(GAT)
GAT(GRAPH ATTENTION NETWORKS)是一种使用了self attention机制图神经网络,该网络使用类似transformer里面self attention的方式计算图里面某个节点相对于每个邻接节点的注意力,将节点本身的特征和注意力特征concate起来作为该节点的特征,在此基础上进行节点的分类等任务。
★节点的特征由节点本身和直接相连的节点共同决定
GAT使用了类似的流程计算节点的self attention,首先计算当前节点和每个邻接节点的注意力score,然后使用该score乘以每个节点的特征,累加起来并经过一个非线性映射,作为当前节点的特征。
这里使用W矩阵将原始的特征映射到一个新的空间,a代表self attention的计算,如前面图2所示,这样计算出两个邻接节点的attention score,也就是Eij,然后对所有邻接节点的score进行softmax处理,得到归一化的attention score。
(1)节点表征学习
本部分的任务是图网络中节点的特征表示学习,并分析节点在SL_Human_FinalCheck关联关系中的表征差异。
本部分用到的数据有
- List_Proteins_in_SL(基因列表)
- SL_Human_FinalCheck(基因关系)
①数据预处理与构建对象
主要做的是从数据集中读出数据并按照结构的形式保存。
从文件中读取基因列表和基因关系,并将基因名称映射为数字标识。然后,构建图的 Data 对象,其中每个基因表示为一个节点,基因关系表示为图的边。
# 读取基因列表
genes_df = pd.read_csv('List_Proteins_in_SL.txt', header=None, names=['Gene'])
genes = genes_df['Gene'].tolist()
# 读取基因关系
relations_df = pd.read_csv('SL_Human_FinalCheck.txt', sep='\t', header=None, names=['Gene1', 'Gene2', 'Confidence'])
relations_df['Confidence'] = relations_df['Confidence'].astype(float)
# 将基因名称映射为数字标识
genes_dict = {gene: idx for idx, gene in enumerate(genes)}
relations_df['Gene1'] = relations_df['Gene1'].map(genes_dict)
relations_df['Gene2'] = relations_df['Gene2'].map(genes_dict)
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
data = Data(x=x, edge_index=edge_index)
②定义图自编码器模型
GraphAutoencoder 是一个简单的图自编码器模型,使用 GATConv 作为编码器和解码器。该模型的目标是将节点嵌入压缩到较低维度,然后再重构回原始维度。
# 定义图自编码器模型
class GraphAutoencoder(nn.Module):
def __init__(self, in_features, hidden_size):
super(GraphAutoencoder, self).__init__()
self.encoder = GATConv(in_features, hidden_size, heads=1)
self.decoder = GATConv(hidden_size, in_features, heads=1)
def forward(self, data):
encoded = self.encoder(data.x, data.edge_index)
decoded = self.decoder(encoded, data.edge_index)
return decoded
③模型训练
使用均方误差损失函数进行无监督训练,通过最小化嵌入节点的重构误差来学习图的表示。
# 无监督学习任务:重构损失
num_epochs = 150
losses = [] # 用于存储每个epoch的损失值
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
reconstructed = model(data)
# 选择适当的无监督损失函数,比如均方误差损失
loss = nn.MSELoss()(reconstructed, data.x)
loss.backward()
optimizer.step()
losses.append(loss.item()) # 记录每个epoch的损失值
print(f'Epoch: {epoch + 1}, Loss: {loss.item()}')
④绘制训练损失曲线
通过观察训练损失曲线率,可以更好地观察大概在什么地方开始收敛。便于观察是否可能过拟合或欠拟合。
# 绘制损失率的曲线图
plt.plot(losses, label='Training Loss')
plt.xlabel('Epoch')
plt.ylabel('Loss')
plt.title('Training Loss Over Time')
plt.legend()
plt.show()
比如,我们调节num_epochs = 150,进行150轮训练,并绘制训练损失曲线如下,
显然在80-100的时候就已经收敛了,没必要到150层,那我们可以修改num_epochs = 100。
感觉还是有点多了,观察详细数值可以发现,在80层左右之后大概lossrate就在10^-5之下了,所以可以取num_epochs = 80,试试效果。
感觉这样是比较合适的,就按num_epochs = 80来。
⑤绘制节点嵌入的散点图
首先提取节点嵌入
# 在训练后提取节点嵌入
with torch.no_grad():
node_embeddings = model.encoder(data.x, data.edge_index).numpy()
然后我们试图绘制图像
plt.scatter(node_embeddings[:, 0], node_embeddings[:, 1], alpha=0.7, c='blue', marker='.')
plt.title('节点嵌入')
plt.show()
绘制图像如下:
感觉有点杂乱,而且这乍看似乎没有包含所有的点(数量有点少了)
⑥在绘制时加入节点密度计算
试试看计算节点密度并使用颜色来表示。
# 计算节点密度
node_density = np.zeros(len(genes))
for i in range(len(genes)):
# 计算每个节点到其他节点的欧氏距离
distances = np.linalg.norm(node_embeddings - node_embeddings[i], axis=1)
# 计算距离小于某个阈值的节点数,可以根据需要调整阈值
density = np.sum(distances < 0.0000001)
node_density[i] = density
# 绘制节点嵌入的散点图,并使用节点密度作为颜色映射
plt.scatter(node_embeddings[:, 0], node_embeddings[:, 1], alpha=0.7, c=node_density, cmap='viridis', marker='.')
plt.colorbar(label='Node Density')
plt.title('节点嵌入(根据节点密度着色)')
plt.show()
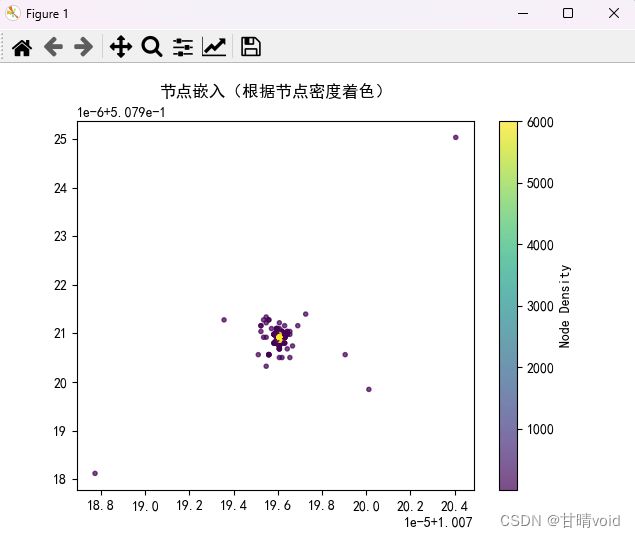
绘制图像如下:
颜色对应于该节点周围节点数量的多少(即密度)
可以看见中间部分节点的密度很高很高,实际上节点数量并没有减少。
(2)关联关系预测
目标不仅仅是节点表征学习,此时需要进行回归任务了。所以需要一些改动。
数据预处理部分与之前一致,其他部分需要改动。
①构建对象
这是之前节点表征学习时构建对象
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
data = Data(x=x, edge_index=edge_index)
但是现在我们的目标变化了,改动如下:
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
y = torch.tensor(relations_df['Confidence'].values, dtype=torch.float).view(-1, 1) # 置信分数作为目标
data = Data(x=x, edge_index=edge_index, y=y)
具体含义如下:
- 基因特征 (
x) 初始化为一维张量。 - 边索引 (
edge_index) 表示基因之间的连接。 - 目标值 (
y) 是置信度分数。
主要就是引入了置信分数。
②定义GAT模型
定义图图模型也要做相应的变化
# 定义GAT模型
class GATModel(nn.Module):
def __init__(self, in_features, out_features, num_heads):
super(GATModel, self).__init__()
self.conv1 = GATConv(in_features, out_features, heads=num_heads)
def forward(self, data):
x = self.conv1(data.x, data.edge_index)
return x
③模型训练
这里也需要做相应的改动
# 训练模型(回归任务)
num_epochs = 200
losses = [] # 用于存储每个epoch的损失值
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
output = model(data)
# 使用均方误差损失
loss = loss_function(output[train_mask], data.y[train_mask])
loss.backward()
optimizer.step()
losses.append(loss.item()) # 记录每个epoch的损失值
print(f'Epoch: {epoch + 1}, Loss: {loss.item()}')
模型训练的时候要根据训练损失曲线以及结果的Loss值及时调整训练层数。
这里我发现num_epochs = 200时效果较好,差不多刚刚收敛。
④预测效果分析
使用RMSE(均方根误差) 和MAE(平均绝对误差)来对预测效果进行衡量。
代码如下:
# 评估模型性能
# 在测试集上计算性能指标
from sklearn.metrics import mean_squared_error, mean_absolute_error
model.eval()
with torch.no_grad():
test_output = model(data)
# 将tensor转换为numpy数组
test_output_np = test_output.cpu().numpy()
y_true_np = data.y[test_mask].cpu().numpy()
# 将test_output_np和y_true_np调整为相同的样本数
test_output_np = test_output[test_mask].cpu().numpy()
# 计算均方根误差和平均绝对误差
rmse = np.sqrt(mean_squared_error(y_true_np, test_output_np))
mae = mean_absolute_error(y_true_np, test_output_np)
print(f'Root Mean Squared Error (RMSE): {rmse}')
print(f'Mean Absolute Error (MAE): {mae}')
评估结果如下:
- Root Mean Squared Error (RMSE): 0.16360943019390106
- Mean Absolute Error (MAE): 0.15059898793697357
一般来说,认为该结果是可以接受的。
⑤可视化分析
使用可视化分析绘制散点图与残差图进行分析
# 散点图(Scatter Plot)
plt.scatter(y_true_np, test_output_np)
plt.xlabel('Actual Values')
plt.ylabel('Predicted Values')
plt.title('Scatter Plot of Actual vs Predicted Values')
plt.show()
# 残差图(Residual Plot)
residuals = y_true_np - test_output_np
plt.scatter(y_true_np, residuals)
plt.xlabel('Actual Values')
plt.ylabel('Residuals')
plt.axhline(y=0, color='r', linestyle='--', linewidth=2) # 添加水平线表示残差为零
plt.title('Residual Plot')
plt.show()
图像如下:
散点图(Scatter Plot)
- 横轴(X轴): 实际观测值(Ground Truth)。
- 纵轴(Y轴): 模型的预测值。
- 每个点: 表示数据集中的一个样本,其中横坐标是实际的观测值,纵坐标是模型的预测值。
- 含义: 散点图的趋势应该是趋近于一条对角线,表示模型的预测值与实际观测值一致。如果散点分布离散,可能意味着模型在某些情况下表现不佳。
残差图(Residual Plot)
- 横轴(X轴): 模型的预测值。
- 纵轴(Y轴): 残差(实际观测值与模型预测值之间的差异)。
- 每个点: 表示一个样本,其中横坐标是模型的预测值,纵坐标是该样本的残差。
- 含义: 残差图用于检查模型是否存在系统性的预测误差。如果残差图呈现出一种无规律的散点分布,说明模型的预测是准确的。如果出现某种模式,可能表示模型在某个范围或条件下存在系统性的预测偏差。
综合分析
从散点图与残差图分析来看,这个关联关系预测的效果其实不太好。
(3)多关系图的联合学习
多关系图的联合学习在前面的节点表征学习基础上进行。不同点主要在于两点,要引入go和ppi这两个各自128维的特征,还有就是要考虑多头注意网络的构建。
下面是在节点表征学习的基础上新增的部分。
注意力头
注意力头是一个超参数,影响模型的表示能力,计算效率,泛化性能和稳健性。每个注意力头负责学习一种节点之间的关系,多个头可以捕捉更丰富和复杂的关系。
①新增MultiHeadGATConv(nn.Module)类
class MultiHeadGATConv(nn.Module):
def __init__(self, in_features, out_features, heads):
super(MultiHeadGATConv, self).__init__()
self.heads = heads
self.attention_heads = nn.ModuleList([GATConv(in_features, out_features, heads=1) for _ in range(heads)])
self.out_linear = nn.Linear(heads * out_features, out_features) # 添加线性层
def forward(self, x, edge_index):
# 各个注意力头的输出
head_outputs = [attention_head(x, edge_index) for attention_head in self.attention_heads]
# 拼接所有注意力头的输出
x = torch.cat(head_outputs, dim=1)
# 使用线性层调整输出维度
x = F.relu(self.out_linear(x))
return x
②修改GraphAutoencoder(nn.Module)类
class GraphAutoencoder(nn.Module):
def __init__(self, in_features, hidden_size, heads):
super(GraphAutoencoder, self).__init__()
self.encoder = MultiHeadGATConv(in_features, hidden_size, heads=heads)
self.decoder = MultiHeadGATConv(hidden_size, in_features, heads=heads)
def forward(self, data):
encoded = self.encoder(data.x, data.edge_index)
decoded = self.decoder(encoded, data.edge_index)
return decoded
③初始化图自编码器模型
model = GraphAutoencoder(in_features=257, hidden_size=32, heads=1)
④测试:设置注意力头参数为1
这是引入feature_ppi_128和feature_go_128这两个128维度特征的情况,但是还没有设置注意力头参数,即还没有启动多头注意力网络。
设置隐藏层为2,开始训练。
呈现节点密度嵌入图如下
可以发现跟之前有很大区别,首先是点变多了,然后颜色变深了,这意味着每个点所代表的实际基因个数变少了,基因下嵌入图变得分散了。
其实这很好理解,因为特征变多了,它们之间的差异逐渐变得很大,不同的点之间肯定会变得更加分散。因此同一个点附近密度变小,颜色变深。
⑤测试:设置注意力头参数为2
这是启用多头注意力网络,在这种情况下,我们要适当增加隐藏层数,获得结果如下:
训练损失率随轮次变化图,可见在60次左右应该已经收敛。
节点嵌入图如下,颜色为深色,但点都附着在其中一条轴上,这个可能与输出维度使用了线性层有关,也可能与数据本身有关,也可能与绘制图像时的比例尺有关。对于该点没有进行更加深入的探究。
附录
节点表征学习代码 GAT1.py
import numpy as np
import pandas as pd
import torch
import torch.nn as nn
import torch.optim as optim
from torch_geometric.data import Data
from torch_geometric.nn import GATConv
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
# 读取基因列表
genes_df = pd.read_csv('List_Proteins_in_SL.txt', header=None, names=['Gene'])
genes = genes_df['Gene'].tolist()
# 读取基因关系
relations_df = pd.read_csv('SL_Human_FinalCheck.txt', sep='\t', header=None, names=['Gene1', 'Gene2', 'Confidence'])
relations_df['Confidence'] = relations_df['Confidence'].astype(float)
# 将基因名称映射为数字标识
genes_dict = {gene: idx for idx, gene in enumerate(genes)}
relations_df['Gene1'] = relations_df['Gene1'].map(genes_dict)
relations_df['Gene2'] = relations_df['Gene2'].map(genes_dict)
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
data = Data(x=x, edge_index=edge_index)
# 定义图自编码器模型
class GraphAutoencoder(nn.Module):
def __init__(self, in_features, hidden_size):
super(GraphAutoencoder, self).__init__()
self.encoder = GATConv(in_features, hidden_size, heads=1)
self.decoder = GATConv(hidden_size, in_features, heads=1)
def forward(self, data):
encoded = self.encoder(data.x, data.edge_index)
decoded = self.decoder(encoded, data.edge_index)
return decoded
# 初始化图自编码器模型
model = GraphAutoencoder(in_features=1, hidden_size=2)
# 定义优化器
optimizer = optim.Adam(model.parameters(), lr=0.01)
# 无监督学习任务:重构损失
num_epochs = 80
losses = [] # 用于存储每个epoch的损失值
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
reconstructed = model(data)
# 选择适当的无监督损失函数,比如均方误差损失
loss = nn.MSELoss()(reconstructed, data.x)
loss.backward()
optimizer.step()
losses.append(loss.item()) # 记录每个epoch的损失值
print(f'Epoch: {epoch + 1}, Loss: {loss.item()}')
# 在测试集上测试模型
model.eval()
with torch.no_grad():
test_output = model(data)
# 绘制损失率的曲线图
plt.plot(losses, label='Training Loss')
plt.xlabel('Epoch')
plt.ylabel('Loss')
plt.title('Training Loss Over Time')
plt.legend()
plt.show()
# 在训练后提取节点嵌入
with torch.no_grad():
node_embeddings = model.encoder(data.x, data.edge_index).numpy()
# 绘制节点嵌入的散点图
plt.rcParams['font.sans-serif'] = ['SimHei'] # 替换为系统中存在的中文字体
plt.rcParams['axes.unicode_minus'] = False # 解决设置中文后符号无法显示的问题
"""
plt.scatter(node_embeddings[:, 0], node_embeddings[:, 1], alpha=0.7, c='blue', marker='.')
plt.title('节点嵌入')
plt.show()
"""
# 计算节点密度
node_density = np.zeros(len(genes))
for i in range(len(genes)):
# 计算每个节点到其他节点的欧氏距离
distances = np.linalg.norm(node_embeddings - node_embeddings[i], axis=1)
# 计算距离小于某个阈值的节点数,可以根据需要调整阈值
density = np.sum(distances < 0.0000001)
node_density[i] = density
# 绘制节点嵌入的散点图,并使用节点密度作为颜色映射
plt.scatter(node_embeddings[:, 0], node_embeddings[:, 1], alpha=0.7, c=node_density, cmap='viridis', marker='.')
plt.colorbar(label='Node Density')
plt.title('节点嵌入(根据节点密度着色)')
plt.show()
关联关系预测代码 GAT2.py
import numpy as np
import pandas as pd
import torch
import torch.nn as nn
import torch.optim as optim
from torch_geometric.data import Data
from torch_geometric.nn import GATConv
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
# 读取基因列表
genes_df = pd.read_csv('List_Proteins_in_SL.txt', header=None, names=['Gene'])
genes = genes_df['Gene'].tolist()
# 读取基因关系
relations_df = pd.read_csv('SL_Human_FinalCheck.txt', sep='\t', header=None, names=['Gene1', 'Gene2', 'Confidence'])
relations_df['Confidence'] = relations_df['Confidence'].astype(float)
# 将基因名称映射为数字标识
genes_dict = {gene: idx for idx, gene in enumerate(genes)}
relations_df['Gene1'] = relations_df['Gene1'].map(genes_dict)
relations_df['Gene2'] = relations_df['Gene2'].map(genes_dict)
# 移除带有非数字标识的行
relations_df = relations_df.dropna(subset=['Gene1', 'Gene2', 'Confidence'])
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
y = torch.tensor(relations_df['Confidence'].values, dtype=torch.float).view(-1, 1) # 置信分数作为目标
data = Data(x=x, edge_index=edge_index, y=y)
# 定义GAT模型
class GATModel(nn.Module):
def __init__(self, in_features, out_features, num_heads):
super(GATModel, self).__init__()
self.conv1 = GATConv(in_features, out_features, heads=num_heads)
def forward(self, data):
x = self.conv1(data.x, data.edge_index)
return x
# 初始化模型
model = GATModel(in_features=1, out_features=1, num_heads=1)
# 定义优化器
optimizer = optim.Adam(model.parameters(), lr=0.01)
# 回归任务的损失函数
loss_function = nn.MSELoss()
# 划分训练集和测试集
train_mask, test_mask = train_test_split(range(len(genes)), test_size=0.2, random_state=42)
# 训练模型(回归任务)
num_epochs = 200
losses = [] # 用于存储每个epoch的损失值
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
output = model(data)
# 使用均方误差损失
loss = loss_function(output[train_mask], data.y[train_mask])
loss.backward()
optimizer.step()
losses.append(loss.item()) # 记录每个epoch的损失值
print(f'Epoch: {epoch + 1}, Loss: {loss.item()}')
# 在测试集上测试模型
model.eval()
with torch.no_grad():
test_output = model(data)
# 绘制损失率的曲线图
plt.plot(losses, label='Training Loss')
plt.xlabel('Epoch')
plt.ylabel('Loss')
plt.title('Training Loss Over Time')
plt.legend()
plt.show()
# 评估模型性能
# 在测试集上计算性能指标
from sklearn.metrics import mean_squared_error, mean_absolute_error
model.eval()
with torch.no_grad():
test_output = model(data)
# 将tensor转换为numpy数组
test_output_np = test_output.cpu().numpy()
y_true_np = data.y[test_mask].cpu().numpy()
# 将test_output_np和y_true_np调整为相同的样本数
test_output_np = test_output[test_mask].cpu().numpy()
# 计算均方根误差和平均绝对误差
rmse = np.sqrt(mean_squared_error(y_true_np, test_output_np))
mae = mean_absolute_error(y_true_np, test_output_np)
print(f'Root Mean Squared Error (RMSE): {rmse}')
print(f'Mean Absolute Error (MAE): {mae}')
# 散点图(Scatter Plot)
plt.scatter(y_true_np, test_output_np)
plt.xlabel('Actual Values')
plt.ylabel('Predicted Values')
plt.title('Scatter Plot of Actual vs Predicted Values')
plt.show()
# 残差图(Residual Plot)
residuals = y_true_np - test_output_np
plt.scatter(y_true_np, residuals)
plt.xlabel('Actual Values')
plt.ylabel('Residuals')
plt.axhline(y=0, color='r', linestyle='--', linewidth=2) # 添加水平线表示残差为零
plt.title('Residual Plot')
plt.show()
多关系图的联合学习代码 GAT3.py
import numpy as np
import pandas as pd
import torch
import torch.nn as nn
import torch.optim as optim
from torch_geometric.data import Data
from torch_geometric.nn import GATConv
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
import torch.nn.functional as F
# 读取基因列表
genes_df = pd.read_csv('List_Proteins_in_SL.txt', header=None, names=['Gene'])
genes = genes_df['Gene'].tolist()
# 读取基因关系
relations_df = pd.read_csv('SL_Human_FinalCheck.txt', sep='\t', header=None, names=['Gene1', 'Gene2', 'Confidence'])
relations_df['Confidence'] = relations_df['Confidence'].astype(float)
# 将基因名称映射为数字标识
genes_dict = {gene: idx for idx, gene in enumerate(genes)}
relations_df['Gene1'] = relations_df['Gene1'].map(genes_dict)
relations_df['Gene2'] = relations_df['Gene2'].map(genes_dict)
# 构建图的 Data 对象
edge_index = torch.tensor(relations_df[['Gene1', 'Gene2']].values.T.astype(np.int64), dtype=torch.long)
x = torch.ones((len(genes), 1)) # 每个基因有一个1维的特征
data = Data(x=x, edge_index=edge_index)
# 读取额外的基因特征
feature_ppi = pd.read_csv('feature_ppi_128.txt', header=None, sep='\s+')
feature_go = pd.read_csv('feature_go_128.txt', header=None, sep='\s+')
# 将额外的特征合并到现有数据中
data.x = torch.cat([data.x, torch.tensor(feature_ppi.values, dtype=torch.float32), torch.tensor(feature_go.values, dtype=torch.float32)], dim=1)
# 修改 GATConv 层为 MultiHeadGATConv
class MultiHeadGATConv(nn.Module):
def __init__(self, in_features, out_features, heads):
super(MultiHeadGATConv, self).__init__()
self.heads = heads
self.attention_heads = nn.ModuleList([GATConv(in_features, out_features, heads=1) for _ in range(heads)])
self.out_linear = nn.Linear(heads * out_features, out_features) # 添加线性层
def forward(self, x, edge_index):
# 各个注意力头的输出
head_outputs = [attention_head(x, edge_index) for attention_head in self.attention_heads]
# 拼接所有注意力头的输出
x = torch.cat(head_outputs, dim=1)
# 使用线性层调整输出维度
x = F.relu(self.out_linear(x))
return x
# 修改 GraphAutoencoder 类
class GraphAutoencoder(nn.Module):
def __init__(self, in_features, hidden_size, heads):
super(GraphAutoencoder, self).__init__()
self.encoder = MultiHeadGATConv(in_features, hidden_size, heads=heads)
self.decoder = MultiHeadGATConv(hidden_size, in_features, heads=heads)
def forward(self, data):
encoded = self.encoder(data.x, data.edge_index)
decoded = self.decoder(encoded, data.edge_index)
return decoded
# 初始化图自编码器模型
model = GraphAutoencoder(in_features=257, hidden_size=64, heads=2)
# 定义优化器
optimizer = optim.Adam(model.parameters(), lr=0.01)
# 无监督学习任务:重构损失
num_epochs = 60
losses = [] # 用于存储每个epoch的损失值
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
reconstructed = model(data)
# 选择适当的无监督损失函数,比如均方误差损失
loss = nn.MSELoss()(reconstructed, data.x)
loss.backward()
optimizer.step()
losses.append(loss.item()) # 记录每个epoch的损失值
print(f'Epoch: {epoch + 1}, Loss: {loss.item()}')
# 在测试集上测试模型
model.eval()
with torch.no_grad():
test_output = model(data)
# 绘制损失率的曲线图
plt.plot(losses, label='Training Loss')
plt.xlabel('Epoch')
plt.ylabel('Loss')
plt.title('Training Loss Over Time')
plt.legend()
plt.show()
# 在训练后提取节点嵌入
with torch.no_grad():
node_embeddings = model.encoder(data.x, data.edge_index).numpy()
plt.rcParams['font.sans-serif'] = ['SimHei'] # 替换为系统中存在的中文字体
plt.rcParams['axes.unicode_minus'] = False # 解决设置中文后符号无法显示的问题
# 计算节点密度
node_density = np.zeros(len(genes))
for i in range(len(genes)):
# 计算每个节点到其他节点的欧氏距离
distances = np.linalg.norm(node_embeddings - node_embeddings[i], axis=1)
# 计算距离小于某个阈值的节点数,可以根据需要调整阈值
density = np.sum(distances < 0.0000001)
node_density[i] = density
# 绘制节点嵌入的散点图,并使用节点密度作为颜色映射
plt.scatter(node_embeddings[:, 0], node_embeddings[:, 1], alpha=0.7, c=node_density, cmap='viridis', marker='.')
plt.colorbar(label='Node Density')
plt.title('节点嵌入(根据节点密度着色)')
plt.show()