Dimensionality Reduction(学习Free Mind知识整理)
阅读http://blog.pluskid.org/?p=290文章中的一些知识整理:学习的知识点降维。
=====================================================================
先看一下这句话“feature selection 和 dimensionality reduction 两个 topic 都有许多很经典的算法。前者通常是选出重要的 feature 的维度(并抛弃不重要的维度),而后者则是更广泛意义上的把一个高维的向量映射为一个低维向量,亦即“降维”,得到的结果 feature 的值已经不一定是原始的值,也可以把 feature selection 看作是 dimensionality reduction 的一种特殊情况。”(阉割和压缩的区别 -----网友解释)
我认为特征选择======>取其精华,去其糟粕;数据降维 ===>合理的信息丢失的范围里面 将原来的高维数据 映射到 一个低维空间里面 这样可以降低模型的复杂度和计算的复杂度 减少过拟合和增加泛化的能力,数据降维后不一定就直接按照降维后的维度作为特征的维度。
因为经常会遇到论文里面的泛化能力的评估,一直不是很清楚泛化能力怎么增强?什么鬼。。。。
所谓泛化能力(generalization ability)是指对新鲜样本的适应能力,对于测试网络模型的泛化能力啊算法的泛化能力啊,这个算法或者模型能有对新来的数据有好的准确率或者学习率,可能解释不是很到位,大概就是这个意思吧。
然后就是一个网络的泛化能力增强?
1、较多的输入样本但不是太多,过多的样本导致过度拟合,泛化能力不佳;样本包括至少一次的转折点数据。
2、隐含层神经元数量的选择,不影响性能的前提下,尽量选择小一点的神经元数量。隐含层节点太多,造成泛化能力下降,造火箭也只要几十个到几百个神经元,拟合几百几千个数据何必要那么多神经元?
3、误差小,则泛化能力好;误差太小,则会过度拟合,泛化能力反而不佳。
4、学习率的选择,特别是权值学习率,对网络性能有很大影响,太小则收敛速度很慢,且容易陷入局部极小化;太大则,收敛速度快,但易出现摆动,误差难以缩小;一般权值学习率比要求误差稍微稍大一点点;另外可以使用变动的学习率,在误差大的时候增大学习率,等误差小了再减小学习率,这样可以收敛更快,学习效果更好,不易陷入局部极小化。
5、训练时可以采用随时终止法,即是误差达到要求即终止训练,以免过度拟合;可以调整局部权值,使局部未收敛的加快收敛。
====================================================================
2.使用中值和方差进行充分统计。
3.大方差向量具有较大重要性。
4.主元正交。PCA方法假设主元向量之间都是正交的,从而可以利用线形代数的一系列有效的数学工具进行求解,大大提高了效率和应用的范围。
它的直观思想是希望相互间有关系的点(在图中相连的点)在降维后的空间中尽可能的靠近。Laplacian Eigenmaps可以反映出数据内在的流形结构。
Laplacian Eigenmaps也通过构建相似关系图(对应的矩阵为![]() )来重构数据流形的局部结构特征。Laplacian Eigenmaps算法的主要思想是,如果两个数据实例i和j很相似,那么i和j在降维后目标子空间中应该尽量接近。设数据实例的数目为n,目标子空间的维度为m。定义
)来重构数据流形的局部结构特征。Laplacian Eigenmaps算法的主要思想是,如果两个数据实例i和j很相似,那么i和j在降维后目标子空间中应该尽量接近。设数据实例的数目为n,目标子空间的维度为m。定义![]() 大小的矩阵
大小的矩阵![]() ,其中每一个行向量是数据实例i在目标m维子空间中的向量表示,Laplacian Eigenmaps要优化的目标函数如下
,其中每一个行向量是数据实例i在目标m维子空间中的向量表示,Laplacian Eigenmaps要优化的目标函数如下
(2.11)
定义对角矩阵![]() ,对角线上
,对角线上![]() 位置元素等于矩阵
位置元素等于矩阵![]() 的第i行之和,经过线性代数变换,上述优化问题可以用矩阵向量形式表示如下:
的第i行之和,经过线性代数变换,上述优化问题可以用矩阵向量形式表示如下:
(2.12)
其中矩阵![]() 是图拉普拉斯矩阵。限制条件保证优化问题有解,并且保证映射后的数据点不会被“压缩”到一个小于m维的子空间中。使得公式最小化的Y的列向量是以下广义特征值问题的m个最小非0特征值(包括重根)对应的特征向量:
是图拉普拉斯矩阵。限制条件保证优化问题有解,并且保证映射后的数据点不会被“压缩”到一个小于m维的子空间中。使得公式最小化的Y的列向量是以下广义特征值问题的m个最小非0特征值(包括重根)对应的特征向量:
(2.13)
使用时算法具体步骤为:
步骤1:构建图
使用某一种方法来将所有的点构建成一个图,例如使用KNN算法,将每个点最近的K个点连上边。K是一个预先设定的值。
步骤2:确定权重
确定点与点之间的权重大小,例如选用热核函数来确定,如果点i和点j相连,那么它们关系的权重设定为:
另外一种可选的简化设定是![]() 如果点i,j相连,否则
如果点i,j相连,否则![]() 。
。
步骤3:特征映射
计算拉普拉斯矩阵L的特征向量与特征值:![]()
其中D是对角矩阵,满足,![]() 。
。
使用最小的m个非零特征值对应的特征向量作为降维后的结果输出。
前面提到过,Laplacian Eigenmap具有区分数据点的特性,可以从下面的例子看出:
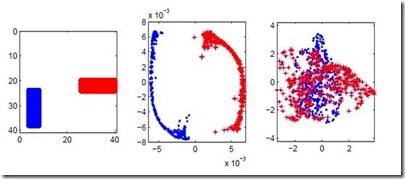
图1 Laplacian Eigenmap实验结果
见图1所示,左边的图表示有两类数据点(数据是图片),中间图表示采用Laplacian Eigenmap降维后每个数据点在二维空间中的位置,右边的图表示采用PCA并取前两个主要方向投影后的结果,可以清楚地看到,在此分类问题上,Laplacian Eigenmap的结果明显优于PCA。
图2 roll数据的降维
图2说明的是,高维数据(图中3D)也有可能是具有低维的内在属性的(图中roll实际上是2D的),但是这个低维不是原来坐标表示,例如如果要保持局部关系,蓝色和下面黄色是完全不相关的,但是如果只用任何2D或者3D的距离来描述都是不准确的。
% Laplacian Eigenmap ALGORITHM (using K nearest neighbors)
%
% [Y] = le(X,K,dmax)
%
% X = data as D x N matrix (D = dimensionality, N = #points)
% K = number of neighbors
% dmax = max embedding dimensionality
% Y = embedding as dmax x N matrix
%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
function [Y] = leigs(X,K,d)
[D,N] = size(X);
fprintf(1,'LE running on %d points in %d dimensions\n',N,D);
% STEP1: COMPUTE PAIRWISE DISTANCES & FIND NEIGHBORS
fprintf(1,'-->Finding %d nearest neighbours.\n',K);
X2 = sum(X.^2,1);
distance = repmat(X2,N,1)+repmat(X2',1,N)-2*X'*X;
[sorted,index] = sort(distance);
neighborhood = index(2:(1+K),:);
% STEP2: Construct similarity matrix W
fprintf(1,'-->Constructing similarity matrix.\n');
W = zeros(N, N);
for ii=1:N
W(ii, neighborhood(:, ii)) = 1;
W(neighborhood(:, ii), ii) = 1;
end
% STEP 3: COMPUTE EMBEDDING FROM EIGENVECTS OF L
fprintf(1,'-->Computing embedding.\n');
D = diag(sum(W));
L = D-W;
% CALCULATION OF EMBEDDING
options.disp = 0; options.isreal = 1; options.issym = 1;
[Y,eigenvals] = eigs(L,d+1,0,options);
Y = Y(:,2:d+1)'*sqrt(N); % bottom evect is [1,1,1,1...] with eval 0
fprintf(1,'Done.\n');
%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%