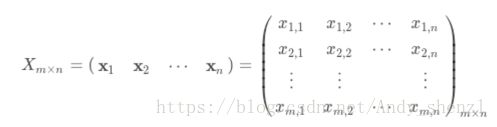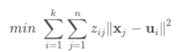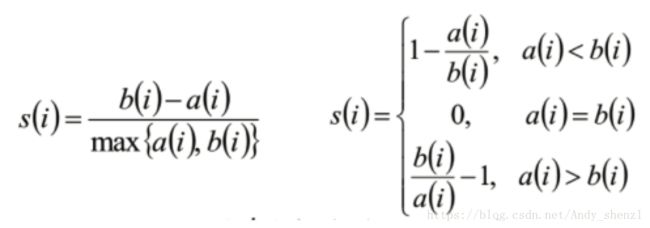K-means算法及python sklearn实现
目录
K-means算法
前言
K-Means算法的概述
K-Means算法的基本原理
K-Means与矩阵分解
实例推演
1、数据准备
2、随机选取重心
3、重新计算重心
4、重复计算
K值的确定
聚类评估:轮廓系数(Silhouette Coefficient )
k-means的优缺点
sklearn中对于kmeans算法的参数
sklearn实例实现
轮廓系数
K-means算法
前言
根据训练样本是否包含标签信息,机器学习可以分为监督学习和无监督学习。聚类算法是典型的无监督学习,其训练样本中只包含样本特征,不包含样本的标签信息。在聚类算法中,利用样本的特征,将具有相似属性的样本划分到同一类别中。
K-means算法,也被称为K-均值或K-平均算法,是一种广泛使用的聚类算法。K-means算法是基于相似性的无监督的算法,通过比较样本之间的相似性,将较为相似的样本划分到同一类别中。由于K-means算法简单、易于实现的特点, K-means算法得到了广泛的应用。
K-Means算法的概述
基本K-Means算法的思想很简单,事先确定常数K,常数K意味着最终的聚类类别数,首先随机选定初始点为质心,并通过计算每一个样本与质心之间的相似度(这里为欧式距离),将样本点归到最相似的类中,接着,重新计算每个类的质心(即为类中心),重复这样的过程,知道质心不再改变,最终就确定了每个样本所属的类别以及每个类的质心。由于每次都要计算所有的样本与每一个质心之间的相似度,故在大规模的数据集上,K-Means算法的收敛速度比较慢。
K-Means算法的基本原理
K-Means算法是较为经典的聚类算法,假设训练数据集X为:{x1,x2,⋯,xn},其中,每一个样本xj为m维的向量。此时的样本为一个m×n的矩阵:
假设有k个类,分别为:{C1,⋯,Ck}。k-Means算法通过欧式距离的度量方法计算每一个样本xj到质心之间的距离,并将其划分到较近的质心所属的类别中并重新计算质心,重复以上的过程,直到质心不再改变为止,上述的过程可以总结为:
- 初始化常数K,随机选取初始点为质心
- 重复计算以下过程,直到质心不再改变
计算样本与每个质心之间的相似度,将样本归类到最相似的类中
重新计算质心
- 输出最终的质心以及每个类
K-Means与矩阵分解
K-Means的目标函数
最终使得质心不再改变,这就意味着每一个样本被划分到了最近的质心所属的类别中,即:
公式推到参见:https://blog.csdn.net/google19890102/article/details/51142299
实例推演
1、数据准备
下面我们用一个案例来看一下:
样本 X0 X1
1 7 5
2 5 7
3 7 7
4 3 3
5 4 6
6 1 4
7 0 0
8 2 2
9 8 7
10 6 8
11 5 5
12 3 7
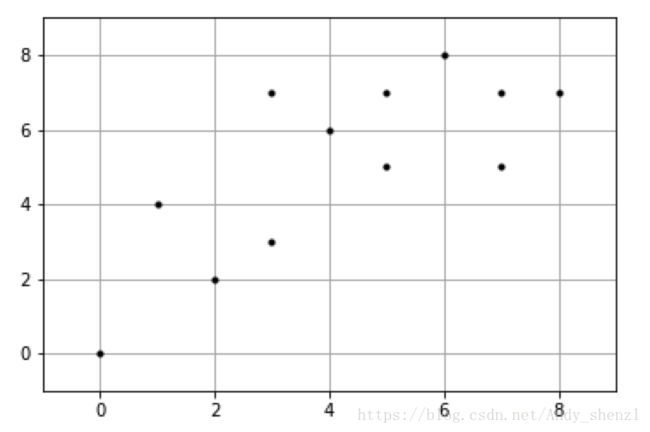
上表有两个解释变量,每个样本有两个特征。用python演示一下
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
X0 = np.array([7, 5, 7, 3, 4, 1, 0, 2, 8, 6, 5, 3])
X1 = np.array([5, 7, 7, 3, 6, 4, 0, 2, 7, 8, 5, 7])
plt.figure()
plt.axis([-1, 9, -1, 9])
plt.grid(True)
plt.plot(X0, X1, 'k.')2、随机选取重心
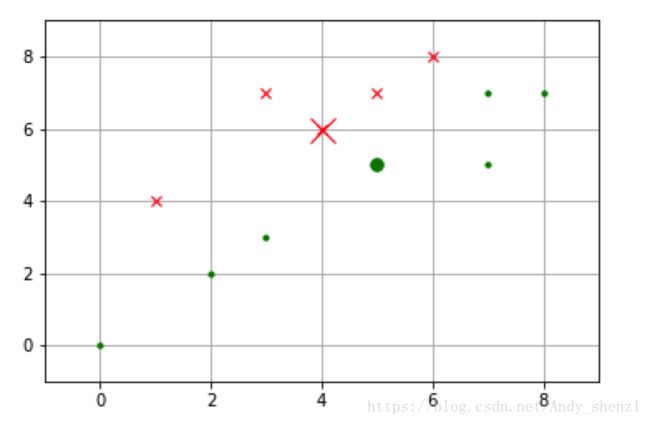
假设K-Means初始化时,随机设定两个重心,将第一个类的重心设置在第5个样本,第二个类的重心设置在第11个样本.那么我们可以把每个实例与两个重心的距离都计算出来,将其分配到最近的类里面。计算结果如下表所示:
| 样本 |
X0 |
X1 |
与C1距离 |
与C2距离 |
上次聚类结果 |
新聚类结果 |
是否改变 |
| 1 |
7 |
5 |
3.16 |
2.00 |
None |
C2 |
YES |
| 2 |
5 |
7 |
1.41 |
2.00 |
None |
C1 |
YES |
| 3 |
7 |
7 |
3.16 |
2.83 |
None |
C2 |
YES |
| 4 |
3 |
3 |
3.16 |
2.83 |
None |
C2 |
YES |
| 5 |
4 |
6 |
0.00 |
1.41 |
None |
C1 |
YES |
| 6 |
1 |
4 |
3.61 |
4.12 |
None |
C1 |
YES |
| 7 |
0 |
0 |
7.21 |
7.07 |
None |
C2 |
YES |
| 8 |
2 |
2 |
4.47 |
4.24 |
None |
C2 |
YES |
| 9 |
8 |
7 |
4.12 |
3.61 |
None |
C2 |
YES |
| 10 |
6 |
8 |
2.83 |
3.16 |
None |
C1 |
YES |
| 11 |
5 |
5 |
1.41 |
0.00 |
None |
C2 |
YES |
| 12 |
3 |
7 |
1.41 |
2.83 |
None |
C1 |
YES |
| C1重心 |
4 |
6 |
|
|
|
|
|
| C2重心 |
5 |
5 |
|
|
|
||
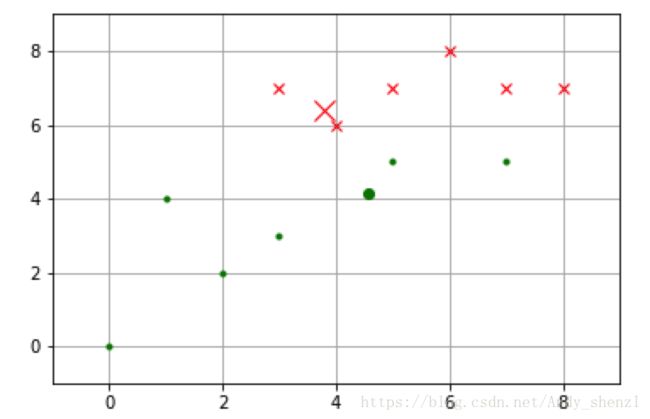
新的重心位置和初始聚类结果如下图所示。第一类用X表示,第二类用点表示。重心位置用稍大的点突出显示。
C1 = [1, 4, 5, 9, 11]#属于C1的index
C2 = list(set(range(12)) - set(C1))#属于C2的index
X0C1, X1C1 = X0[C1], X1[C1]#属于C1的坐标
X0C2, X1C2 = X0[C2], X1[C2]#属于C2的坐标
plt.figure()
plt.axis([-1, 9, -1, 9])
plt.grid(True)
plt.plot(X0C1, X1C1, 'rx')
plt.plot(X0C2, X1C2, 'g.')
plt.plot(4,6,'rx',ms=15.0)
plt.plot(5,5,'g.',ms=15.0)3、重新计算重心
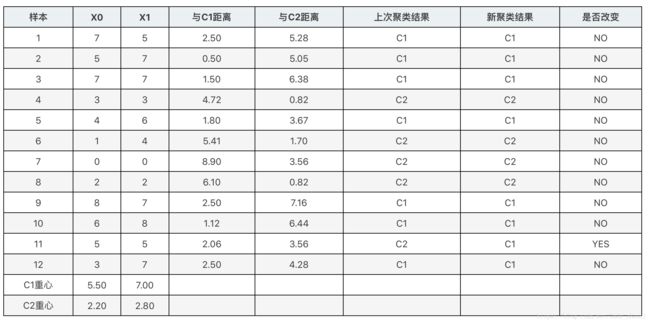
接下来我们需要重新计算两个类的重心,把重心移动到新位置,并重新计算各个样本与新重心的距离,并根据距离远近为样本重新归类。结果如下表所示:
| 样本 |
X0 |
X1 |
与C1距离 |
与C2距离 |
上次聚类结果 |
新聚类结果 |
是否改变 |
| 1 |
7 |
5 |
3.49 |
2.58 |
C2 |
C2 |
NO |
| 2 |
5 |
7 |
1.34 |
2.89 |
C1 |
C1 |
NO |
| 3 |
7 |
7 |
3.26 |
3.75 |
C2 |
C1 |
YES |
| 4 |
3 |
3 |
3.49 |
1.94 |
C2 |
C2 |
NO |
| 5 |
4 |
6 |
0.45 |
1.94 |
C1 |
C1 |
NO |
| 6 |
1 |
4 |
3.69 |
3.57 |
C1 |
C2 |
YES |
| 7 |
0 |
0 |
7.44 |
6.17 |
C2 |
C2 |
NO |
| 8 |
2 |
2 |
4.75 |
3.35 |
C2 |
C2 |
NO |
| 9 |
8 |
7 |
4.24 |
4.46 |
C2 |
C1 |
YES |
| 10 |
6 |
8 |
2.72 |
4.11 |
C1 |
C1 |
NO |
| 11 |
5 |
5 |
1.84 |
0.96 |
C2 |
C2 |
NO |
| 12 |
3 |
7 |
1.00 |
3.26 |
C1 |
C1 |
NO |
| C1重心 |
3.80 |
6.40 |
|
|
|
|
|
| C2重心 |
4.57 |
4.14 |
|||||
C1 = [1, 2, 4, 8, 9, 11]
C2 = list(set(range(12)) - set(C1))
X0C1, X1C1 = X0[C1], X1[C1]
X0C2, X1C2 = X0[C2], X1[C2]
plt.figure()
plt.axis([-1, 9, -1, 9])
plt.grid(True)
plt.plot(X0C1, X1C1, 'rx')
plt.plot(X0C2, X1C2, 'g.')
plt.plot(3.8,6.4,'rx',ms=12.0)
plt.plot(4.57,4.14,'g.',ms=12.0)4、重复计算
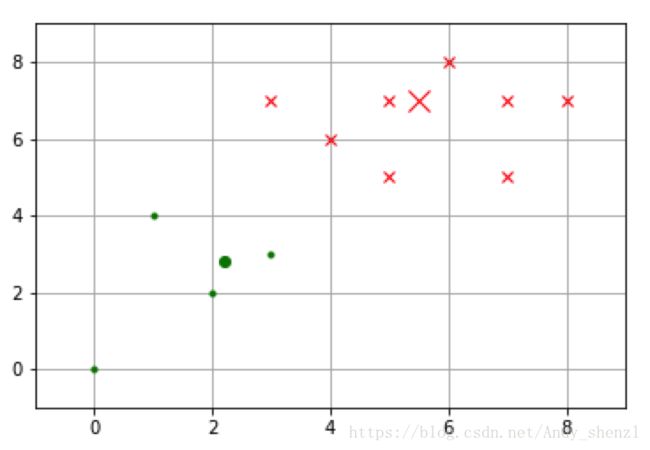
接下来再重复一次上面的做法,把重心移动到新位置,并重新计算各个样本与新重心的距离,并根据距离远近为样本重新归类。结果如下表所示:
C1 = [0, 1, 2, 4, 8, 9, 10, 11]
C2 = list(set(range(12)) - set(C1))
X0C1, X1C1 = X0[C1], X1[C1]
X0C2, X1C2 = X0[C2], X1[C2]
plt.figure()
plt.axis([-1, 9, -1, 9])
plt.grid(True)
plt.plot(X0C1, X1C1, 'rx')
plt.plot(X0C2, X1C2, 'g.')
plt.plot(5.5,7.0,'rx',ms=12.0)
plt.plot(2.2,2.8,'g.',ms=12.0)再重复上面的方法就会发现类的重心不变了,K-Means会在条件满足的时候停止重复聚类过程。通常,条件是前后两次迭代的成本函数值的差达到了限定值,或者是前后两次迭代的重心位置变化达到了限定值。如果这些停止条件足够小,K-Means就能找到最优解。不过这个最优解不一定是全局最优解。
局部最优解
前面介绍过K-Means的初始重心位置是随机选择的。有时,如果运气不好,随机选择的重心会导致K-Means陷入局部最优解。
K-Means最终会得到一个局部最优解,这些类可能没有实际意义,可能是更有合理的聚类结果。为了避免局部最优解,K-Means通常初始时要重复运行十几次甚至上百次。每次重复时,它会随机的从不同的位置开始初始化。最后把最小的成本函数对应的重心位置作为初始化位置。
K值的确定
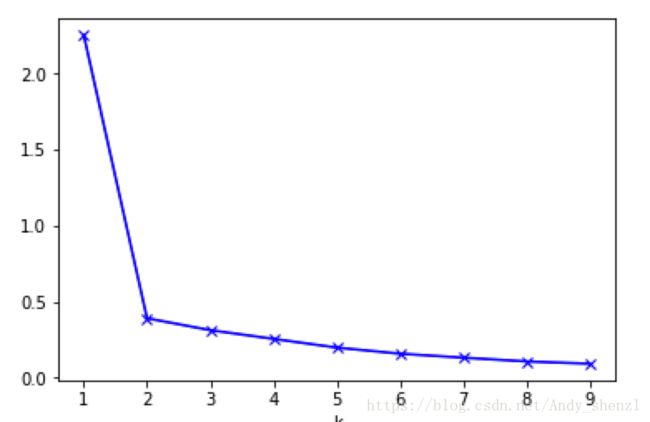
如果问题中没有指定的值,可以通过肘部法则这一技术来估计聚类数量。肘部法则会把不同值的成本函数值画出来。随着值的增大,平均畸变程度会减小;每个类包含的样本数会减少,于是样本离其重心会更近。但是,随着值继续增大,平均畸变程度的改善效果会不断减低。值增大过程中,畸变程度的改善效果下降幅度最大的位置对应的值就是肘部。下面让我们用肘部法则来确定最佳的值。
其中需要解释的一点是:
每个类的畸变程度等于该类重心与其内部成员位置距离的平方和。也即我们前面所说的每个类的类内离差平方和。若类内部的成员彼此间越紧凑则类的畸变程度越小,反之,若类内部的成员彼此间越分散则类的畸变程度越大。
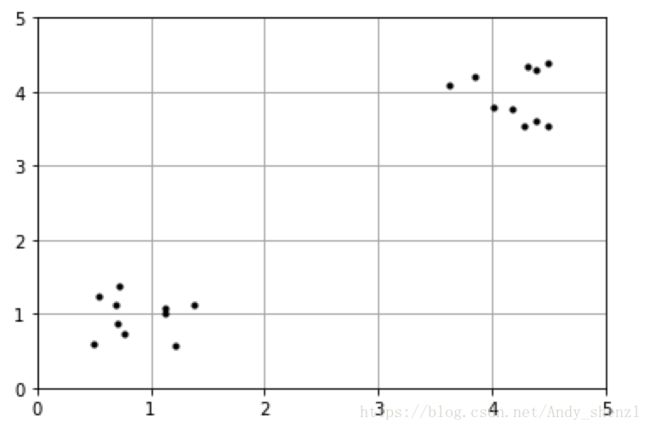
下图数据明显可分成两类:
import numpy as np
cluster1 = np.random.uniform(0.5, 1.5, (2, 10))
cluster2 = np.random.uniform(3.5, 4.5, (2, 10))
X = np.hstack((cluster1, cluster2)).T
plt.figure()
plt.axis([0, 5, 0, 5])
plt.grid(True)
plt.plot(X[:,0],X[:,1],'k.')from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
import matplotlib.pyplot as plt
K = range(1, 10)
meandistortions = []
for k in K:
kmeans = KMeans(n_clusters=k)
kmeans.fit(X)
meandistortions.append(sum(np.min(cdist(X, kmeans.cluster_centers_, 'euclidean'), axis=1)) / X.shape[0])
plt.plot(K, meandistortions, 'bx-')
plt.xlabel('k')
聚类评估:轮廓系数(Silhouette Coefficient )
- 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik}
- si接近1,则说明样本i聚类合理
- si接近-1,则说明样本i更应该分类到另外的簇
- 若si 近似为0,则说明样本i在两个簇的边界上。
import numpy as np
from sklearn.cluster import KMeans
from sklearn import metrics
plt.figure(figsize=(8, 10))
plt.subplot(3, 2, 1)
x1 = np.array([1, 2, 3, 1, 5, 6, 5, 5, 6, 7, 8, 9, 7, 9])
x2 = np.array([1, 3, 2, 2, 8, 6, 7, 6, 7, 1, 2, 1, 1, 3])
X = np.array(list(zip(x1, x2))).reshape(len(x1), 2)
plt.xlim([0, 10])
plt.ylim([0, 10])
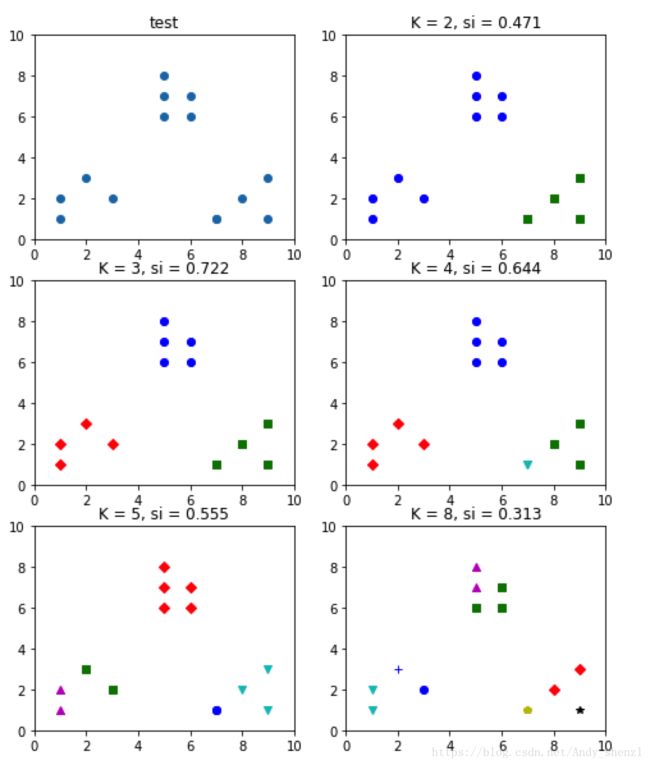
plt.title('test')
plt.scatter(x1, x2)
colors = ['b', 'g', 'r', 'c', 'm', 'y', 'k', 'b']
markers = ['o', 's', 'D', 'v', '^', 'p', '*', '+']
tests = [2, 3, 4, 5, 8]
subplot_counter = 1
for t in tests:
subplot_counter += 1
plt.subplot(3, 2, subplot_counter)
kmeans_model = KMeans(n_clusters=t).fit(X)
for i, l in enumerate(kmeans_model.labels_):
plt.plot(x1[i], x2[i], color=colors[l], marker=markers[l],ls='None')
plt.xlim([0, 10])
plt.ylim([0, 10])
plt.title('K = %s, si = %.03f' % (t, metrics.silhouette_score(X, kmeans_model.labels_,metric='euclidean')))
很显然,这个数据集包括三个类。在k=3的时候轮廓系数是最大的。在k=8的时候,每个类的样本不仅彼此很接近,而且与其他类的样本也非常接近,因此这时轮廓系数是最小的。
k-means的优缺点
优点:
1、理解容易,聚类效果好;
2、处理大数据时,该算法可以保证较好的伸缩性和高效性;
3、当簇近似高斯分布时,效果很好
缺点:
1、K值需要自己设定,不同K值结果不同;
2、对于初试设定的重心非常敏感;
3、不适合发现非凸形状的簇或者大小差别较大的簇
4、特殊值(离群值)对模型的影响较大
https://www.naftaliharris.com/blog/visualizing-k-means-clustering/
这个网址可以展现聚类可视化的的过程,大家可以参考。
本次K-means主要是对表十大数据挖掘算法,所以不展开讲,之后会对K-means的延伸算法包括K-means++、MiniBatchKMeans、K-Mediods等进行讲解
sklearn中对于kmeans算法的参数
参数:
n_clusters:整形,缺省值=8 【生成的聚类数,即产生的质心(centroids)数。】
max_iter:整形,缺省值=300
执行一次k-means算法所进行的最大迭代数。
n_init:整形,缺省值=10
用不同的质心初始化值运行算法的次数,最终解是在inertia意义下选出的最优结果。
init:有三个可选值:’k-means++’, ‘random’,或者传递一个ndarray向量。
此参数指定初始化方法,默认值为 ‘k-means++’。
(1)‘k-means++’ 用一种特殊的方法选定初始质心从而能加速迭代过程的收敛
(2)‘random’ 随机从训练数据中选取初始质心。
(3)如果传递的是一个ndarray,则应该形如 (n_clusters, n_features) 并给出初始质心。
precompute_distances:三个可选值,‘auto’,True 或者 False。
预计算距离,计算速度更快但占用更多内存。
(1)‘auto’:如果 样本数乘以聚类数大于 12million 的话则不预计算距离。This corresponds to about 100MB overhead per job using double precision.
(2)True:总是预先计算距离。
(3)False:永远不预先计算距离。
tol:float形,默认值= 1e-4 与inertia结合来确定收敛条件。
n_jobs:整形数。 指定计算所用的进程数。内部原理是同时进行n_init指定次数的计算。
(1)若值为 -1,则用所有的CPU进行运算。若值为1,则不进行并行运算,这样的话方便调试。
(2)若值小于-1,则用到的CPU数为(n_cpus + 1 + n_jobs)。因此如果 n_jobs值为-2,则用到的CPU数为总CPU数减1。
random_state:整形或 numpy.RandomState 类型,可选
用于初始化质心的生成器(generator)。如果值为一个整数,则确定一个seed。此参数默认值为numpy的随机数生成器。
copy_x:布尔型,默认值=True
当我们precomputing distances时,将数据中心化会得到更准确的结果。如果把此参数值设为True,则原始数据不会被改变。如果是False,则会直接在原始数据
上做修改并在函数返回值时将其还原。但是在计算过程中由于有对数据均值的加减运算,所以数据返回后,原始数据和计算前可能会有细小差别。
属性:
cluster_centers_:向量,[n_clusters, n_features] (聚类中心的坐标)
Labels_: 每个点的分类
inertia_:float形
每个点到其簇的质心的距离之和。
Notes:
这个k-means运用了 Lioyd’s 算法,平均计算复杂度是 O(k*n*T),其中n是样本量,T是迭代次数。
计算复杂读在最坏的情况下为 O(n^(k+2/p)),其中n是样本量,p是特征个数。(D. Arthur and S. Vassilvitskii, ‘How slow is the k-means method?’ SoCG2006)
在实践中,k-means算法时非常快的,属于可实践的算法中最快的那一类。但是它的解只是由特定初始值所产生的局部解。所以为了让结果更准确真实,在实践中要用不同的初始值重复几次才可以。
Methods:
fit(X[,y]):
计算k-means聚类。
fit_predictt(X[,y]):
计算簇质心并给每个样本预测类别。
fit_transform(X[,y]):
计算簇并 transform X to cluster-distance space。
get_params([deep]):
取得估计器的参数。
predict(X):predict(X)
给每个样本估计最接近的簇。
score(X[,y]):
计算聚类误差
set_params(**params):
为这个估计器手动设定参数。
transform(X[,y]): 将X转换为群集距离空间。
在新空间中,每个维度都是到集群中心的距离。 请注意,即使X是稀疏的,转换返回的数组通常也是密集的。
sklearn实例实现
我们还是基于上面推倒的数据案例:
import numpy as np
X0 = np.array([7, 5, 7, 3, 4, 1, 0, 2, 8, 6, 5, 3])
X1 = np.array([5, 7, 7, 3, 6, 4, 0, 2, 7, 8, 5, 7])
X = np.array(list(zip(X0, X1))).reshape(len(X0), 2)#压缩数据整合在一起
from sklearn.cluster import KMeans#导入KMeans包
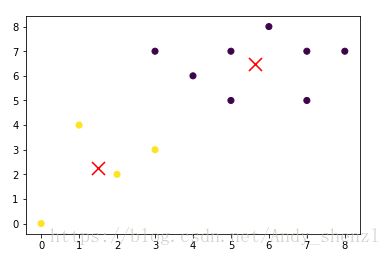
km = KMeans(n_clusters=2).fit(X)#按照k=2进行计算接下来我们看下结果:
km.labels_
out:array([0, 0, 0, 1, 0, 1, 1, 1, 0, 0, 0, 0], dtype=int32)
centroids = km.cluster_centers_
print(centroids)
out:
[[5.625 6.5 ]
[1.5 2.25 ]]import matplotlib.pyplot as plt
plt.figure()
plt.scatter(X[:, 0], X[:, 1], c=km.labels_)#原始数据散点图,按照分类查看
centroids = km.cluster_centers_
plt.scatter(centroids[:, 0], centroids[:, 1],
marker='x', s=169, linewidths=3,
color='r', zorder=10)#重心红色X进行突出轮廓系数
from sklearn.cluster import KMeans
from sklearn import metrics
scores = []
for k in range(2,12):
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
scoresout: [0.5438942330933946, 0.3842109168420305, 0.355936245851519, 0.32130908377531114, 0.29601716489116875, 0.2552005855998481, 0.19930860195228847, 0.15131817710529702, 0.07047330194279122, 0.07047330194279122]