医疗图像切割FCN的Keras实现
最近看了几篇关于视网膜层切割的处理论文,现在比较流行的方法是用FCN(全卷积神经网络来做)。在医疗领域中,通常使用一种称之为U-net的FCN来做图像切割,效果不错。
本文基于U-net来做实现,详细介绍了如何搭建一个U-net神经网络。
全卷积神经网络
关于FCN的介绍详见我的FCN全卷积网络的博客,这里不再赘述啦。
医学图像分割框架
现在的医学图像分割(尤其是眼科OCT图像(光学相关层析图像))主要有两种框架,一个是基于CNN加上图搜索等算法的,另一个就是基于FCN的U-net。这里我们主要说后者,关于两者的区别也在前面提到的博文中有介绍。
U-net(FCN)
在医学图像处理领域,有一个应用很广泛的网络结构—U-net ,网络结构如下(这个图是德国弗莱堡大学关于FCN的介绍):
U-net architecture (example for 32x32 pixels in the lowest resolution). Each blue box corresponds to a multi-channel feature map. The number of channels is denoted on top of the box. The x-y-size is provided at the lower left edge of the box. White boxes represent copied feature maps. The arrows denote the different operations.
这段话简单翻译过来就是:“U-net架构中的每个蓝色box都对应一个多通道特征图(multi-channel feature map)。通道的个数在蓝色box的上面。长和宽在蓝色box的左边。白色的box表示两层的merge。箭头表示不同的操作(比如卷积,池化等等)。”
可以看出来,一个全卷积神经网络,输入和输出都是图像,没有全连接层。较浅的高分辨率层用来解决像素定位的问题,较深的层用来解决像素分类的问题。
问题分析
参考博客全卷机神经网络图像分割(U-net)-keras实现
采用的数据集是一个isbi挑战的数据集,网址为: http://brainiac2.mit.edu/isbi_challenge/
数据集需要注册下载,图片格式为tif。需要用工具将其中的多个图片拆分出来。Windows下我用的是软件TiffToy,如果非Windows可以用Github的split_merge_tif.py函数来做tif文件的切割。
这个挑战就是提取出细胞边缘,属于一个二分类问题,问题不算难,可以当做一个练手。
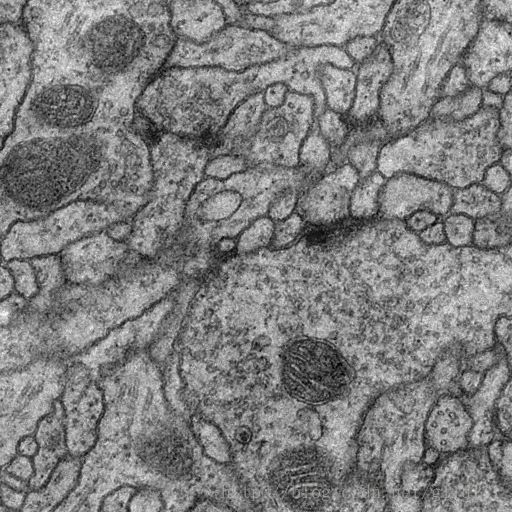
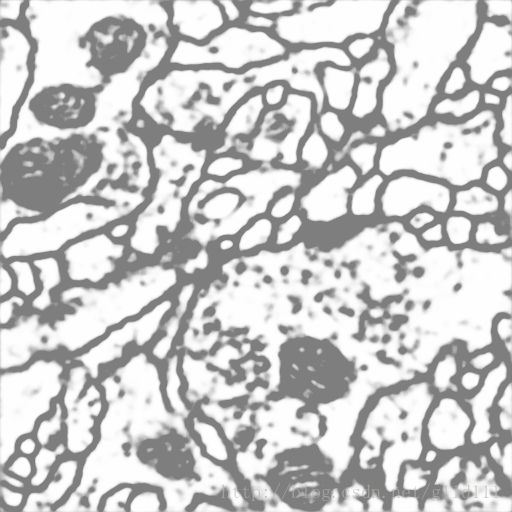
① 图片详情

上图是样本,下图为该图的训练结果,或者说是对样本边界分割后的结果。

② 训练集特点
这里最大的挑战就是数据集很小,只有30张512*512的训练图像。我们这里直接用这个数据集做训练。
③ U-net实现
下面是U-net的Keras架构实现,具体代码参见zhixuhao的开源项目。我额外采用了别的模型再跑了一下,效果也不错,大家可以试试不同的U-net模型。此外,还写了一个存成图片的脚本,提交给pull主了。
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(inputs)
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv1)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(pool1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(pool2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv3)
pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(pool3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv4)
# drop4 = Dropout(0.5)(conv4)
pool4 = MaxPooling2D(pool_size=(2, 2))(conv4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(pool4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv5)
#drop5 = Dropout(0.5)(conv5)
up6 = Conv2D(512, 2, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(UpSampling2D(size = (2,2))(conv5))
merge6 = merge([conv4,up6], mode = 'concat', concat_axis = 3)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(merge6)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv6)
up7 = Conv2D(256, 2, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(UpSampling2D(size = (2,2))(conv6))
merge7 = merge([conv3,up7], mode = 'concat', concat_axis = 3)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(merge7)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'glorot_normal')(conv7)
up8 = Conv2D(128, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv7))
merge8 = merge([conv2,up8], mode = 'concat', concat_axis = 3)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge8)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv8)
up9 = Conv2D(64, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv8))
merge9 = merge([conv1,up9], mode = 'concat', concat_axis = 3)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge9)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
conv9 = Conv2D(2, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
conv10 = Conv2D(1, 1, activation = 'sigmoid')(conv9)
model = Model(input = inputs, output = conv10)
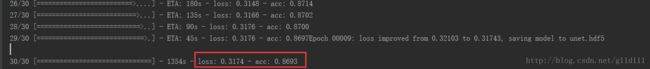
model.compile(optimizer = Adam(lr = 1e-4), loss = 'binary_crossentropy', metrics = ['accuracy'])④效果
在没有使用图像增强的情况下,我的准确率可以达到87%。(ps:墙裂建议大家用GPU训练!在CPU下,30张图片10个epoch的训练,每个epoch都得20多分钟···)
致谢
感谢zhixuhao的博客和开源代码。我主要是在其内容基础上做的模型和参数调整,效果是很好的。
关于图像增强的问题我不是很了解,有兴趣的同学可以移步到他的博客。