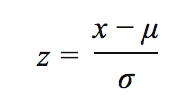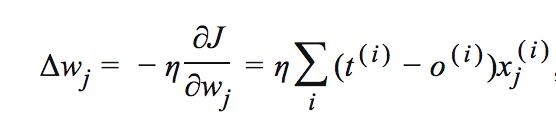浅析数据标准化和归一化,优化机器学习算法输出结果
作者:chen_h
微信号 & QQ:862251340
微信公众号:coderpai
关于标准化(standardization)
数据标准化能将原来的数据进行重新调整(一般也称为 z-score 规范化方法),以便他们具有标准正态分布的属性,即 μ=0 和 σ=1。其中,μ 表示平均值,σ 表示标准方差。数据标准化之后的形式可以按照如下公式进行计算:
如果我们是比较两个不同大小维度的数据,那么将这些数据标准化到以 0 为中心并且标准差为 1 的范围,这对许多的机器学习算法来说也是一般的要求。比如,从直觉上来说,我们可以将梯度下降看作一个特殊的例子。由于特征值 xj 在权重更新中发挥作用,那么某些权重可能比其他权重更新的速度更快,即:
其中,wj:=wj+Δwj,η 表示学习率,t 表示目标正确分类结果,o 表示模型的输出分类结果。
其他直观的例子包括 KNN 算法和一些聚类算法上面,都会使用这种数据标准化的方法。
事实上,我能想到唯一不需要数据标准化的算法就应该是基于决策树的算法了。我们来看一般的 CART 决策树算法。在这里我们不深入的分析信息熵的含义,我们可以把这个决策简单的想象成 is feature x_i >= some_val ?。从直观上来讲,我们真的不需要来关心数据特征在哪个大小维度(比如,不同数量级,不同领域 —— 这些真的不关心)。
那么,在哪些算法中特征数据标准化是比较重要的呢?比如下面这些算法:
- 对于基于欧几里得距离的 KNN 算法,如果你想要所有的数据对算法都有贡献,那么必须进行标准化;
- k-means 算法;
- 逻辑回归,支持向量机,感知器,神经网络等,如果你正是在使用梯度下降(上升)来作为优化器,那么采用标准化会让权重更快的收敛;
- 线性判别分析,PCA,核方法;
另外,我们还要考虑我们的数据是需要进行“标准化(standardize)”还是“归一化(normalize)”(这里是缩放到 [0, 1] 范围)。因为有些算法假设我们的数据是以 0 为中心分布的,那么这时候进行标准化还是归一化就需要自己思考了。例如,如果我们对一个小型多层感知器(利用 tanh 激活函数)进行权重初始化,权重应该是 0 ,或者是以零为中心的小随机数,这样能更好的更新模型权重。作为一个经验法则,我想说的是:如果你不确定对数据进行标准化还是归一化,那么你就对数据进行标准化吧,至少它不会对数据和结果造成伤害。
标准化(Standardization)
数据的标准化是将数据按比例缩放,使之落入一个小的特定区间,标准化之后的数据可正可负,但是一般绝对值不会太大,一般是 z-score 规范化方法。
归一化(Normalization)
主要是为了数据处理提出来的,把数据映射到 0~1 范围之间处理,更加便捷快速,这应该归到数字信号处理范畴之内。一般方法是最小-最大缩放方法。
关于最小-最大缩放处理
还有一种数据处理的方法是最小 - 最大缩放。在这种方法中,数据被缩放到一个固定的范围 —— 通常是 0 到 1。与标准化相比,有限范围的损失值计算最终将得到较小的标准偏差,这也可以抑制一些异常值的影响。
最小 - 最大缩放处理可以通过以下公式完成:
选择 Z-score 标准化还是最小 - 最大缩放?
具体选择哪一个数据处理方法没有很明确的答案,它主要取决于具体的应用程序。
例如,在聚类分析中,为了比较基于特定距离度量的特征数据之间的相似性,数据标准化可能是一个特别重要的方式。另一个比较突出的例子就是主成分分析,我们通常采用标准化来做数据进行缩放。因为我们对最大化方差的方向感兴趣。
如何使用 scikit-learn 来实现标准化和归一化
当然,我们可以利用 Numpy 包来计算数据的 z-score,并使用前面提到的公式来进行标准化。但是,如果我们使用 Python 的开源机器学习库 scikit-learn 中的预处理模块来做,会更加便捷。
为了下面更好的讨论,我们采用 UCI 机器学习库中的 “Wine” 数据集来进行代码编写。
import pandas as pd
import numpy as np
df = pd.io.parsers.read_csv(
'https://raw.githubusercontent.com/rasbt/pattern_classification/master/data/wine_data.csv',
header=None,
usecols=[0,1,2]
)
df.columns=['Class label', 'Alcohol', 'Malic acid']
df.head()| Class label | Alcohol | Malic acid |
|---|---|---|
| 0 | 1 | 14.23 |
| 1 | 1 | 13.20 |
| 2 | 1 | 13.16 |
| 3 | 1 | 14.37 |
| 4 | 1 | 13.24 |
正如我们在上面的表格中所看到的,Alcohol 特征(百分比 / 体积)和 Malic acid(g/l)是在不同大小维度上面进行描述的,所有在比较或者组合这些特征之前,进行数据缩放是非常有必要的。
标准化和最小-最大缩放
from sklearn import preprocessing
std_scale = preprocessing.StandardScaler().fit(df[['Alcohol', 'Malic acid']])
df_std = std_scale.transform(df[['Alcohol', 'Malic acid']])
minmax_scale = preprocessing.MinMaxScaler().fit(df[['Alcohol', 'Malic acid']])
df_minmax = minmax_scale.transform(df[['Alcohol', 'Malic acid']])
print('Mean after standardization:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_std[:,0].mean(), df_std[:,1].mean()))
print('\nStandard deviation after standardization:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_std[:,0].std(), df_std[:,1].std()))Mean after standardization:
Alcohol=0.00, Malic acid=0.00
Standard deviation after standardization:
Alcohol=1.00, Malic acid=1.00
print('Min-value after min-max scaling:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_minmax[:,0].min(), df_minmax[:,1].min()))
print('\nMax-value after min-max scaling:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_minmax[:,0].max(), df_minmax[:,1].max()))Min-value after min-max scaling:
Alcohol=0.00, Malic acid=0.00
Max-value after min-max scaling:
Alcohol=1.00, Malic acid=1.00
画图
%matplotlib inline
from matplotlib import pyplot as plt
def plot():
plt.figure(figsize=(8,6))
plt.scatter(df['Alcohol'], df['Malic acid'],
color='green', label='input scale', alpha=0.5)
plt.scatter(df_std[:,0], df_std[:,1], color='red',
label='Standardized ', alpha=0.3)
plt.scatter(df_minmax[:,0], df_minmax[:,1],
color='blue', label='min-max scaled [min=0, max=1]', alpha=0.3)
plt.title('Alcohol and Malic Acid content of the wine dataset')
plt.xlabel('Alcohol')
plt.ylabel('Malic Acid')
plt.legend(loc='upper left')
plt.grid()
plt.tight_layout()
plot()
plt.show()上面的图包括所有三个不同比例的葡萄酒数据点:原始酒精含量数据(绿色),标准化之后的数据(红色)和归一化之后的数据(蓝色)。在下面的图中,我们将放大三个不同的坐标轴。
fig, ax = plt.subplots(3, figsize=(6,14))
for a,d,l in zip(range(len(ax)),
(df[['Alcohol', 'Malic acid']].values, df_std, df_minmax),
('Input scale',
'Standardized',
'min-max scaled [min=0, max=1]')
):
for i,c in zip(range(1,4), ('red', 'blue', 'green')):
ax[a].scatter(d[df['Class label'].values == i, 0],
d[df['Class label'].values == i, 1],
alpha=0.5,
color=c,
label='Class %s' %i
)
ax[a].set_title(l)
ax[a].set_xlabel('Alcohol')
ax[a].set_ylabel('Malic Acid')
ax[a].legend(loc='upper left')
ax[a].grid()
plt.tight_layout()
plt.show()自己动手,丰衣足食
当然,我们也可以手动编写标准化方程和最小-最大缩放。但是,实际正真项目中,还是比较推荐使用 scikit-learn 。比如:
std_scale = preprocessing.StandardScaler().fit(X_train)
X_train = std_scale.transform(X_train)
X_test = std_scale.transform(X_test)接下来,我们采用纯 Python 代码来实现这几个指标,并且也会用 numpy 来进行计算加速。回想一下我们用的几个参数指标:
纯 python
# Standardization
x = [1,4,5,6,6,2,3]
mean = sum(x)/len(x)
std_dev = (1/len(x) * sum([ (x_i - mean)**2 for x_i in x]))**0.5
z_scores = [(x_i - mean)/std_dev for x_i in x]
# Min-Max scaling
minmax = [(x_i - min(x)) / (max(x) - min(x)) for x_i in x]Numpy
import numpy as np
# Standardization
x_np = np.asarray(x)
z_scores_np = (x_np - x_np.mean()) / x_np.std()
# Min-Max scaling
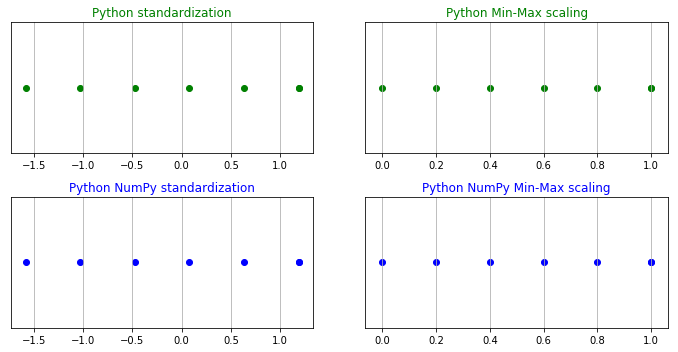
np_minmax = (x_np - x_np.min()) / (x_np.max() - x_np.min())可视化
为了检验我们的代码是否正常工作,我们通过可视化来进行查看。
from matplotlib import pyplot as plt
fig, ((ax1, ax2), (ax3, ax4)) = plt.subplots(nrows=2, ncols=2, figsize=(10,5))
y_pos = [0 for i in range(len(x))]
ax1.scatter(z_scores, y_pos, color='g')
ax1.set_title('Python standardization', color='g')
ax2.scatter(minmax, y_pos, color='g')
ax2.set_title('Python Min-Max scaling', color='g')
ax3.scatter(z_scores_np, y_pos, color='b')
ax3.set_title('Python NumPy standardization', color='b')
ax4.scatter(np_minmax, y_pos, color='b')
ax4.set_title('Python NumPy Min-Max scaling', color='b')
plt.tight_layout()
for ax in (ax1, ax2, ax3, ax4):
ax.get_yaxis().set_visible(False)
ax.grid()
plt.show()实战:PCA 中是否进行数据标准化对分类任务的影响
在文章的前面,我们提到了在 PCA 中对数据进行标准化是至关重要的,因为它是分析不同特征之间的差异。现在,让我们看看标准化是如何影响 PCA 对整个葡萄酒数据分类结果产生的影响。
接下来,我们主要通过这些步骤进行描述:
- 读取数据集;
- 将原始数据集拆分为训练集和测试集;
- 特征数据标准化;
- PCA 降维;
- 训练朴素贝叶斯分类器;
- 利用标准化数据和不利用标准化数据分别对分类器进行评估;
读取数据集
import pandas as pd
df = pd.io.parsers.read_csv(
'https://raw.githubusercontent.com/rasbt/pattern_classification/master/data/wine_data.csv',
header=None,
)将原始数据集拆分为训练集和测试集
在这一步中,我们将数据随机的分为一个训练集和一个测试集,其中训练集包含整个数据集的 70%,测试集包含整个数据集的 30%。
from sklearn.cross_validation import train_test_split
X_wine = df.values[:,1:]
y_wine = df.values[:,0]
X_train, X_test, y_train, y_test = train_test_split(X_wine, y_wine,
test_size=0.30, random_state=12345)特征数据标准化
from sklearn import preprocessing
std_scale = preprocessing.StandardScaler().fit(X_train)
X_train_std = std_scale.transform(X_train)
X_test_std = std_scale.transform(X_test)PCA 降维
现在,我们对标准化的数据和非标准化的数据分别进行 PCA 操作,将数据集转化为二维特征子空间。在一个真实的应用程序中,我们还会有一个交叉验证的过程,以便找出一些过度拟合的信息。但是,在这里我们就不做这个过程了,因为我们不是要设计一个完美的分类器,我们在这里只是想要去比较标准化对分类结果的影响。
from sklearn.decomposition import PCA
# on non-standardized data
pca = PCA(n_components=2).fit(X_train)
X_train = pca.transform(X_train)
X_test = pca.transform(X_test)
# om standardized data
pca_std = PCA(n_components=2).fit(X_train_std)
X_train_std = pca_std.transform(X_train_std)
X_test_std = pca_std.transform(X_test_std)让我们快速的查看一下我们的新特征。如下图:
from matplotlib import pyplot as plt
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(10,4))
for l,c,m in zip(range(1,4), ('blue', 'red', 'green'), ('^', 's', 'o')):
ax1.scatter(X_train[y_train==l, 0], X_train[y_train==l, 1],
color=c,
label='class %s' %l,
alpha=0.5,
marker=m
)
for l,c,m in zip(range(1,4), ('blue', 'red', 'green'), ('^', 's', 'o')):
ax2.scatter(X_train_std[y_train==l, 0], X_train_std[y_train==l, 1],
color=c,
label='class %s' %l,
alpha=0.5,
marker=m
)
ax1.set_title('Transformed NON-standardized training dataset after PCA')
ax2.set_title('Transformed standardized training dataset after PCA')
for ax in (ax1, ax2):
ax.set_xlabel('1st principal component')
ax.set_ylabel('2nd principal component')
ax.legend(loc='upper right')
ax.grid()
plt.tight_layout()
plt.show() 训练朴素贝叶斯分类器
接下来,我们使用一个朴素贝叶斯分类器来进行分类任务。也就是说,我们假设每一个特征都是独立分布的。总而言之,这是一个简单的分类器,但是具有很好的鲁棒性。
贝叶斯规则:
其中:
- ω:表示分类标签;
- P(ω | x):表示后验概率;
- P(x | ω):表示先验概率;
判别规则如下:
我不想在本文介绍很多的贝叶斯内容,如果你对这方面感兴趣,可以自己上网看看材料,网上有很多这方面的资料。
from sklearn.naive_bayes import GaussianNB
# on non-standardized data
gnb = GaussianNB()
fit = gnb.fit(X_train, y_train)
# on standardized data
gnb_std = GaussianNB()
fit_std = gnb_std.fit(X_train_std, y_train)利用标准化数据和不利用标准化数据分别对分类器进行评估
from sklearn import metrics
pred_train = gnb.predict(X_train)
print('\nPrediction accuracy for the training dataset')
print('{:.2%}'.format(metrics.accuracy_score(y_train, pred_train)))
pred_test = gnb.predict(X_test)
print('\nPrediction accuracy for the test dataset')
print('{:.2%}\n'.format(metrics.accuracy_score(y_test, pred_test)))Prediction accuracy for the training dataset
81.45%
Prediction accuracy for the test dataset
64.81%
pred_train_std = gnb_std.predict(X_train_std)
print('\nPrediction accuracy for the training dataset')
print('{:.2%}'.format(metrics.accuracy_score(y_train, pred_train_std)))
pred_test_std = gnb_std.predict(X_test_std)
print('\nPrediction accuracy for the test dataset')
print('{:.2%}\n'.format(metrics.accuracy_score(y_test, pred_test_std)))Prediction accuracy for the training dataset
96.77%
Prediction accuracy for the test dataset
98.15%
正如我们所看到的,在 PCA 之前进行标准化,确实对模型的正确率增加了不少。
来源:sebastianraschka