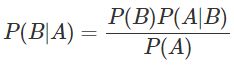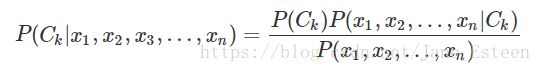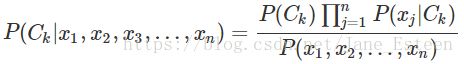朴素贝叶斯学习笔记
1.1 贝叶斯公式
P(B|A): 事件A发生条件下事件B发生的概率,即后验概率;
P(B): 事件B发生的概率,即先验概率;
P(A|B): 事件B发生条件下事件A发生的概率,即条件概率。
1.2 特征条件独立假设
要求后验概率,麻烦的点在于条件概率的参数太多,有着指数级数量的参数,假设:
![]()
![]()
(上头的参数个数是《统计学习方法》第47页给出的 ,我不太理解,举个例子,如果有两个特征X1,X2,各有3种取值,标签为Y,有两个取值,按李航老师的说法,要求的参数就是2*3*3 = 18种。可是
P(X1,X2 | Y) = P(X1 | X2,Y) * P(X2 | Y), 分解后,第一部分的参数应该是3 * 3 * 2,第二部分的参数是3 * 2,加起来要求的参数就是18 + 6 = 24种。你可能会想不要分解啊,P(X1,X2 | Y) 里头就有3 * 3 * 2 = 18种组合,那就是18个参数,可是不分解你咋求?问题未完,等我整理完条件概率独立再接着说。。。)
所以朴素贝叶斯要对条件概率分布作条件独立性的假设,这样一来后验概率求解公式就是:
(续上头的例子,这里说参数规模降到2 * (3 + 3) = 12种,这很合理啊,因为条件概率独立的时候,P(X1,X2 | Y) = P(X1 | Y) * P(X2 | Y),这里要求的参数的确是3 * 2 + 3 * 2 = 12种。所以说要分解之后你才会知道究竟要算多少个参数。假设要写个程序,给定一个数据集,随便给特征,就要算出归为哪一类,那么通常做法就是先定义一个函数,把数据集所有要求的参数求出来,在定义一个计算的函数,用来算后验概率,然后给啥特征,查啥特征相应的条件概率,先验概率,再用定义的计算函数算出后验概率。条件概率分布独立之后参数个数没有任何疑惑,可是没有独立的个数真只有3 * 3 * 2种?有没有人理解了我的困惑,并且不吝爱心回答下我~)
补充一句,我们的目标就是后验概率最大化,用以确定给定一系列特征究竟该归为哪一类。
1.3 极大似然估计
我真诚的希望我写的博客能兼具实用与美观,可是我又在欺骗我自己了。
以下公式暂时似懂非懂就可以了,因为自己动手算过才会理解公式之美。
1.4 贝叶斯估计
贝叶斯估计啊就是在极大似然估计的基础上加一个laplace平滑,这样做的原因是,如果你输入了一个特征值,可是在训练集里,给定标签是找不到对应的这个特征值的,那么这个特征值的条件概率肯定为0,后果就是条件概率的连乘就会变成0,紧接着后验概率也变成0,其他特征的条件概率大概会觉得很委屈。
所以呢贝叶斯估计就考虑到了这点,条件概率公式分子分母都加一个平滑项,这样分子为0不会造成条件概率为0,分子和分母一样的时候,分子加1就会概率大于1,分母也加一个平滑项就能解决这个问题了。看公式(4.10)
1.5 动手实践(R)
# 多项式模型
a = data.frame(x1 = c(rep(1:3,each = 5)),x2 = c("S",rep("M",2),rep("S",3),rep("M",2),rep("L",3),rep("M",2),rep("L",2)),
y = c(-1,-1,1,1,-1,-1,-1,rep(1,7),-1)) # 数据
a$y <- factor(a$y) # 把类别变量变成因子型变量
laplace <- 1 # 定义平滑值
vars = names(a) # 所有变量名
vars = vars[-length(vars)] # 剔除因变量后的变量
prior <- table(a$y,dnn = c("y")) # 求先验概率的准备工作
prior_prob <- (prior + laplace)/(sum(prior) + laplace * nlevels(a$y)) # 先验概率
tables <- sapply(vars,function(x){
tab = table(a$y,a[[x]],dnn = c("",x))
t((tab + laplace)/(rowSums(tab) + laplace * ncol(tab)))
},simplify = FALSE) # 条件概率
# 给定x = (2,S)计算:
p1 <- prior_prob[[2]] * tables$x1["2","1"] * tables$x2["S","1"] # P(y = 1) * P(x1 = 1|y = 1) * P(x2 = "S"|y = 1)
p2 <- prior_prob[[1]] * tables$x1["2","-1"] * tables$x2["S","-1"] # P(y = -1) * P(x1 = 1|y = -1) * P(x2 = "S"|y = -1)上面的代码是针对特征是离散变量来的,如果特征是连续的呢,这个时候就可以假装特征变量都是服从正太分布,算出均值标准差,不就可以很轻易的随意赋值,再算出相应的概率密度值了吗,概率密度值虽然不是概率,可是还是能说明这个特征值出现的相对概率。
# 高斯模型
b = data.frame("height" = c(6,5.92,5.58,5.92,5,5.5,5.42,5.75),
"weight" = c(180,190,170,165,100,150,130,150),
"foot" = c(12,11,12,10,6,8,7,9),
"gender" = c(rep("male",4),rep("female",4)))
b$gender <- factor(b$gender)
vars = names(b) # 所有变量名
vars = vars[-length(vars)] # 剔除因变量后的变量
prior <- table(b$gender,dnn = c("gender")) # 求先验概率的准备工作
prior_prob <- (prior + laplace)/(sum(prior) + laplace * nlevels(b$gender)) # 先验概率
tables <- sapply(vars,function(x){
tab <- rbind(tapply(b[[x]],b$gender,mean),
tapply(b[[x]],b$gender,stats::sd))
rownames(tab) <- c("mean", "sd")
as.table(tab)
},simplify = FALSE) # 条件概率
# 给定x = (6,130,8)计算:
p1 <- prior_prob[[2]] * dnorm(6,tables$height["mean","male"], tables$height["sd","male"])*
dnorm(130,tables$weight["mean","male"], tables$weight["sd","male"])*
dnorm(8,tables$foot["mean","male"], tables$foot["sd","male"])
p2 <- prior_prob[[1]] * dnorm(6,tables$height["mean","female"], tables$height["sd","female"])*
dnorm(130,tables$weight["mean","female"], tables$weight["sd","female"])*
dnorm(8,tables$foot["mean","female"], tables$foot["sd","female"])
p2/p1 > 1000还有一种模型叫做伯努利模型,这种和多项式模型很像,只不过特征变量必须非零即1,所以要对特征二值化,其他都一样啦。
最后,理解这个算法怎么实现之后,真要用的时候就可以调包啦。
library(e1071)
a$x1 <- as.character(a$x1); a$x2 <- as.character(a$x2) ; a$y <- as.factor(a$y) # 如果特征不是数值变量,这个包就会算概率
a2 = naiveBayes(y ~., data = a,laplace = 1)
(a3 = predict(a2,data.frame(x1 = 2,x2= "S"),type = "class"))
b$gender <- as.factor(b$gender) # 如果特征是数值变量,就默认数值变量服从高斯分布,用概率密度值来代替概率
b2 = naiveBayes(gender ~., data = b)
(b3 = predict(b2,data.frame(height = 6,weight= 130,foot = 8),type = "class"))还没完,我还想补充下这个算法的优缺点:
缺点:假设过强,分类性能不一定高。
优点:算法高效,易于实现。