笔者初入小麦育种这行,最近一段时间看了不少QTL mapping方面的文章。没有对比就没有伤害,有的文章写的很优美,引人入胜;而有的文章只是单纯的罗列结果部分,在讨论部分也是机械的罗列别人发表的结果,故事性和逻辑性较差。虽说做小麦育种志在应用,但发文章最好还是好好构思一下,不要这么随意,偶尔也要提高下档次。不然长此以往,无论对研究还是名声影响都不好。
最近看到一篇刚发表在Molecular Breeding上的文章,题目是Mapping of QTLs associated with resistance to common bunt, tan spot, leaf rust, and stripe rust in a spring wheat population。第一作者Jun Zou,通讯作者 Dean Spaner,第一完成单位是Department of Agricultural, Food and Nutritional Science,Canada和University of Alberta。从题目看是一篇multiple traits的QTL mapping文章,这种类型的文章最容易出现机械的罗列结果,当然也能玩出一些花样。从本文参考文献中可以了解到,使用同样的群体,作者今年已经在PLoS One 和 Crop Science上分别发了一篇文章。对于MB上这篇,个人觉得写得还不错,起码有一个抗病育种故事的轮廓,而且讨论部分占了文章一半以上的篇幅,非常详细。 闲话少说,下面结合这篇文章列举几个可以提高QTL mapping文章档次的做法,欢迎大家补充。 1、育种课题组肯定有株高,开花日期的data,而很多病是跟株高相关的,比如common bunt。与其把四种基本毫不相干的病写在一起,不如稍微深挖一步,找一些相关的traits一起来做,这样文章的结论就丰满多了。比如这篇 “Genetic mapping of common bunt resistance and plant height QTL in wheat” 。
2、同样是在育种上做文章,因为育种课题组的优势是population的两个亲本一般都是advanced lines甚至cultivars,那为何不在mapping population中在鉴定一些“好的”line,把它们的各种表型(抗病然后结合产量,株高,开花日期)以及基因型都列出来,直接开发成cultivar candidates 不是没有可能的,这也可以算是文章中的一个亮点。
3、接下来就是往Genetics上走远一点了。首先比较简单的,开发 User-friendly markers for MAS (e.g. KASP), 然后再在一些种质资源中validate和apply一下,顺便总结一下QTL的优势allele在当地种质资源中的出现频率。比如这篇“A novel QTL associated with dwarf bunt resistance in Idaho 444 winter wheat” 以及公众号介绍过的这篇 “Rapid identification of an adult plant stripe rust resistance gene in hexaploid wheat by high-throughput SNP array genotyping of pooled extremes”
4、既然KASP都开发了,如果可行的话那就再稍微往前走一点,毕竟现在小麦基因组序列已经有了,根据820K SNP marker system 以及Gene Capture marker system,把QTL缩小到10 MB甚至更小也是有可能的,然后做个candidate gene预测。 比如还是公众号中介绍过的这篇“Rapid identification of an adult plant stripe rust resistance gene in hexaploid wheat by high-throughput SNP array genotyping of pooled extremes” (
原文中此处为链接,暂不支持采集
)。
5、当然,经过第4步以后,要是还觉得不过瘾,那就看看凯凯推荐过的这篇文章(
原文中此处为链接,暂不支持采集
)。
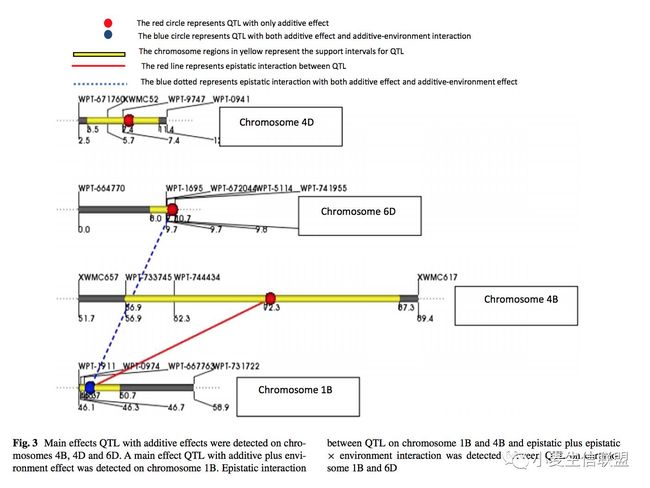
6、最后,实在不想再做实验了,那就再用个软件分析现有的数据吧:Additive/epistatic and QTL by environment interactions using QTLNetwork,如下图:
来源(Genetic mapping of common bunt resistance and plant height QTL in wheat)
参考文献:三篇都是近两年TAG上抗病的文章,大家可以看一下
1 Singh et al., 2016 Genetic mapping of common bunt resistance and plant height QTL in wheat
2 Chen et al., 2016 A novel QTL associated with dwarf bunt resistance in Idaho 444 winter wheat
3 Wu et al., 2017 Rapid identification of an adult plant stripe rust resistance gene in hexaploid wheat by high-throughput SNP array genotyping of pooled extremes
一直没有介绍,我们小编联盟又迎来一位好兄弟,王瑞,目前在爱达荷大学做博后,可以访问他的researchgate主页(https://www.researchgate.net/profile/Rui_Wang78)进一步了解他,也可以在群里与他联系和交流。
欢饮关注“小麦生信联盟”,了解小麦最新研究进展