深度学习如何在医学影像分割上大显神通?——分割网络的三个改进思路
今天看到了一篇介绍深度学习在医学影像分割中的应用的文章,写的很好,分享给更多需要的朋友。原文链接:https://cloud.tencent.com/developer/article/1029037
一、医学影像分割有助于临床工作
图像分割在影像学诊断中大有用处。自动分割能帮助医生确认病变肿瘤的大小,定量评价治疗前后的效果。除此之外,脏器和病灶的识别和甄别也是一项影像科医生的日常工作。CT和磁共振的数据都是三维数据,这意味着对器官和病灶的分割就需要逐层进行。如果都是手工分割的话,会给医生带来繁重的工作量。实际上,已经有很多学者提出了许多医学影像的分割方法,但由于医学影像复杂,分割目标多变,仍有很多自动分割问题等待解决。
近年来深度学习在计算机视觉的各个细分邻域都取得了出色的成绩,那么,深度学习如何帮助医生得到更满意的图像分割结果呢?本文就从三个深度学习分割网络的改进思路谈起,聊一聊这些改进的出发点和具体实现策略。
二、医学影像分割的特点
2.1 分割问题有别于分类问题
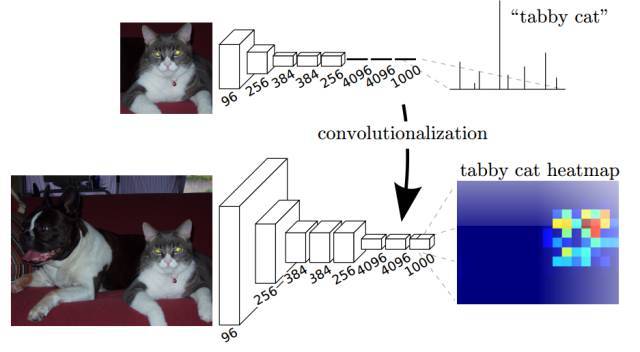
深度学习开始大放异彩的工作,莫过于在ImageNet数据集上,对输入图片的分类了。只要输入图片,就能判断图片中主体所属的类别。然而,和分类问题输入图像输出主体的类别不一样,分割问题需要对每个像素点的类别进行识别。图1就展示了分类和分割的不同点,上图的网络只需要对图片中的主体识别,得到图中有一只小猫的结论,而对下图的分割问题,则需要得出关于这只小猫所在像素点的热力分布图。
图1 分割和分类的区别[1]
2.2 医学影像的分割精度要求高
经典的语义分割更重视像素点的多分类信息,往往对多个类别进行分割,对分割细节要求不高。一个典型的例子就是今年发表于CVPR的文章[2]中,对街景的分割。图2可以看出,它出色的捕捉到了像素的类别信息,但是在分割细节上就不是那么的让人满意了。
图2 工作[2]分割结果,左图为待分割图像,中图为金标准,右图为分割结果
而对于医学影像分割,有一个好消息和一个坏消息:好消息是对于医学影像而言,往往不需要进行多分类,只需要进行病灶或器官的区分即可;而坏消息在于,医学影像所需的分割精度较高,同时稳定性也需要很高,但医学影像往往信噪比相对较低,即使是医生也需要长期的专业训练,而一致性也往往会受到医生经验、疲劳程度和耐心程度的限制。
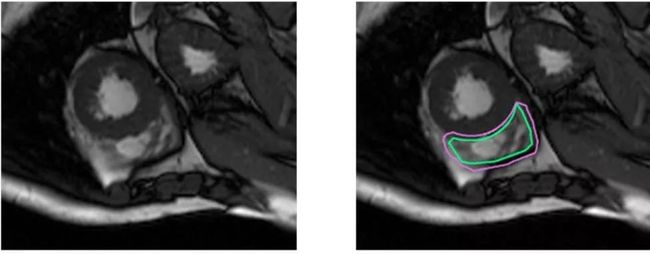
在Kaggle上举办的心室分割比赛要求参赛选手们对心脏的内包膜和外包膜同时进行分割,如图3所示,该任务对分割细节的要求较高。和图2的街景分割任务相比,医学影像的分割细致程度要求较高,不正确或是不稳定的分割将会直接影响心脏射血量的定量计算,从而失去了对医学图像分割的本来意义。
图3 左图为待分割图像,右图为心脏的内包膜和外包膜的分割
三、深度学习分割的开山鼻祖——FCN网络
Fully convolutional network[1]作为分割的代表性工作得到了广泛关注,其核心思想在于将在ImageNet数据集上已经训练好的网络中,深层网络的全连接层改为全卷积层,从而保存分割的位置信息,但是由于最终的分割结果只利用了深层特征映射(feature map),分割结果不够精确。
文章作者也尝试将不同池化(pooling)层的输出进行不同倍数的上采样得到如图4的结果。可以看出,上采样倍数越多(更深层的池化输出)的结果显得越模糊但分类的语义信息较为精确,上采样倍数越少(更浅层的池化输出)的结果保留的分割细节更多而语义信息却不精确。因此,作者指出,该网络中像素类别的精确性和位置的精确性存在着此消彼长的关系。
图4 不同池化层做上采样操作后的结果,最右侧为金标准[1]
那么,面对这样此消彼长的网络特性我们能够通过哪些思路进行改进呢?这些改进思路之间究竟存在怎样的内在逻辑?如果我们不仅仅只利用深层的特征映射会怎么样?
四、分割精确度的提升之道
4.1.1 利用合并操作共享信息:UNet
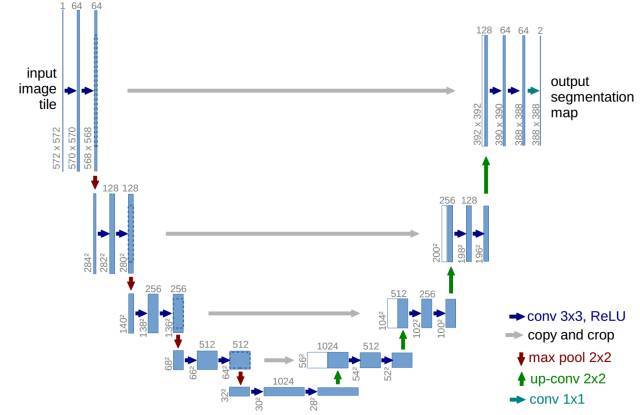
于2015年提出的UNet[3]首先将跨越连接的思想应用于分割问题,并且将其运用在了细胞图像的分割和肝脏CT图像的分割上,得到了当时最为精确的结果。其特点在于利用底层信息补充高层信息,使得分割的精确度大大提升。
图5上图即为UNet的网络结构图,由于它将浅层网络中的输出和深层网络的输出合并(concatenate)在了一起,使得网络在最终输出的时候能够同时考虑浅层信息和深层信息的贡献。和FCN这种对不同层的池化结果进行上采样的思路不同,UNet的这个合并操作有效避免了FCN网络中语义信息和分割细节此消彼长的情况。浅层的输出保存了空域细节信息,深层的输出则是保存了相对抽象的语义信息,二者合二为一就提升了分割效果。
图5 上图为UNet网络结构图,下图为分割结果示意图。UNet由于其外形酷似字母“U”即被取名为UNet。
4.1.2 利用残差操作共享:短程、长程连接的残差网络
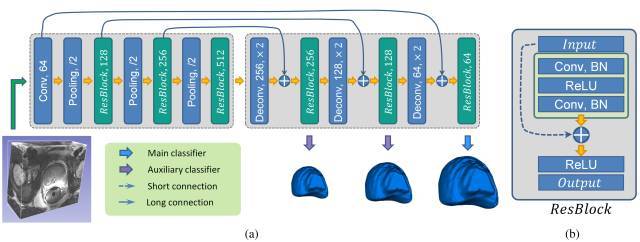
目前,在前列腺分割数据集MICCAI PROMISE12上得分最高的工作[4]也采用了类似的思路——分割细节不清晰怎么办?将网络的浅层输出和网络的高层输出相连接不就行了。图6所示的即为该工作的网络结构图,和UNet类似,它也用一些短程和长程连接将网络的抽象特征和底层的细节相互结合在一起。
图6 工作[4]的网络结构图
不过和UNet的思路不同是,工作[4]将两个网络层连接的方式为矩阵的简单相加操作。图7展示了矩阵相加的朴素计算方式。
图7 矩阵相加操作
而在UNet中,连接浅层网络输出和深层网络输出时,它选取的计算操作为合并(concatenate)。如图8所示,UNet中的合并操作将两个矩阵进行了叠加。
图8 UNet中的合并操作
可以看出,在残差操作中使用的矩阵相加操作增加了矩阵内元素的大小,而UNet中的合并操作则是增加了矩阵的维度大小。最初将不同层相加用于深度学习领域的研究人员[5]认为相加操作增加了网络中数据流的大小,从而能够避免在极深的网络中梯度消失的问题,使得训练极深的网络提升分类准确性成为可能。而有研究人员认为[6],虽然网络深了,但实际上很多神经元在训练的过程中对预测结果的贡献很小,这样的操作有些得不偿失。相加操作对网络的深层和浅层的信息是进行简单的加和操作,而合并操作是原原本本地保存了深层信息和浅层信息,因此,本文倾向于认为合并操作能够更大限度的保留底层信息,在分割问题上更加行之有效。
4.1.2 共享各个网络层的信息:稠密连接的DenseNet
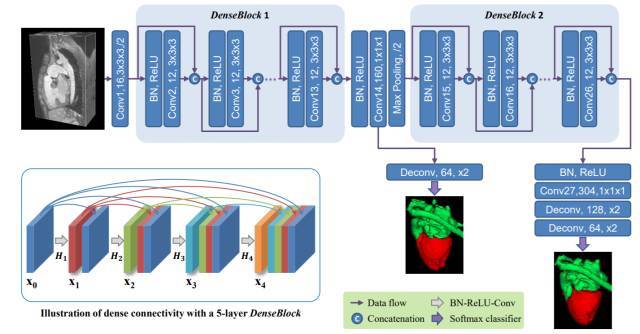
既然网络的某几层能够互相连接提升分割精确度,那么为什么不把网络中任意两层连接起来呢?DenseNet[7]就将网络中任意两层网络连接起来,形成稠密连接的卷积网络(Densely Connected Convolutional Networks)。这样的稠密连接设计能够使得网络最大限度地保存网络的信息流和梯度流,一定程度上抑制网络在迭代过程中梯度消失的问题,并且降低网络中需要学习的参数数量。
目前,在心脏和大血管分割的问题上就有人[8]应用了这种稠密连接的思路,取得了非常棒的结果(图9)。
图9 [8]工作中的网络结构图
4.2 共享图像层间信息
实际上,作为天然的三维数据,CT和MR图像层间的信息也能进行互补,这个时候,三维卷积就能够大展身手了。机智的你或许也已经发现,前文所述的前列腺工作[4]和心脏分割工作[8]都无一例外的使用了三维卷积核进行卷积操作,依靠图像层间的互补信息大大提升了分割精确度。不过,三维的卷积核比较适用于层间变化不是特别明显的图像,例如前列腺MR图像,前列腺在轴位、冠状位以及矢状位上的像素都相对连续,因此使用三维卷积核能够有很大的提升,而对于小血管这样局部变化较大的组织而言,三维的分割似乎不是一个好主意。
五、未来工作展望
聊到这儿,相信大家对深度学习在医学影像分割上的应用有了一定的了解,也对网络的设计与优化有了认识。但是挑战远没有结束,医疗影像数据中还有很多值得挖掘的地方和待解决的问题。在这里笔者就简单地抛砖引玉,其实除了上述的优化网络结构的方法外,还有很多可以用来提升分割效果的思路和策略。
5.1 多模态医学信息结合
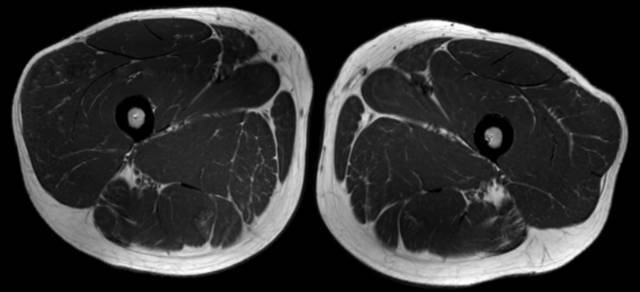
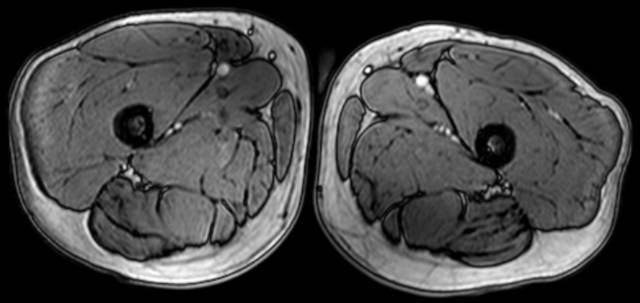
医学检查中往往有多个模态的数据或是多个扫查序列,这些信息可以互相结合提高分割精度。例如,对于下肢肌肉的MR扫查而言,压脂的MR图像中肌肉的边缘信息保存的比较好,而T1w图像中的肌肉纹理却比较清晰,边缘信息可以有助与分割较大的肌肉,而小块的肌肉分割就需要借助肌肉的纹理信息了。
图10 上图为肌肉的T1w图像,下图为肌肉压脂图像
5.1 图像上下文信息互助
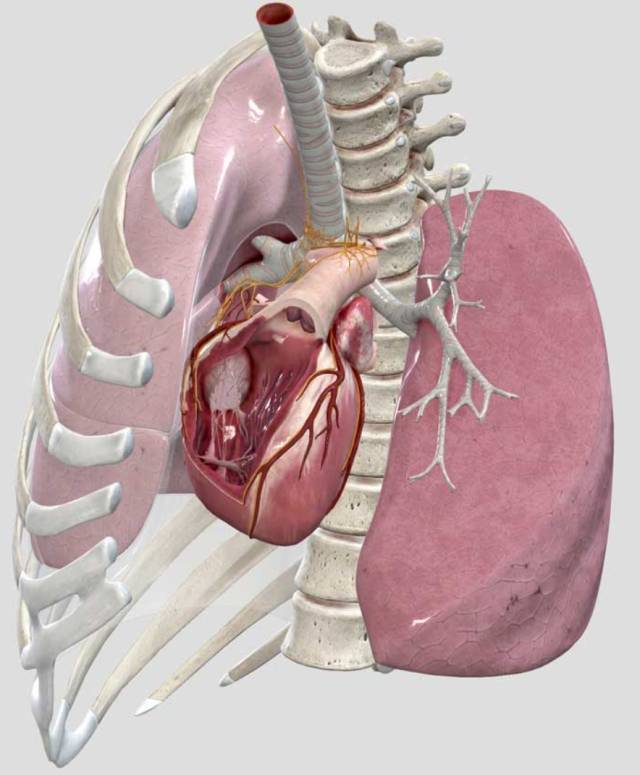
医学影像中除了待查的部位外,总是包含有身体其他部位的信息,这些信息中含有一些解剖标志定位,例如:心脏的位置在3-7肋骨处。而在CT图像中,由于肋骨的亨氏单位(hounsfield unit)较为特殊,导致肋骨相对于各类软组织而言非常容易识别。这样就可以根据CT图像中的肋骨的位置信息,在分割心脏时大大减少算法的求解空间。
图11 心脏和肋骨的相对位置关系示意图
小结一下,本文介绍了三个在医学影像分割问题上深度学习网络的改进思路:1、网络深层抽象信息与底层细节信息共享,用底层信息补充分割细节;2、网络各个层互补信息,通过最大限度的保留网络信息流来提升分割精度;3、将二维卷积操作换为三维卷积操作从而利用图像层间信息互补提升分割精度。不难看出,当前在医学影像分割竞赛上表现出众的三个工作的改进思路都在于扩大参与计算的信息,希望这点能够提供各位读者在改进分割网络时更多的灵感。
对于医学影像分割,深度学习已经有了非常出色的表现,而且越来越多的新思路和新方法用于不断提高分割精度和稳健性,并逐渐减轻医生繁琐的日常工作,降低视觉疲劳的压力,成为临床医生的有力工具。
参考文献
[1]J. Long, E. Shelhamer, and T. Darrell, “Fully convolutional networks for semantic segmentation,” Proc. IEEE Comput. Soc. Conf. Comput. Vis. Pattern Recognit., vol. 07–12–June, pp. 3431–3440, 2015.
[2]H. Zhao, J. Shi, X. Qi, X. Wang, and J. Jia, “Pyramid Scene Parsing Network,” 2016.
[3]O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,” Miccai, pp. 234–241, 2015.
[4]L. Yu, X. Yang, H. Chen, J. Qin, and P.-A. Heng, “Volumetric ConvNets with Mixed Residual Connections for Automated Prostate Segmentation from 3D MR Images,” Thirty-First AAAI Conf. Artif. Intell., pp. 66–72, 2017.
[5]S. Wu, S. Zhong, and Y. Liu, “Deep residual learning for image steganalysis,” Multimed. Tools Appl., pp. 1–17, 2017.
[6]G. Huang, Y. Sun, Z. Liu, D. Sedra, and K. Weinberger, “Deep Networks with Stochastic Depth,” 2016.
[7]G. Huang, Z. Liu, K. Q. Weinberger, and L. van der Maaten, “Densely Connected Convolutional Networks,” 2016.
[8]L. Yu et al., “Automatic 3D cardiovascular MR segmentation with densely-connected volumetric convnets,” Lect. Notes Comput. Sci. (including Subser. Lect. Notes Artif. Intell. Lect. Notes Bioinformatics), vol. 10434 LNCS, pp. 287–295, 2017.