新型冠状病毒包含艾滋病毒序列?是科学家蓄意改造的吗?
太长不看版
印度某研究团队认为,新型冠状病毒带有4个来自于HIV的短肽序列。误导舆论认为新型冠状病毒可能来自人为蓄意改造。
辟谣:印度团队的研究方法和逻辑存在根本性错误。其观点、推论和结论均不成立,有蹭热度之嫌,已受到各国学者质疑和抨击。
1月31日,来自印度理工学院的某科研团队,在美国冷泉港实验室旗下的BioRxiv预印本(不是正规学术期刊)平台上上传(不是发表)了一篇关于新型冠状病毒2019-nCoV的文章[1],引发了广泛的关注。

BioRxiv预印本是啥?

首先要提一句,BioRxiv是个旨在为科学家们提供更快速的文章公开渠道的平台。通常,发表在正规学术期刊上的论文需要经历投稿、初审、完善数据、二次评审等复杂而坎坷的流程。这一过程消耗的时间往往是以月甚至是年来计算。
但有时重要的科学数据等不及漫长的审稿,为了保障时效性,就有了BioRxiv预印本这样的不需要任何审稿过程的预印本平台。任何人任何单位做的任何内容的研究都可以上传到此平台,于是,这个平台上的很多文章都不正规,没经过同行评议和广泛认可。那些上传了预印本却未能在正规学术期刊上发表的文章,大多都有着致命的问题。我们今天要讨论讨论的这篇印度论文也不例外。
这篇文章研究了啥?
让我们来看看本文到底讲了些什么。
首先,作者精心“挑选”了一系列冠状病毒的基因组序列,建立的系统进化树,发现新型冠状病毒和SARS病毒亲缘关系最近。
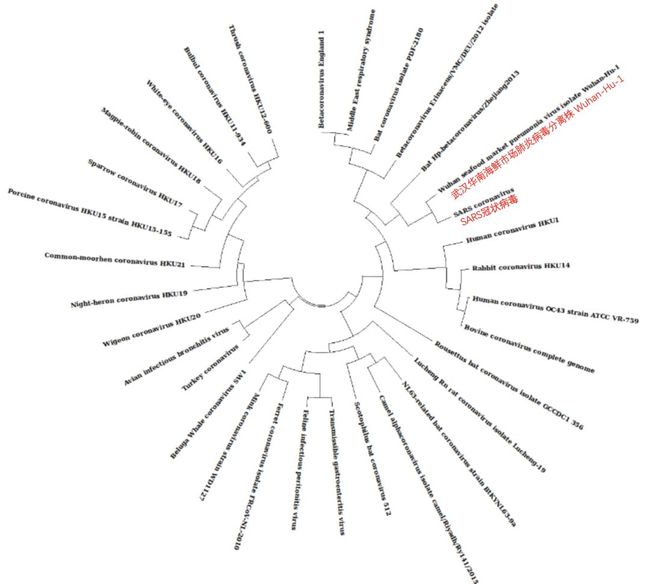
系统进化树解析:软件根据基因序列的一致性进行聚类,一致性越高,那么两者间分支的数量就越少,说明亲缘关系越近。反之则越远,作者指出2019-nCoV和SARS病毒亲缘关系最近 | 参考文献[1]
于是,他们用这次新型冠状病毒的刺突糖蛋白(Spike glycoprotein)序列和SARS病毒的同源蛋白进行了一下比对。然后发现2019-nCoV的刺突糖蛋白比SARS病毒的同源蛋白上多出来了4个独立的短肽段(6-12个氨基酸左右)。作者由此指出,多出来的4个短肽段是2019-nCoV独有的,其他冠状病毒没有。
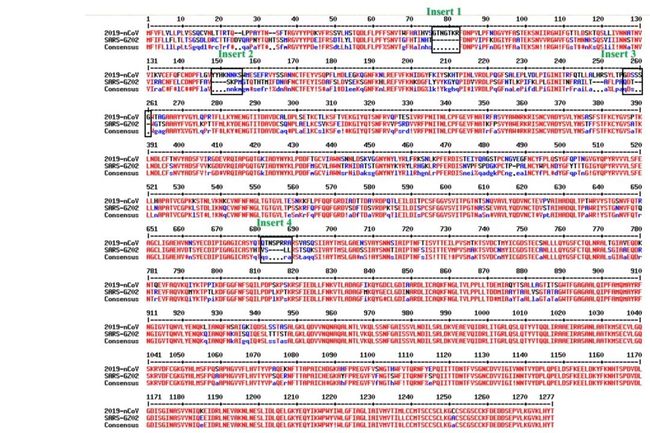
蛋白序列比对解析:数字代表蛋白质中氨基酸的排列序号,每个字母代表一种氨基酸。例如M=甲硫氨酸,F=苯丙氨酸等。每大行中有三小行,第一行是新型冠状病毒2019-nCoV,第二行是SARS,第三行是他们之间的一致序列。其中标记为红色的部分说明两者的刺突糖蛋白在这些位置的氨基酸完全一致;蓝色的部分就是不同的;而“----”代表缺失的。可以看到2019-nCoV的刺突糖蛋白在标记的Insert1-4的位置确实比SARS的刺突糖蛋白多了一些肽段 | 参考文献[1]
为了找到这些肽段的来源,作者又在数据库里搜索了一番——什么?这些多出来的短肽段竟然和HIV-1中的gp120和Gag蛋白高度相似。作者认为这一切并非偶然,通过一些结构模拟,他们猜测这些多出来的短肽段可能对新型冠状病毒与其受体的结合有关。

Insert1和2均可与HIV-1的gp120的部分肽段完全100%匹配。而insert3和4则分别与HIV-1的gp120和gag蛋白有不完全的匹配 | 参考文献[1]
接着,作者含沙射影地将论点往人为操作的方向引导。例如,该文的图3的图注原文说“Modelled homo-trimer spike glycoprotein of 2019-nCoV virus. The inserts from HIV envelop proteinare shown with colored beads, present at the binding site of the protein.”翻译过来的意思是:“新型冠状病毒病毒刺突糖蛋白的同源三聚体结构模拟。我们用几种不同的颜色分别标记了那些来自HIV包膜蛋白的短肽段”。作者故意用了“from”一词让人错以为这些短肽段好像是由HIV嫁接过来的一样。
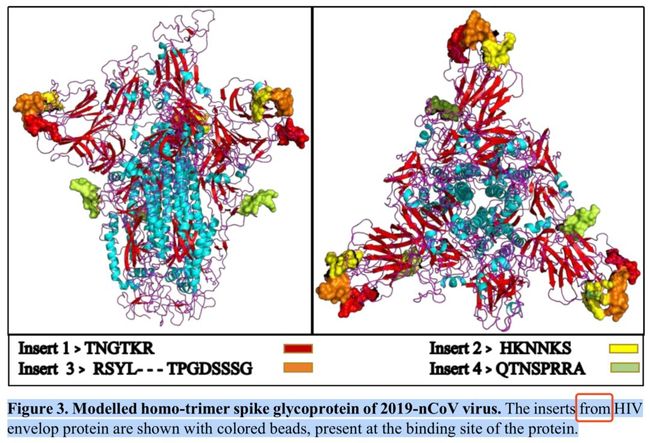
图 | 参考文献[1]
该文在网络上公开之后引发了众人的各种臆想和猜测。有人说,这是中国病毒研究的切尔诺贝利,还有人说这是美国改造病毒的阴谋。

这篇未经同行评议的文章,被当成正经论文在网络上传播
这些论调都基于论文观点正确的前提下衍生出来的。
但事实上,这篇文章无论在研究方法和还是逻辑上都犯了致命的错误,甚至完全经不起推敲。
问题1
以偏概全脱离事实
作者在文章开篇建立的系统进化树可以说异常粗糙。在建立进化树时选择的数据越少,其推导出的结论可信度就越低。作者为了得出自己的观点,挑选了一些不同来源的冠状病毒数据:例如,蝙蝠、兔子、猪、骆驼、家禽,以及2003年的SARS病毒和2014年的MERS病毒来进行聚类分析。

图 | 参考文献[1]
从作者的进化树不难看出,新型冠状病毒和SARS的亲缘关系最近,和蝙蝠来源的冠状病毒反而还有点远。
这一结果与近期我国多位科研工作者所给出的研究结论相悖。我们找到了与这篇论文同一天刊登在《科学》杂志上的一张系统进化树,其中的关键数据就是我国著名病毒学家——石正丽的科研团队贡献的。
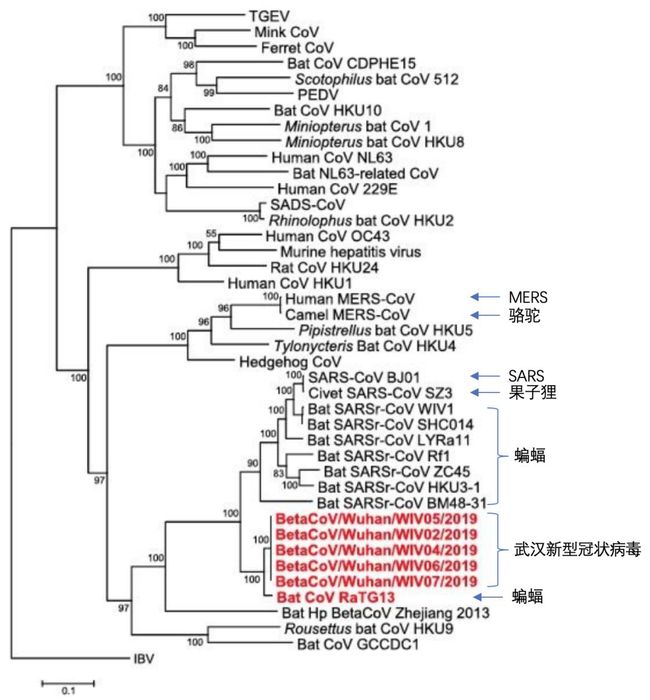
图 | 参考文献[2]
这张图中引用的数据更多,其结论也更加清晰明了。我们可以清楚地看到几个大分支,首先SARS和来自果子狸的SZ3分离株同源度最高,处于同一分支,它们又和一些蝙蝠来源的冠状病毒相似度较高,附属于一个更大的分支内。由此我们可以推断这个病毒传播途径是蝙蝠→果子狸→人。这次的武汉新型冠状病毒和来自蝙蝠的RaTG13分离株的同源度最高,处于另一分支内。两者尽管相似,但大概率具有不同的进化和传播途径。目前由于时间短暂,缺乏更多测序数据,尚无法确定蝙蝠和人之间是否还有中间宿主。
而来自印度的作者无视了RaTG13这个分离株的数据。坚称自己发现的4个短肽段的是武汉新型冠状病毒特有的(暗示人为加入)。事实上,我们在来自蝙蝠粪便的RaTG13病毒分离株的刺突糖蛋白上也能找到这4个短肽段,或完全一致或高度相似。说明新型冠状病毒的这些肽段早就天然存在。作者的论点不攻自破。
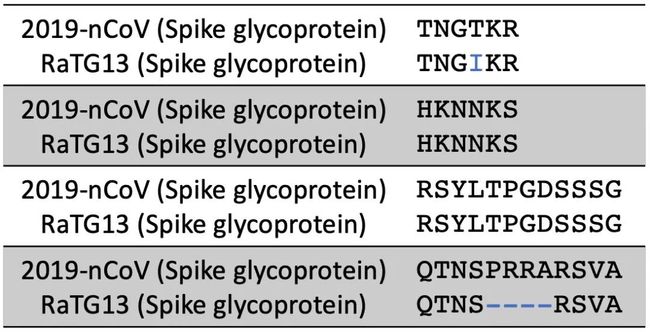
比对结果显示,来自蝙蝠粪便的RaTG13病毒蛋白中也能找到文章中提到的4个短肽段,或完全一致或高度相似 |Nekout
问题2
欲加之罪何患无辞
这篇文章的另一个关键论点,是这4个短肽段和HIV的蛋白上一些肽段高度同源。然而这一论点同样“不小心”忽略了一些数据。
下面这个网站是生物学界最权威的序列比对工具BLAST,有了它,就可以足不出户访问世界上最全的生物学序列数据库,比对任何序列数据。
我们把1号肽段的6个氨基酸放进查询序列框内,然后直接点击页面底部的BLAST按钮。
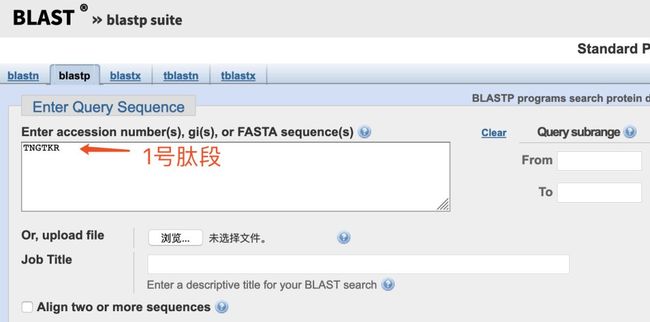

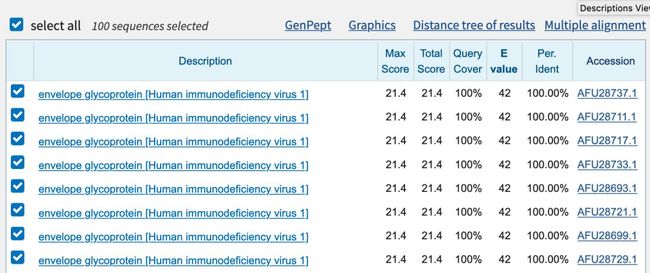
我们会发现,许多蛋白都具有这段6个氨基酸的序列,全都是100%的一致性。它们分别来自不同物种的不同蛋白,从哺乳动物到细菌都有,可能翻完一整页都看不到HIV的信息。

包括细菌和鱼类等很多物种都有这个序列,而作者偏偏“别有用心”地选出了HIV
那么我们再缩小一下搜索范围,把物种限定在HIV里找寻一次,看看效果如何。

嗯。确实能在HIV上找到具有100%一致性的肽段。
以上这些说明了什么呢?
我们打个比方帮助你理解:假设你写了一段话发表在网上,有好事者想证明你的话是抄来的。怎么证明呢?他从你的句子里挑出“我快无聊死了”6个字扔进搜索栏,同时输入某明星的名字,发现完全一致,然后就说你的话是从明星那儿搬来的。但事实上,几乎所有人都在网络上说过相同的话,并不单单是某明星的专属。
蛋白质序列和文字一样,序列越短,随机出现在各个物种的概率就越高,拿6个片段比对出的结果和“我快无聊死了”一样,是没有任何意义的。
说到这里你应该能体会到该论文的荒诞之处了吧。
问题3
牵强附会故弄玄虚
另外,文章的作者一直在引导大家把新型冠状病毒和HIV病毒联系在一起,实际上这两种病毒的感染方式截然不同。
病毒感染的第一步是和细胞上的受体间特异性结合。HIV的常见受体蛋白是人免疫细胞上的表面抗原分化簇4受体蛋白(CD4蛋白)。人体只有部分免疫细胞会合成这个受体蛋白,没有这个受体的细胞很难被感染。而且HIV无法感染除了人以外的其他动物。想在小鼠或其他实验动物中研究HIV的感染过程,首先需要通过转基因技术将人的CD4蛋白转入小鼠的免疫细胞中,否则研究无法进行。

HIV感染细胞的示意图
而2019-nCoV的刺突糖蛋白所结合的受体是血管紧张素转化酶2(ACE2),它在很多黏膜组织细胞中都广泛存在的,这使得它能通过呼吸道及其他黏膜进行传播。
可见,这是两种井水不犯河水的病毒,受体和感染过程都有天壤之别,作者仅从几个的零星的肽段就推断2019-nCoV从HIV那里获得了“超能力”,实属天方夜谭。
这篇文章上传后,已经饱受来自世界各国科学家的质疑和批评,作者自己也表示会根据大家的意见重新修改文章,避免带来更多误解。
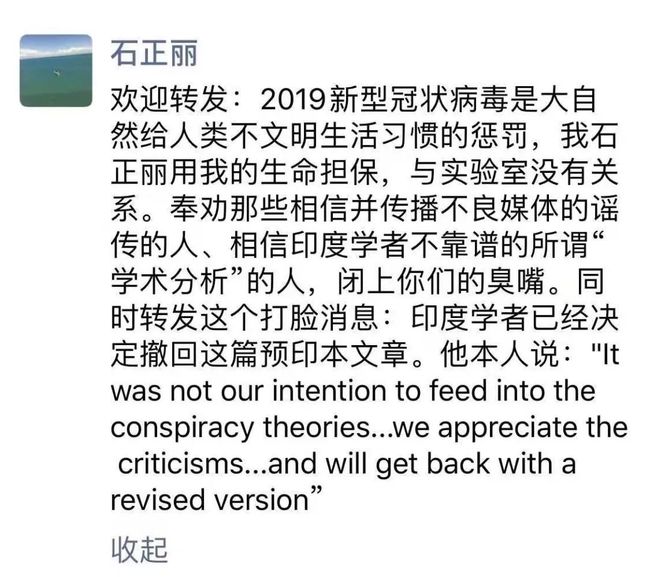
国内外著名科学家纷纷对这篇不靠谱的论文提出批评
所以,这篇文章根本就是一个闹剧。希望大家擦亮双眼,对这类凑热闹不嫌事大的报道提高警惕,莫被阴谋论误导。
*感谢没鹿角的乔巴、南昊提供的写作帮助

作者:Nekout
编辑:Yuki
排版:凝音
题图来源:图虫创意
参考资料:
[1] https://www.biorxiv.org/content/10.1101/2020.01.30.927871v1
[2] https://www.sciencemag.org/news/2020/01/mining-coronavirus-genomes-clues-outbreak-s-origins
欢迎个人转发到朋友圈
