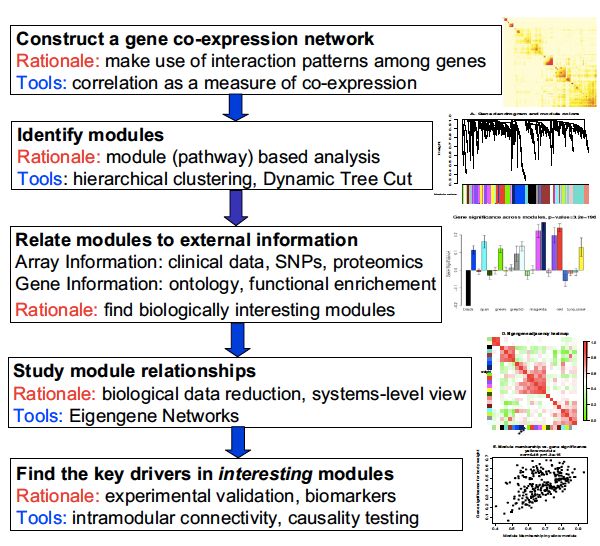
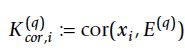- android系统selinux中添加新属性property
辉色投像
1.定位/android/system/sepolicy/private/property_contexts声明属性开头:persist.charge声明属性类型:u:object_r:system_prop:s0图12.定位到android/system/sepolicy/public/domain.te删除neverallow{domain-init}default_prop:property
- 【iOS】MVC设计模式
Magnetic_h
iosmvc设计模式objective-c学习ui
MVC前言如何设计一个程序的结构,这是一门专门的学问,叫做"架构模式"(architecturalpattern),属于编程的方法论。MVC模式就是架构模式的一种。它是Apple官方推荐的App开发架构,也是一般开发者最先遇到、最经典的架构。MVC各层controller层Controller/ViewController/VC(控制器)负责协调Model和View,处理大部分逻辑它将数据从Mod
- 怎么起诉借钱不还的人?怎样起诉欠款不还的人?
影子爱学习
怎么起诉借钱不还的人?怎样起诉欠款不还的人?如果遇到难以解决的法律问题,我们可以匹配专业律师。例如:婚姻家庭(离婚纠纷)、刑事辩护、合同纠纷、债权债务、房产(继承)纠纷、交通事故、劳动争议、人身损害、公司相关法律事务(法律顾问)等咨询推荐手机/微信:15633770876【全国案件皆可】借钱不还起诉对方需要哪些资料起诉欠钱不还的,一般需要的材料包括以下这些:借据、收据、欠条、付款凭证等证据,以及向
- 基于社交网络算法优化的二维最大熵图像分割
智能算法研学社(Jack旭)
智能优化算法应用图像分割算法php开发语言
智能优化算法应用:基于社交网络优化的二维最大熵图像阈值分割-附代码文章目录智能优化算法应用:基于社交网络优化的二维最大熵图像阈值分割-附代码1.前言2.二维最大熵阈值分割原理3.基于社交网络优化的多阈值分割4.算法结果:5.参考文献:6.Matlab代码摘要:本文介绍基于最大熵的图像分割,并且应用社交网络算法进行阈值寻优。1.前言阅读此文章前,请阅读《图像分割:直方图区域划分及信息统计介绍》htt
- DIV+CSS+JavaScript技术制作网页(旅游主题网页设计与制作)云南大理
STU学生网页设计
网页设计期末网页作业html静态网页html5期末大作业网页设计web大作业
️精彩专栏推荐作者主页:【进入主页—获取更多源码】web前端期末大作业:【HTML5网页期末作业(1000套)】程序员有趣的告白方式:【HTML七夕情人节表白网页制作(110套)】文章目录二、网站介绍三、网站效果▶️1.视频演示2.图片演示四、网站代码HTML结构代码CSS样式代码五、更多源码二、网站介绍网站布局方面:计划采用目前主流的、能兼容各大主流浏览器、显示效果稳定的浮动网页布局结构。网站程
- CX8903:Ebike自行车仪表电源方案开发,Ebike智能仪表电源芯片
诚芯微科技
社交电子
CX8903:电动Ebike自行车仪表电源方案开发,Ebike智能仪表电源芯片推荐。电动助力自行车EBIKE凭借其环保、健康、低噪、和便捷等特点,成为了越来越受欢迎的骑行便利交通工具。提供电动Ebike自行车仪表电源方案开发、E-BIKE电动助力自行车仪表供电电源解决方案。CX8903采用100V高压制造工艺(芯片最高耐压可到100V以上),SOP-8L贴片封装,CX8903内置100V/90mΩ
- CX8836:小体积大功率升降压方案推荐(附Demo设计指南)
诚芯微科技
社交电子
CX8836是一颗同步四开关单向升降压控制器,在4.5V-40V宽输入电压范围内稳定工作,持续负载电流10A,能够在输入高于或低于输出电压时稳定调节输出电压,可适用于USBPD快充、车载充电器、HUB、汽车启停系统、工业PC电源等多种升降压应用场合,为大功率TYPE-CPD车载充电器提供最优解决方案。提供CX8836Demo测试、CX8836样品申请及CX8836方案开发技术支持。CX8836同升
- 冬天短期的暴利小生意有哪些?那些小生意适合新手做?
一起高省
短期生意不失为创业的一个商机,不过短期生意的商机是转瞬即逝的,而且这类生意也很难作为长期的生意去做,那冬天短期暴利小生意查看更多关于短期暴利小生意的文章有哪些呢?给大家先推荐一个2023年风口项目吧,真很不错的项目,全程零投资,当做副业来做真的很稳定,不管你什么阶层的人,或多或少都网购吧?你们知道网购是可以拿提成,拿返利,拿分佣的吗?你们知道很多优惠券群里面,天天群主和管理发一些商品吗?他们其实在
- 穷人做什么生意最赚钱?10个适合穷人赚钱的路子?
氧惠爱高省
不管在什么地方,一般都是穷人占大量数,而富人只有少数,但是它们却掌握着大量的财富。对于穷人来说,想要买车、买房等奢侈品就难如登天,因为他们只能通过打工来赚取几千元的月薪。➤推荐网购返利app“氧惠”,一个领隐藏优惠券+现金返利的平台。氧惠只提供领券返利链接,下单全程都在淘宝、京东、拼多多等原平台,更支持抖音、快手电商、外卖红包返利等。(应用市场搜“氧惠”下载,邀请码:521521,全网优惠上氧惠!
- 学习“论语”-第59天
春峰轩
12.14子张问政。子曰:“居之无倦,行之以忠。”子张问为政之道。孔子说:“在位尽职不懈怠,执行政令要忠诚。”12.15子曰:“博学于文,约之以礼,亦可以弗畔矣夫!”孔子说:“君子广泛地学习文献,并且用礼节约束自己,也就不会离经叛道了。”12.16子曰:“君子成人之美,不成人之恶。小人反是。”孔子说:“君子成全别人的好事,而不助长别人的坏处。小人则与此相反行事。”知识点:“成人之美,不成人之恶”贯
- 目前哪里有卖高仿包包,推荐十个渠道已更新
富腕表之家
1、工厂购买,推荐微信:【76929666】目前买的人最多的渠道。2、某宝购买,价格较高,质量没有保障。3、拼夕夕,价格是便宜,但是质量不敢想象。4、专柜购买,数量较少,经常断货,价格也太高不好接受。5、批发市场购买,可遇不可求,一般生活在批发市场附近的,根本不用考虑在哪里买高仿包包分几个级别?在当今的包类市场中,广州作为一个知名的货源地,已经成为高仿包行业的一个重要标志。随着市场的需求增加,高仿
- 一比一复刻手表哪里可以买到?推荐三个可靠渠道
腕表世界
在我国,提及一比一复刻手表,人们总是充满好奇与争议。这种高度仿真的复刻手表,凭借其精湛的工艺、时尚的设计,以及与正品相差无几的质感,深受一部分消费者的喜爱。但与此同时,其背后的侵权争议也一直不断。那么,究竟哪里可以买到这些令人心动的一比一复刻手表呢?腕表咨询微信:10428850一、何为一比一复刻手表?一比一复刻手表,指的是严格按照正版手表的设计、尺寸和工艺制作的仿制品。这些手表在材质、外观、功能
- 高仿包包批发在哪里买最便宜?推荐6个购买渠道
鸿运工作室
高仿包包作为一种时尚单品,受到很多人的喜爱。然而,对于批发高仿包包的人来说,如何找到最便宜的购买渠道是一个关键问题。本文将为您推荐6个购买高仿包包最便宜的渠道,帮助您更好地满足批发需求。咨询加微信:FB2260(下单赠送精美礼品)1.义乌国际商贸城义乌国际商贸城是中国最大的小商品批发市场之一,也是高仿包包批发的热门地点。这里有众多的批发商聚集,提供了各种各样的高仿包包,价格相对较低。您可以在这里找
- 《如不承诺天长地久,怎会相遇细水长流》文/苏暖人
北京大数据苏焕之
《如不承诺天长地久,怎会相遇细水长流》文/苏暖人原创——莫转载粘贴有人选择昙花一现,如大理的花海,有人选择细水长流,如雨夜的浪漫。都说,五分喜欢的人恨不得将他挂在嘴边,十分喜欢的人却只舍得放在心里边了,在爱情眼里,对方说的每一句话都在乎你的感受,TA的眼里也只有你,我想也是这样!说起我的爱情,我也喜欢过一个忧郁的女孩,她喜欢的男孩不喜欢她,于是我成了她倾诉的朋友+备胎,一年来我们互相推荐伤感的歌曲
- 关于Mysql 中 Row size too large (> 8126) 错误的解决和理解
秋刀prince
mysqlmysql数据库
提示:啰嗦一嘴,数据库的任何操作和验证前,一定要记得先备份!!!不会有错;文章目录问题发现一、问题导致的可能原因1、页大小2、行格式2.1compact格式2.2Redundant格式2.3Dynamic格式2.4Compressed格式3、BLOB和TEXT列二、解决办法1、修改页大小(不推荐)2、修改行格式3、修改数据类型为BLOB和TEXT列4、其他优化方式(可以参考使用)4.1合理设置数据
- 推荐算法_隐语义-梯度下降
_feivirus_
算法机器学习和数学推荐算法机器学习隐语义
importnumpyasnp1.模型实现"""inputrate_matrix:M行N列的评分矩阵,值为P*Q.P:初始化用户特征矩阵M*K.Q:初始化物品特征矩阵K*N.latent_feature_cnt:隐特征的向量个数max_iteration:最大迭代次数alpha:步长lamda:正则化系数output分解之后的P和Q"""defLFM_grad_desc(rate_matrix,l
- CentOS 7官方源停服,配置本机光盘yum源
码哝小鱼
linux运维centoslinux运维
1、挂载系统光盘mkdir/mnt/isomount-oloop/tools/CentOS-7-x86_64-DVD-1810.iso/mnt/isocd/mnt/iso/Packages/rpm-ivh/mnt/iso/Packages/yum-utils-1.1.31-50.el7.noarch.rpm(图形界面安装,默契已安装)如安装yum-utils依赖错误,按提示安装依赖包rpm-ivh
- 用Python实现读取统计单词个数
程序媛了了
python游戏java
完整实例代码:fromcollectionsimportCounterdefpythonit():danci={}withopen("pythonit.txt","r",encoding="utf-8")asf:foriinf:words=i.strip().split()forwordinwords:ifwordnotindanci:danci[word]=1else:danci[word]+=
- 我与《红楼梦》‖纪念曹雪芹出生307周年!归海逸舟是周成功子阳佳乐
归海逸舟是周成功子阳佳乐
【今日作家推荐】中国古典小说之首《红楼梦》,其作者曹雪芹是文坛泰斗。约1715年5月28日,曹雪芹出生。所以,今天推荐的是中国人众所周知的作家——曹雪芹。曹雪芹在世界读者心目中也影响广大,可以与西方世界引以为豪的莎士比亚、歌德等媲美。1、我与《红楼梦》我一直想写一篇和《红楼梦》相关的文章,现在机会终于来了!《红楼梦》作为我国家喻户晓的文学名著,其影响是空前的。还在我很小的时候,姥姥经常讲《红楼梦》
- 可以赚钱的app,你们都在用哪些?
配音新手圈
1.七猫免费小说2.有柿3.番茄小说兼职副业推荐公众号,配音新手圈,声优配音圈,新配音兼职圈,配音就业圈,鼎音副业,有声新手圈,每天更新各种远程工作与在线兼职,职位包括:写手、程序开发、剪辑、设计、翻译、配音、无门槛、插画、翻译、等等。。。每日更新兼职。4.速读免费小说5.得间免费小说6.快手7.快手极速8.抖音火山版(可提0.2,可能我懒赚的慢,但真不推荐)9.拼多多10.淘宝11.点淘12.美
- 上班族可以做线上副业兼职有哪些?盘点7个适合上班族做的副业兼职!
高省APP大九
对于许多上班族来说,工资往往不能满足他们的生活需求,因此许多人开始寻找副业来增加收入。以下是一些适合普通人的副业赚钱路子,希望能给您带来一些灵感。1、做好物推荐现在很多职场人其实有大量的个人时间,只不过这些个人时间比较碎片化,他们不能够很好的利用起来,其实可以利用这些碎片化的时间去做副业,比如做好物推荐。在网上有很多的平台,比如头条抖音等等都开通了一个商品的分销功能,只要你发布相关的视频或者文章,
- 手机上有什么兼职可以做?网上兼职 一单一结 手机就可以做?
优惠券高省
建议上班族和全职宝妈把空闲时间拿出来一点做做副业,什么也不耽搁还能多一笔收入!推荐大家一定要试一试!!!只要有手机就可以做,下面小编就为大家推荐用手机就可以做的三类网上兼职工作。一,高省APP高省APP佣金更高,模式更好,终端用户不流失。【高省】是一个自用省钱佣金高,分享推广赚钱多的平台,百度有几百万篇报道,也期待你的加入。万方导师高省邀请码005500,注册送双皇冠会员,送万元推广大礼包,教你如
- 一分钟学会刷牙,受用终生!
好易康
讲真,刷了十几二十年牙,没刷对过一次......来来来,划重点,更重要的是执行:①每天刷牙2次,②每次刷牙2~3分钟,③每3个月更换牙刷。最后,请使用正确的刷牙方法:巴氏(BASS)刷牙法undefined_腾讯视频视频来源ADA美国牙医协会巴氏刷牙法又称龈沟清扫法或水平颤动法。是由美国牙科协会推荐的一种有效去除龈缘附近及龈沟内菌斑的方法。刷牙不仅是刷牙齿,同时也要刷牙龈。因为口腔与细菌的战场就在
- 光盘文件系统 (iso9660) 格式解析
穷人小水滴
光盘文件系统iso9660denoGNU/Linuxjavascript
越简单的系统,越可靠,越不容易出问题.光盘文件系统(iso9660)十分简单,只需不到200行代码,即可实现定位读取其中的文件.参考资料:https://wiki.osdev.org/ISO_9660相关文章:《光盘防水嘛?DVD+R刻录光盘泡水实验》https://blog.csdn.net/secext2022/article/details/140583910《光驱的内部结构及日常使用》ht
- 莆田鞋在哪买?推荐二个靠谱渠道
美鞋之家
莆田鞋在哪里买,莆田鞋一般在实体店或莆田鞋店购买,我觉得很多莆田鞋都是在莆田、广州、上海的鞋类批发市场购买的,价格非常方便。如果你想做莆田鞋生意,你可以去这些地方,如果你只想买一双莆田鞋穿,你可以在天猫和淘宝上买,因为淘宝的费用一般比较便宜。详细咨询VX→a40273莆田鞋在哪里买1、微商上购买,做莆田鞋微商代理的人群非常多,价格也比较实惠,但是也难免遇见高价卖的微商人群。其实莆田鞋的水很深,买之
- Table列表复现框实现【勾选-搜索-再勾选】
~四时春~
java开发语言elementuivue
Table列表复现框实现【勾选-搜索-再勾选】概要整体架构流程代码实现技术细节注意参考文献概要最近在开发时遇到一个问题,在进行表单渲染时,正常选中没有问题,单如果需要搜索选中时,一个是已选中的不会回填,二是在搜索的结果中进行选中,没有实现,经过排查,查找资料后实现。例如:整体架构流程具体的实现效果如下:代码实现{{scope.row.userName}}已选区{{userItem.userName
- 分享十个渠道:高仿手表在哪里买最好的优选渠道
百奢之家
1、工厂购买,推荐靠谱手表商家微信:【15266277】目前买的人最多的渠道。2、某宝购买,价格较高,质量没有保障。3、拼夕夕,价格是便宜,但是质量不敢想象。4、专柜购买,数量较少,经常断货,价格也太高不好接受。5、批发市场购买,可遇不可求,一般生活在批发市场附近的,根本不用考虑在哪里买手表作为一件配饰品,不仅可以提升整体造型品味,还能展现个人的时尚态度。而对于很多人来说,高仿手表是一种经济实惠又
- 《跃迁》5/7-5组-橙子-张静12.16
静言物于
【便签5】【片段来源】《跃迁:成为高手的技术》第四章【R原文】一位客户咨询时抱怨:“这个我做不到。”我问他:“如果我请你现在出去裸奔,你能做到吗?”“这个我也做不到”“其实并不是做不到,而是不愿意做,或者不想承担裸奔的代价吧。你不是做不到,而是选择不去做。如果有一天你裸奔能救自己家人、孩子,也许就能做到了。”为什么要做这个区分?如果一个人经常和自己说“做不到”,他的能力范围会越来越小,会成为一个无
- 【树一线性代数】005入门
Owlet_woodBird
算法
Index本文稍后补全,推荐阅读:https://blog.csdn.net/weixin_60702024/article/details/141874376分析实现总结本文稍后补全,推荐阅读:https://blog.csdn.net/weixin_60702024/article/details/141874376已知非空二叉树T的结点值均为正整数,采用顺序存储方式保存,数据结构定义如下:t
- 更改npm镜像源为淘宝镜像
骆小骆
基于node.js
npm常用指令后缀*最近复习了一下node.js整理了一下跟node.js相关的指令后缀*--save、-S参数意思是把模块的版本信息保存到dependencies(生产环境依赖)中,即你的package.json文件的dependencies字段中;–--save-dev、-D参数意思是把模块版本信息保存到devDependencies(开发环境依赖)中,即你的package.json文件的de
- 多线程编程之理财
周凡杨
java多线程生产者消费者理财
现实生活中,我们一边工作,一边消费,正常情况下会把多余的钱存起来,比如存到余额宝,还可以多挣点钱,现在就有这个情况:我每月可以发工资20000万元 (暂定每月的1号),每月消费5000(租房+生活费)元(暂定每月的1号),其中租金是大头占90%,交房租的方式可以选择(一月一交,两月一交、三月一交),理财:1万元存余额宝一天可以赚1元钱,
- [Zookeeper学习笔记之三]Zookeeper会话超时机制
bit1129
zookeeper
首先,会话超时是由Zookeeper服务端通知客户端会话已经超时,客户端不能自行决定会话已经超时,不过客户端可以通过调用Zookeeper.close()主动的发起会话结束请求,如下的代码输出内容
Created /zoo-739160015
CONNECTEDCONNECTED
.............CONNECTEDCONNECTED
CONNECTEDCLOSEDCLOSED
- SecureCRT快捷键
daizj
secureCRT快捷键
ctrl + a : 移动光标到行首ctrl + e :移动光标到行尾crtl + b: 光标前移1个字符crtl + f: 光标后移1个字符crtl + h : 删除光标之前的一个字符ctrl + d :删除光标之后的一个字符crtl + k :删除光标到行尾所有字符crtl + u : 删除光标至行首所有字符crtl + w: 删除光标至行首
- Java 子类与父类这间的转换
周凡杨
java 父类与子类的转换
最近同事调的一个服务报错,查看后是日期之间转换出的问题。代码里是把 java.sql.Date 类型的对象 强制转换为 java.sql.Timestamp 类型的对象。报java.lang.ClassCastException。
代码:
- 可视化swing界面编辑
朱辉辉33
eclipseswing
今天发现了一个WindowBuilder插件,功能好强大,啊哈哈,从此告别手动编辑swing界面代码,直接像VB那样编辑界面,代码会自动生成。
首先在Eclipse中点击help,选择Install New Software,然后在Work with中输入WindowBui
- web报表工具FineReport常用函数的用法总结(文本函数)
老A不折腾
finereportweb报表工具报表软件java报表
文本函数
CHAR
CHAR(number):根据指定数字返回对应的字符。CHAR函数可将计算机其他类型的数字代码转换为字符。
Number:用于指定字符的数字,介于1Number:用于指定字符的数字,介于165535之间(包括1和65535)。
示例:
CHAR(88)等于“X”。
CHAR(45)等于“-”。
CODE
CODE(text):计算文本串中第一个字
- mysql安装出错
林鹤霄
mysql安装
[root@localhost ~]# rpm -ivh MySQL-server-5.5.24-1.linux2.6.x86_64.rpm Preparing... #####################
- linux下编译libuv
aigo
libuv
下载最新版本的libuv源码,解压后执行:
./autogen.sh
这时会提醒找不到automake命令,通过一下命令执行安装(redhat系用yum,Debian系用apt-get):
# yum -y install automake
# yum -y install libtool
如果提示错误:make: *** No targe
- 中国行政区数据及三级联动菜单
alxw4616
近期做项目需要三级联动菜单,上网查了半天竟然没有发现一个能直接用的!
呵呵,都要自己填数据....我了个去这东西麻烦就麻烦的数据上.
哎,自己没办法动手写吧.
现将这些数据共享出了,以方便大家.嗯,代码也可以直接使用
文件说明
lib\area.sql -- 县及县以上行政区划分代码(截止2013年8月31日)来源:国家统计局 发布时间:2014-01-17 15:0
- 哈夫曼加密文件
百合不是茶
哈夫曼压缩哈夫曼加密二叉树
在上一篇介绍过哈夫曼编码的基础知识,下面就直接介绍使用哈夫曼编码怎么来做文件加密或者压缩与解压的软件,对于新手来是有点难度的,主要还是要理清楚步骤;
加密步骤:
1,统计文件中字节出现的次数,作为权值
2,创建节点和哈夫曼树
3,得到每个子节点01串
4,使用哈夫曼编码表示每个字节
- JDK1.5 Cyclicbarrier实例
bijian1013
javathreadjava多线程Cyclicbarrier
CyclicBarrier类
一个同步辅助类,它允许一组线程互相等待,直到到达某个公共屏障点 (common barrier point)。在涉及一组固定大小的线程的程序中,这些线程必须不时地互相等待,此时 CyclicBarrier 很有用。因为该 barrier 在释放等待线程后可以重用,所以称它为循环的 barrier。
CyclicBarrier支持一个可选的 Runnable 命令,
- 九项重要的职业规划
bijian1013
工作学习
一. 学习的步伐不停止 古人说,活到老,学到老。终身学习应该是您的座右铭。 世界在不断变化,每个人都在寻找各自的事业途径。 您只有保证了足够的技能储
- 【Java范型四】范型方法
bit1129
java
范型参数不仅仅可以用于类型的声明上,例如
package com.tom.lang.generics;
import java.util.List;
public class Generics<T> {
private T value;
public Generics(T value) {
this.value =
- 【Hadoop十三】HDFS Java API基本操作
bit1129
hadoop
package com.examples.hadoop;
import org.apache.hadoop.conf.Configuration;
import org.apache.hadoop.fs.FSDataInputStream;
import org.apache.hadoop.fs.FileStatus;
import org.apache.hadoo
- ua实现split字符串分隔
ronin47
lua split
LUA并不象其它许多"大而全"的语言那样,包括很多功能,比如网络通讯、图形界面等。但是LUA可以很容易地被扩展:由宿主语言(通常是C或 C++)提供这些功能,LUA可以使用它们,就像是本来就内置的功能一样。LUA只包括一个精简的核心和最基本的库。这使得LUA体积小、启动速度快,从 而适合嵌入在别的程序里。因此在lua中并没有其他语言那样多的系统函数。习惯了其他语言的字符串分割函
- java-从先序遍历和中序遍历重建二叉树
bylijinnan
java
public class BuildTreePreOrderInOrder {
/**
* Build Binary Tree from PreOrder and InOrder
* _______7______
/ \
__10__ ___2
/ \ /
4
- openfire开发指南《连接和登陆》
开窍的石头
openfire开发指南smack
第一步
官网下载smack.jar包
下载地址:http://www.igniterealtime.org/downloads/index.jsp#smack
第二步
把smack里边的jar导入你新建的java项目中
开始编写smack连接openfire代码
p
- [移动通讯]手机后盖应该按需要能够随时开启
comsci
移动
看到新的手机,很多由金属材质做的外壳,内存和闪存容量越来越大,CPU速度越来越快,对于这些改进,我们非常高兴,也非常欢迎
但是,对于手机的新设计,有几点我们也要注意
第一:手机的后盖应该能够被用户自行取下来,手机的电池的可更换性应该是必须保留的设计,
- 20款国外知名的php开源cms系统
cuiyadll
cms
内容管理系统,简称CMS,是一种简易的发布和管理新闻的程序。用户可以在后端管理系统中发布,编辑和删除文章,即使您不需要懂得HTML和其他脚本语言,这就是CMS的优点。
在这里我决定介绍20款目前国外市面上最流行的开源的PHP内容管理系统,以便没有PHP知识的读者也可以通过国外内容管理系统建立自己的网站。
1. Wordpress
WordPress的是一个功能强大且易于使用的内容管
- Java生成全局唯一标识符
darrenzhu
javauuiduniqueidentifierid
How to generate a globally unique identifier in Java
http://stackoverflow.com/questions/21536572/generate-unique-id-in-java-to-label-groups-of-related-entries-in-a-log
http://stackoverflow
- php安装模块检测是否已安装过, 使用的SQL语句
dcj3sjt126com
sql
SHOW [FULL] TABLES [FROM db_name] [LIKE 'pattern']
SHOW TABLES列举了给定数据库中的非TEMPORARY表。您也可以使用mysqlshow db_name命令得到此清单。
本命令也列举数据库中的其它视图。支持FULL修改符,这样SHOW FULL TABLES就可以显示第二个输出列。对于一个表,第二列的值为BASE T
- 5天学会一种 web 开发框架
dcj3sjt126com
Web框架framework
web framework层出不穷,特别是ruby/python,各有10+个,php/java也是一大堆 根据我自己的经验写了一个to do list,按照这个清单,一条一条的学习,事半功倍,很快就能掌握 一共25条,即便很磨蹭,2小时也能搞定一条,25*2=50。只需要50小时就能掌握任意一种web框架
各类web框架大同小异:现代web开发框架的6大元素,把握主线,就不会迷路
建议把本文
- Gson使用三(Map集合的处理,一对多处理)
eksliang
jsongsonGson mapGson 集合处理
转载请出自出处:http://eksliang.iteye.com/blog/2175532 一、概述
Map保存的是键值对的形式,Json的格式也是键值对的,所以正常情况下,map跟json之间的转换应当是理所当然的事情。 二、Map参考实例
package com.ickes.json;
import java.lang.refl
- cordova实现“再点击一次退出”效果
gundumw100
android
基本的写法如下:
document.addEventListener("deviceready", onDeviceReady, false);
function onDeviceReady() {
//navigator.splashscreen.hide();
document.addEventListener("b
- openldap configuration leaning note
iwindyforest
configuration
hostname // to display the computer name
hostname <changed name> // to change
go to: /etc/sysconfig/network, add/modify HOSTNAME=NEWNAME to change permenately
dont forget to change /etc/hosts
- Nullability and Objective-C
啸笑天
Objective-C
https://developer.apple.com/swift/blog/?id=25
http://www.cocoachina.com/ios/20150601/11989.html
http://blog.csdn.net/zhangao0086/article/details/44409913
http://blog.sunnyxx
- jsp中实现参数隐藏的两种方法
macroli
JavaScriptjsp
在一个JSP页面有一个链接,//确定是一个链接?点击弹出一个页面,需要传给这个页面一些参数。//正常的方法是设置弹出页面的src="***.do?p1=aaa&p2=bbb&p3=ccc"//确定目标URL是Action来处理?但是这样会在页面上看到传过来的参数,可能会不安全。要求实现src="***.do",参数通过其他方法传!//////
- Bootstrap A标签关闭modal并打开新的链接解决方案
qiaolevip
每天进步一点点学习永无止境bootstrap纵观千象
Bootstrap里面的js modal控件使用起来很方便,关闭也很简单。只需添加标签 data-dismiss="modal" 即可。
可是偏偏有时候需要a标签既要关闭modal,有要打开新的链接,尝试多种方法未果。只好使用原始js来控制。
<a href="#/group-buy" class="btn bt
- 二维数组在Java和C中的区别
流淚的芥末
javac二维数组数组
Java代码:
public class test03 {
public static void main(String[] args) {
int[][] a = {{1},{2,3},{4,5,6}};
System.out.println(a[0][1]);
}
}
运行结果:
Exception in thread "mai
- systemctl命令用法
wmlJava
linuxsystemctl
对比表,以 apache / httpd 为例 任务 旧指令 新指令 使某服务自动启动 chkconfig --level 3 httpd on systemctl enable httpd.service 使某服务不自动启动 chkconfig --level 3 httpd off systemctl disable httpd.service 检查服务状态 service h