最近在使用Gretna对fMRI数据进行预处理的时候,总是遇到卡死的状态,仔细研究了一下Gretna的源代码,发现其Workflow部分使用了PSOM的工具包,这个问题就没办法调试了,于是开始思考是否能够有其他的工具来完成同样的东东。
通过一段时间的调研,发现了这个Python的框架(截止目前为止,该框架的版本为1.0.4),于是有了这篇文章。这是一个自己学习的笔记,准备慢慢记录Nipype,主要参考的是Nipype的网站和文档,具体链接如下:
http://nipype.readthedocs.io/en/latest/quickstart.html
http://nipype.readthedocs.io/en/latest/documentation.html
Nipype是一个用Python编写的框架,主要用于处理神经影像学的各类数据,其集成了大部分常用的神经影像学数据处理软件,包括ANTS, SPM, FSL, FreeSurfer, Camino, MRtrix, MNE, AFNI, Slicer等,其中我只用过SPM和FSL,用这个框架可以自定义工作流,使不同的软件协同工作。
1. 安装
需要说明的是,截止目前为止,Nipype在类Unix系统上会运行的更稳定一些,例如Linux、MacOS都是完美支持的,对于Windows则不完全支持(虽然能够通过Conda和Pip安装上,但是运行的时候会报错,似乎其中引用了一个getpwd的包,而这个包只支持Linux等系统)。
Nipye的安装主要有三种方式:
1.1 Docker
如果想要在Windows系统上使用Nipype,恐怕除了虚拟机意外,最简单便捷的方式就是使用Docker了,在使用Docker时,只需要从镜像库将其拖下来就好了,需要执行如下命令:
docker pull nipype/nipype
至于拖下来之后怎么用,目前我还没有研究,等有机会的看一下。
1.2 Conda
Conda的安装也非常简单,只需要执行如下命令就可以了,当然为了便于演示和操作,建议也安装Jupyter。(我用的就是这种方法)
conda install --channel conda-forge nipype
conda install jupyter
如果要绘制图片,切记要安装以下两个包,否则会报错的。
conda install graphviz, pydot
1.3 Pip
Pip的安装同样简单(我都不好意思再打这几个字了)
pip install nipype
如果想要安装nipype的所有可选的特征,就需要使用如下命令(我没有使用)
pip install nipype[all]
2.基本概念
从我目前的理解来看,Nipype应该是一个比较面向于开发人员的框架,并不太适合普通的医师进行数据处理,这可能也是国内并未普及的原因。Nipype是一个有些类似于TensorFlow这样的先定义计算,在进行执行的函数式编程框架(目前的理解,并未仔细研究函数式编程的定义),要想充分运用Nipype,发挥它的最大功效,先要需要充分理解三个基本概念:
- Interface
- Node
- Workflow
2.1 Interface
Interface是封装了各类脑图像处理软件的Python包。在使用时需要进行如下引用。由于没有实际使用过,暂时先用官方的例子来进行一下演示,待日后对Interface的理解进一步深入了,再来演示更复杂的例子。(话说,似乎软件文档的编写者,对于FSL更熟悉一些,所选的例子全部都是FSL的)
from nipype.interfaces.fsl import BET
要想知道BET这个包应该怎么用,可以运行:
BET.help()
我自己试了一下SPM的接口,与FSL的使用方法基本一致。
import nipype.interfaces.spm as spm
spm.DicomImport.help()
运行上述代码之后,会看到如下结果:
Uses spm to convert DICOM files to nii or img+hdr.
Examples
--------
>>> import nipype.interfaces.spm.utils as spmu
>>> di = spmu.DicomImport()
>>> di.inputs.in_files = ['functional_1.dcm', 'functional_2.dcm']
>>> di.run() # doctest: +SKIP
Inputs::
[Mandatory]
in_files: (a list of items which are an existing file name)
dicom files to be converted
[Optional]
format: ('nii' or 'img', nipype default value: nii)
output format.
icedims: (a boolean, nipype default value: False)
If image sorting fails, one can try using the additional SIEMENS
ICEDims information to create unique filenames. Use this only if
there would be multiple volumes with exactly the same file names.
ignore_exception: (a boolean, nipype default value: False)
Print an error message instead of throwing an exception in case the
interface fails to run
matlab_cmd: (a unicode string)
matlab command to use
mfile: (a boolean, nipype default value: True)
Run m-code using m-file
output_dir: (a unicode string, nipype default value:
./converted_dicom)
output directory.
output_dir_struct: ('flat' or 'series' or 'patname' or 'patid_date'
or 'patid' or 'date_time', nipype default value: flat)
directory structure for the output.
paths: (a list of items which are a directory name)
Paths to add to matlabpath
use_mcr: (a boolean)
Run m-code using SPM MCR
use_v8struct: (a boolean, nipype default value: True)
Generate SPM8 and higher compatible jobs
Outputs::
out_files: (a list of items which are an existing file name)
converted files
References::
None
从中可知,该接口是用于将DICOM数据转换为nii或者img+hdr,其中有一个必要的输入参数in_files,有一堆可选参数,例如:用format确定转换为nii还是img。
2.2 Node
Node是一个用于执行某一项功能的对象,执行的功能可以由Interface完成,也可以由自己定义的函数完成,还可以使用外部的脚本。
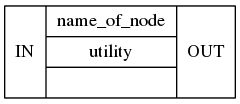
一个Node通常由以下几个部分组成:
- IN:至少一个输入
- OUT:至少一个输出
- name:节点的名称
-
interface:执行的单元(如函数、Interface等)
仍然使用上面的例子,制作一个执行将DICOM转换为Nifti任务的Node,需要执行以下代码:
import nipype.interfaces.spm as spm
from nipype import Node
node = Node(spm.DicomImport(), name='dcm2nii')
此时,node就变成了一个能执行Dicom2nii任务的节点了,执行help命令,可以看到其参数与DicomImport的参数是完全一样的。
除了使用Interface以外(这恐怕是最常见的应用了),还可以自定义函数,比如这样(这个例子来源于官方的文档):
from nipype import Node, Function
def add_two(x_input):
return x_input + 2
addtwo = Node(Function(input_names=["x_input"],
output_names=["val_output"],
function=add_two),
name='add_node')
addtwo.inputs.x_input = 4
addtwo.run()
其实,前面没有提到,单独的Interface也可以执行run
2.3 Workflow
从直观上而言,Workflow可以认为是由多个Node组成的一张有向无环图(Directed Acyclic Graphs,DAG),其中定义了数据的走向。但是,Workflow又不是简单的一个Node的组成,其中还执行了Cache,也就是说如果某些步骤已经执行过,而参数又没有进行其他的调整,那么再次运行Workflow的时候,就不会再重复执行已经执行过的步骤,这一点比Gretna这种软件要好太多了。
官方文档提供了一张表格,写明了其和Interface的主要区别:
Workflow的例子我也还没有写过,先用官方的例子代替一下吧,官方给出的例子仍然是FSL的:
from nipype import Node, Workflow
from nipype.interfaces import fsl
from os.path import abspath
in_file = abspath("/data/ds000114/sub-01/ses-test/anat/sub-01_ses-test_T1w.nii.gz")
#先定义三个Node
skullstrip = Node(fsl.BET(in_file=in_file, mask=True), name="skullstrip")
smooth = Node(fsl.IsotropicSmooth(in_file=in_file, fwhm=4), name="smooth")
mask = Node(fsl.ApplyMask(), name="mask")
#再定义一个Workflow
wf = Workflow(name="smoothflow", base_dir="/output/working_dir")
# 需要使用connect函数将Node加入到Workflow中并进行连接
wf.connect(skullstrip, "mask_file", mask, "mask_file")
wf.connect([(smooth, mask, [("out_file", "in_file")])])
这样就定义完了一个Workflow,之后就可以调用run来执行这个Workflow了。需要注意的是,connect函数有两种形式,分别是:
connect(source, "source_output", dest, "dest_input")
connect([(source, dest, [("source_output1", "dest_input1"),
("source_output2", "dest_input2")
])
])
3. Workflow可视化
我们都知道,人对于抽象的东西并不十分敏感,其中最有名的莫过于安斯库姆四重奏。在编程过程中也是一样的,对于简单的处理程序,我们很容易看出其中的问题,但是对于特别复杂的处理流程,要想一眼看出其中的问题就没有那么容易了,为了帮助用户看出Workflow中的各种各样的问题,Nipype提供了一种可视化的函数作为工具——write_graph。其基本用法十分简单:
wf.write_graph(dotfilename = 'workflow_graph.dot')
from IPython.display import Image
Image(filename="/output/working_dir/smoothflow/workflow_graph.png")
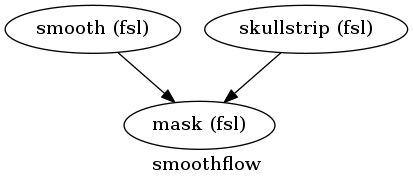
执行上述命令后,就会生成一个不是十分漂亮的图形:
上面的图片只是最最基础的显示方式,write_graph共提供了五种图片模式,分别是:
- orig 默认的显示方式,只会显示最顶层的Workflow,不会显示嵌套Workflow的内部结构,默认的就是这种形式
- flat 在绘图时,会展开Workflow的内部结构
- hierarchical 在绘图时,同样会展开Workflow的内部结构。相比于flat方式,该方式会更加清楚的标记出哪些是嵌套Workflow的内部结构
- colored 该方式会在hierarchical的基础上添加颜色信息
- exec 这一种我也没有理解,如果大家能够理解的话希望能够给与指点,在此附上英文原文This visualization is the most different from the rest. Like the flat visualization, it depicts all individual nodes. But additionally, it drops the utility nodes from the workflow and expands workflows to depict iterables (can be seen in the detailed_graph visualization further down below).
在使用时,只需要对write_graph的graph2use进行指定即可。
下面我们用实际的图片来演示一下几种绘图模式之间的区别:
使用上述方法,仅仅能够看到节点之间的连接关系。事实上,在调用write_graph的时候,如果模式为 orig、flat或exec,系统已经帮我们生成了更加细粒度的图片,如果要查看的话,只要在文件名后添加detailed即可,例如在执行如下代码
Image(filename="graph_flat_detailed.png")
就能看到这样的图片
目前写作的电脑上没有安装相应的环境,因此都是从官方网站上抓取的图片,代码也没有经过测试。
未完待续...