声明:本文用到的clusterProfiler, DOSE, enrichplot均是Y叔的作品,本人只是辛勤的搬运工,如想学习更多,请扫下方二维码,关注Y叔的公众号。生信路上自学愉快~
Step 1 输入数据
输入:
实验组:敲出掉某基因的基因symbol及FPKM值
对照组:没有敲出掉某基因的基因symbol及FPKM值
##读入数据
setwd("/Users/XXX/desktop")
d1=read.table("knockdown.txt",sep="\t",header=T)
d2=read.csv("control.csv",sep=",",header=T)
##将第一列(symbol)变为向量型(原来是factor)
d1[,1]<-as.character(d1[,1])
d2[,1]<-as.character(d2[,1])
Step 2 整理数据
1. 首先要将实验组和对照组中相同的基因symbol及表达值整理到一起
d3<-merge(d1,d2,by.x="Symbol",by.y = "Symbol")
##计算log2-foldchange值
d3$FC_value<-log(d3[,3]/d3[,2])
2. 我们有的是symbol,需要转化成entrezID
library(clusterProfiler)
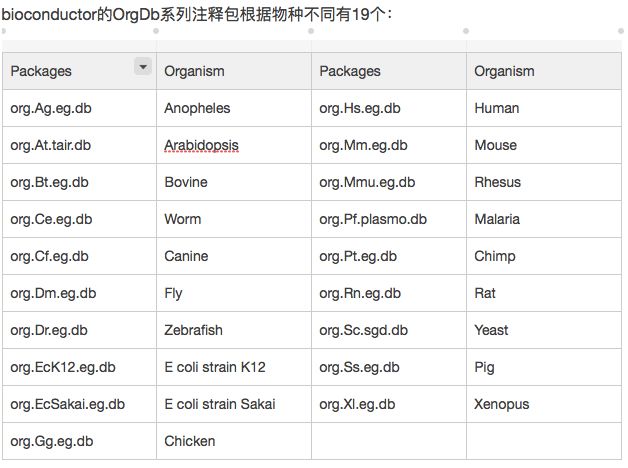
##数据是小鼠的,所以用的是org.Mm.eg.db数据库
anno<-bitr(d3$Symbol, fromType = "SYMBOL", toType = "ENTREZID",OrgDb = "org.Mm.eg.db")
##整理注释过的entrezID号
d4<-merge(d3,anno,by.x="Symbol",by.y = "SYMBOL")
d5<-d4[,-c(1:3)]
根据自己的物种选择数据库和缩写名
3. 整理genelist, 用于GSEA分析
geneList<-d5[,1]
names(geneList)<-d5[,2]
geneList<-sort(geneList,decreasing = T)
如果想更详细了解整理genelist的原理,请移步https://mp.weixin.qq.com/s/aht5fQ10nH_07CYttKFH7Q
Step 3 做GSEA
GSEA分两大类:
- KEGG(通路)
- GO(网络)
GO又分三类
- BP biological process
- MF molecular function
- CC cellular component
分别给出每一种的代码:
1. KEGG
require(enrichplot)
kk <- gseKEGG(geneList, organism = "mmu")
2. GO
- BP
kk <- gseGO(geneList,ont = "BP" OrgDb = org.Mm.eg.db)
- MF
kk <- gseGO(geneList,ont = "MF" OrgDb = org.Mm.eg.db)
- CC
kk <- gseGO(geneList,ont = "CC" OrgDb = org.Mm.eg.db)
按照得分(enrichment score)排列并保存
sortkk<-kk[order(kk$enrichmentScore,decreasing=T)]
Step 4 可视化
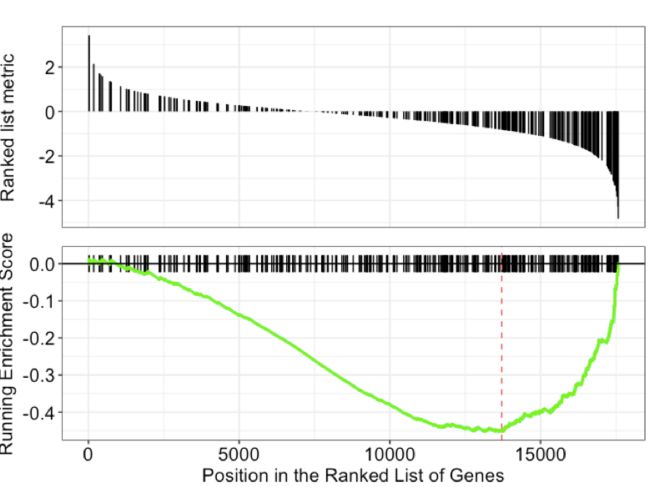
方法一:
library(enrichplot)
gseaplot(kk, "mmu05165")
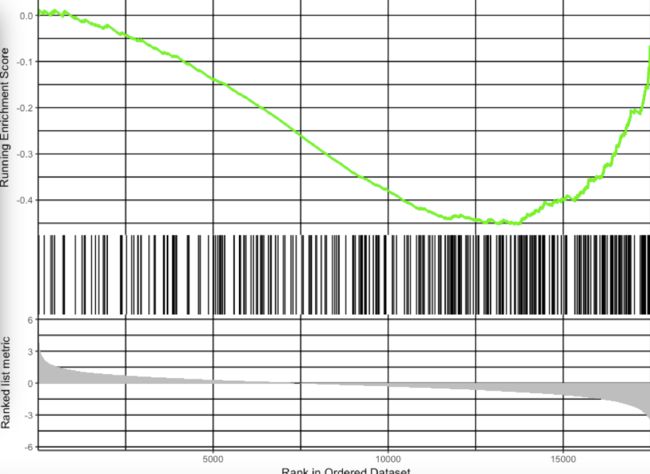
方法二:
gseaplot2(kk, "mmu05165")
方法三:
### 将ES排名前4的画在一张图上
gseaplot2(kk, row.names(sortkk)[1:4], pvalue_table = TRUE)
老规矩:想要数据集的话,点喜欢,然后私信我就可以了。