万事开头难,从前都是碎片化的学习生信,一些基本的概念还是能粗略的理解,但知识始终无法结构化,更别说在公开的场合写下文字记录自己的学习心得。这次在徐班长的带领下,终于鼓起勇气记录下学习过程。以下是我对这次作业浅显的解答,望各位达人斧正。
学习的过程本身就是解决问题的过程,那么重点在于“过程”如何实现,在于如何解决学习过程中遇到的问题。接触电脑开始,当我遇到问题的时候,我相信我不会是第一个遇到这个问题的人,总有人遇到了相同或者类似的问题,那么就要求我们如何更好的掌握搜索引擎这把利器。当然,如果搜索不到你想要的答案还可以到一些热门的论坛进行提问,其实我知道的也就是生信技能术和Biostar。
生物学基础
DNA
定义:它是一种携带生物体的发育,功能和繁殖所需的遗传指令的大分子,真核生物中,其存在于细胞核;原核生物中,它存在于细胞浆。
核苷酸:A、T、C、G;其配对原则为A-T,C-G;
组成:由两股小分子相互缠绕组成的双螺旋结构。
forward strain(+ortop strainorwaston strain)(正向链)和reverse strain(-orbottom strainorcirck strain)(反向链)仅是一种标签,并不是由DNA的内在属性所决定的,这是在研究者研究某DNA时就定义好的,虽然是该决定是随意的,但必须保持前后一致。
DNA链是具有方向性的,sense strain/antisense strain与forward strain/reverse strain是完全不同的概念,后者体现的序列方向性。DNA的转录是不对称的,基因转录只能以双链DNA分子中的一条链作为模板,其中与mRNA具有相同序列的DNA单链称为sense strain,作为转录模板的单链称为模板链,也叫antisense strain。
RNA
RNA与DNA的主要区别:RNA含核糖,DNA含脱氧核糖;RNA的碱基 A-U、C-G;
RNA的种类:rRNA、mRNA、tRNA、hnRNA、lncRNA、snRNA、SiRNA等。
RNA降解速度快
DNA测序
是指获得DNA序列的技术。获得原始文件格式为FASTA、FASTQ、uBAM
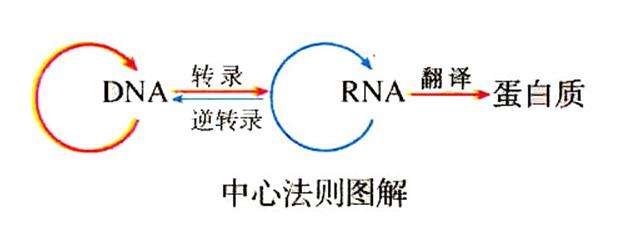
中心法则
CpG islands
在人类基因组内,GC的含量大约为40%;这些GC并不是平均分布在基因组内,在某些DNA片段上其含量可高达60%以上,而在另一些区域则只有33%左右。这种GC含量的差别,在基因表达的调控和基因突变上都可能扮演着重要的角色。例如,在基因的末端和启动子区域通常存在一些富含双核苷酸“CG”的区域,称为“CpG岛”(CpG island),这些CpG岛不仅是基因的一种标志,而且还参与基因表达的调控和影响染色质的结构通常这些位点很容易发生甲基化。正常细胞的CpG岛由于被保护而处于非甲基化状态.全基因组低甲基化,维持甲基化模式酶的调节失控和正常非甲基化CpG岛的高甲基化是人类肿瘤中普遍存在的现象. 以往的研究证明启动子区的高甲基化导致抑癌基因失活是人类肿瘤所具有的共同特征之一,而且这种高甲基化是导致抑癌基因失活的又一个机制.
来源:http://www.dxy.cn/bbs/topic/1487479
启动子(promoter)
又称启动基因,是DNA分子可与RNA聚合酶特异结合,并使转录起始地一段DNA序列,本身不被转录。
增强子(enhancer)
沉默子(silencer)
SNV
单核苷酸位点变异(single nucleotide variants, SNV)。个体间基因组DNA序列同一位置单个核苷酸变异(替代、插入或缺失)所引起的多态性。
InDel
(insertion-deletion) 插入缺失标记,指的是两种亲本中在全基因组中的差异,相对另一个亲本而言,其中一个亲本的基因组中有一定数量的核苷酸插入或缺失(Jander et al., 2002)。根据基因组中插入缺失位点,设计一些扩增这些插入缺失位点的PCR 引物,这就是InDel标记。
CNV
拷贝数目变异也称拷贝数目多态,是一种长度大于1kb的DNA片段的变异,在人类基因组中广泛分布,CNV位点的突变率远高于SNP(Single nucleotide polymorphism),是人类疾病的重要致病因素之一。
SV
Structure Variation(染色体)结构变异。包括插入、缺失、倒位、易位及重复等,长度在1kb~3Mb。
同源性
Ubuntu16.04LTS安装
网上教程很多
安装后第一步
sudo apt-get update
sudo apt-get upgrade -y
安装所需的库文件
sudo apt-get install -y curl unzip build-essential ncurses-dev
sudo apt-get install -y byacc zlib1g-dev python-dev git cmake
sudo apt-get install -y default-jdk ant
Miniconda的安装
'''
wget https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash Miniconda3-latest-Linux-x86_64.sh
'''
可用conda -h了解conda命令的用法。
之前徐班长在微信公众号上有过添加国内镜像的代码,直接拿过来
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/msys2/
conda config --add channels bioconda
conda config --add channels r
conda config --set show_channel_urls yes
不过在今年的8月22日,bioconda已经有国内镜像了
第四行可以改为:
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
创建名为bioinfo的环境
conda create -y --name bioinfo python=3
激活bioinfo环境
source activate bioinfo
为bioinfo安装书上所需的生信软件,作者将软件列表存在http://data.biostarhandbook.com/install/conda.txt
(entrez-direct bwa htslib bowtie2 emboss bedtools samtools bamtools bwa sra-tools cutadapt seqtk datamash bcftools freebayes subread bioawk hisat2 bbmap trimmomatic fastqc snpeff picard blast perl-list-moreutils)
curl http://data.biostarhandbook.com/install/conda.txt | xargs conda install -y
数据的可重复性分析
目前没有正在开始一个项目,暂时也不具备这种能力,但我想如想让自己分析的项目让别人重复出来必须遵循一些基本原则:
统一的分析思路
详细数据的处理方法说明
关于系统版本、配置、所用软件的版本的详细说明。