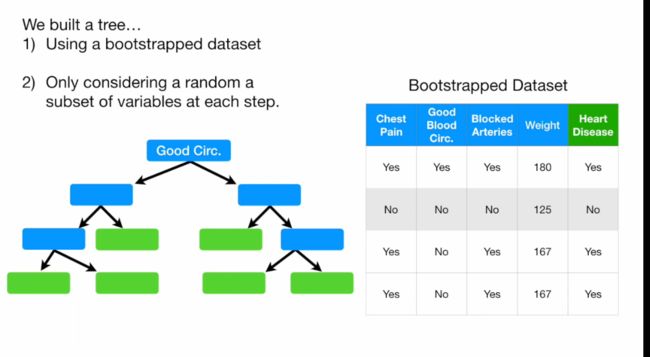
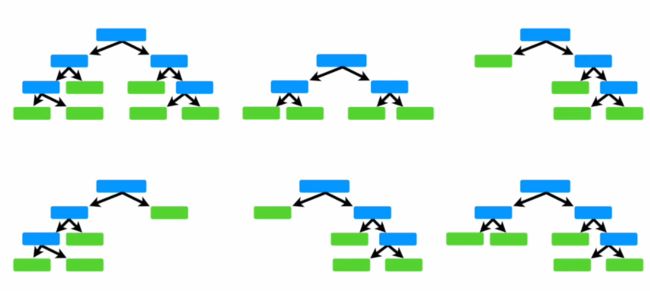
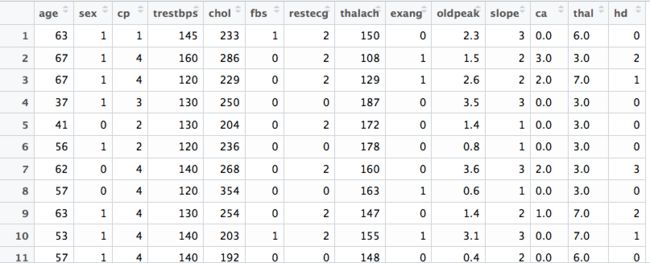
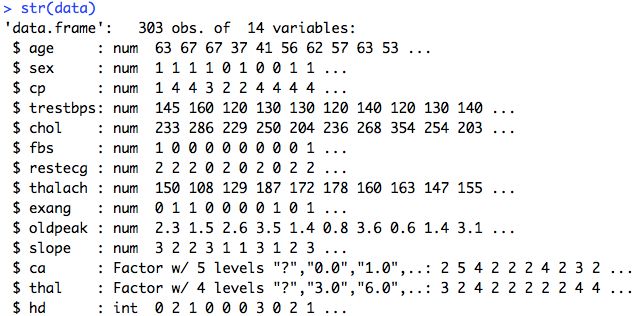
关于随机森林,你要知道:
- 基于决策树,比决策树更加稳定的分类器
- 无监督,随机抽取组成小样本。再放回,再抽取,即bootstrap
- 建树足够多,没用的树就会被抵消。树越多,模型精确度越高,增加到一定程度后变缓
- 可能会过拟合。当样本数量小于变量数量时,会发生过拟合,即用一个方程,求解两个未知数。
构建过程:
第一步 构建bootstrap
- 从原来数据集中随机挑选样本构建新的数据集,同一个样本可以挑选两次
第二步 建立决策树
- 用bootstrap的数据集构建决策树
- 但是每构建一步决策树,只能用bootstrap数据集变量的子集
-
如图中,表里显示有五个变量,但每次构建决策树,只能用<=5个变量。
第三步 重复这个过程若干次
-
这就是若干个决策树,随机森林让结果更加准确
问题:
- 原数据集中有1/3的数据在bootstrap中,没有落入新产生的数据集,这部分称为 out-of-bag dataset
- 这一部分out-of-bag dataset容易产生分类错误,我们称为out-of-bag error
代码实战
1. 获取数据。我们选择UCI上的machine learning的数据集
library(ggplot2)
library(cowplot)
library(randomForest)
url <- "http://archive.ics.uci.edu/ml/machine-learning-databases/heart-disease/processed.cleveland.data"
data <- read.csv(url, header=FALSE)
colnames(data) <- c(
"age",
"sex",# 0 = female, 1 = male
"cp", # chest pain
# 1 = typical angina,
# 2 = atypical angina,
# 3 = non-anginal pain,
# 4 = asymptomatic
"trestbps", # resting blood pressure (in mm Hg)
"chol", # serum cholestoral in mg/dl
"fbs", # fasting blood sugar greater than 120 mg/dl, 1 = TRUE, 0 = FALSE
"restecg", # resting electrocardiographic results
# 1 = normal
# 2 = having ST-T wave abnormality
# 3 = showing probable or definite left ventricular hypertrophy
"thalach", # maximum heart rate achieved
"exang", # exercise induced angina, 1 = yes, 0 = no
"oldpeak", # ST depression induced by exercise relative to rest
"slope", # the slope of the peak exercise ST segment
# 1 = upsloping
# 2 = flat
# 3 = downsloping
"ca", # number of major vessels (0-3) colored by fluoroscopy
"thal", # this is short of thalium heart scan
# 3 = normal (no cold spots)
# 6 = fixed defect (cold spots during rest and exercise)
# 7 = reversible defect (when cold spots only appear during exercise)
"hd" # (the predicted attribute) - diagnosis of heart disease
# 0 if less than or equal to 50% diameter narrowing
# 1 if greater than 50% diameter narrowing
)
head(data)
2. 清洗数据
str(data)
发现,sex, cp, fbs, restecg等明明是factor类型,但这里给的是num。另外有些地方填充的是?,需要改成NA
## First, replace "?"s with NAs.
data[data == "?"] <- NA
## Now add factors for variables that are factors and clean up the factors
## that had missing data...
data[data$sex == 0,]$sex <- "F"
data[data$sex == 1,]$sex <- "M"
data$sex <- as.factor(data$sex)
data$cp <- as.factor(data$cp)
data$fbs <- as.factor(data$fbs)
data$restecg <- as.factor(data$restecg)
data$exang <- as.factor(data$exang)
data$slope <- as.factor(data$slope)
data$ca <- as.integer(data$ca)
data$ca <- as.factor(data$ca) # ...then convert the integers to factor levels
data$thal <- as.integer(data$thal) # "thal" also had "?"s in it.
data$thal <- as.factor(data$thal)
## This next line replaces 0 and 1 with "Healthy" and "Unhealthy"
data$hd <- ifelse(test=data$hd == 0, yes="Healthy", no="Unhealthy")
data$hd <- as.factor(data$hd)
3. 构建模型
- 大多数的机器学习模型,需要人为把数据集划分成训练集和验证集,但是随机森林省去了这步,因为它用了bootstrap,不是所有的数据集都用来建树。
- 训练数据集是bootstrap数据,验证数据集是剩下的样本,又称为out-of-bag data (OOB)
a) 先用临近值填补缺失值
set.seed(43)
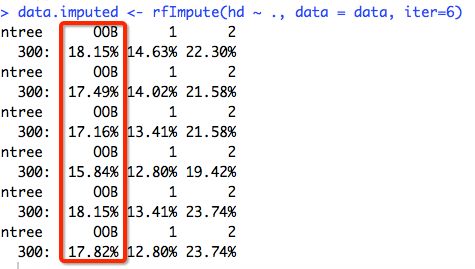
data.imputed<-rfImpute(hd~.,data=data,iter=6)
iter: 迭代数 breiman说4-6次就好,过多的迭代数不会让OOB error变小
set.seed:保证抽取的过程是随机的
hd~: 我们想通过所有参数预测hd
红框部分为每次迭代的OOB error
b) 构建随机森林模型
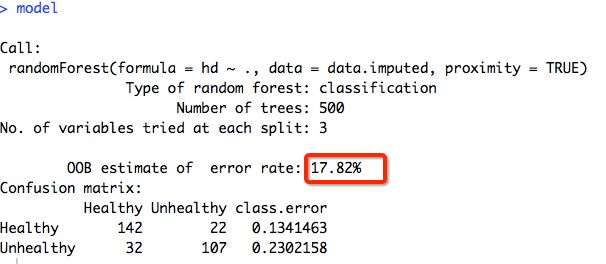
model <- randomForest(hd ~ ., data=data.imputed, proximity=TRUE)
mtry:
如果我们想预测的是连续变量,该值为总的变量值/3
如果想预测的是factor,该值为总变量数的根号
本例子中,hd是factor,mtry的默认值为sqrt(13)=3.6约等于3
number of tree:500 种树个数,默认500个
no. of variables tried at each split: 3 (即mtry)节点个数
OOB误差:17.82% 这个很重要
cofusion matrix的意义:
22个unhealthy被分入healthy中
32个healthy被分入unhealthy中
c) 更换mtry和number of trees的数量,使随机森林达到最优
核心思想:使OOB,healthy, unhealthy的error rate达到最低
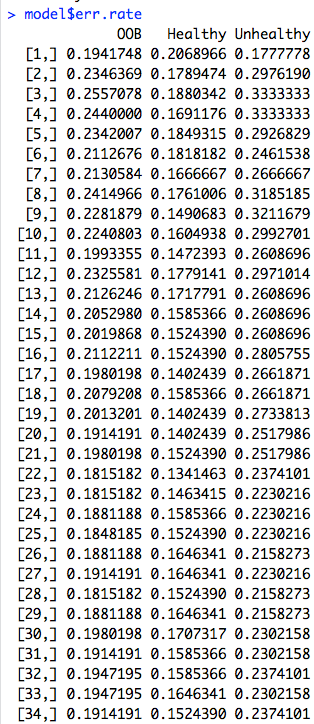
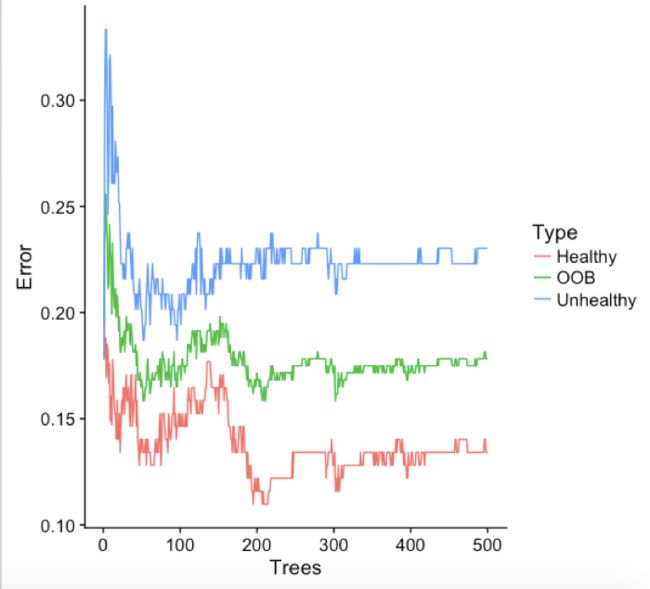
model$err.rate
横行:种的第i颗树,i=1:500,依次类推
纵行:OOB error rate; healthy error rate; unhealthy error rate
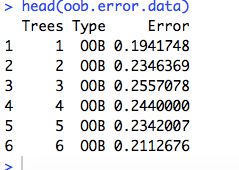
- 整理出矩阵,画图
oob.error.data <- data.frame(
Trees=rep(1:nrow(model$err.rate), times=3),
Type=rep(c("OOB", "Healthy", "Unhealthy"), each=nrow(model$err.rate)),
Error=c(model$err.rate[,"OOB"],
model$err.rate[,"Healthy"],
model$err.rate[,"Unhealthy"]))
ggplot(data=oob.error.data, aes(x=Trees, y=Error)) +
geom_line(aes(color=Type))
可以看出:当树种到400以后,三者的误差基本不变了
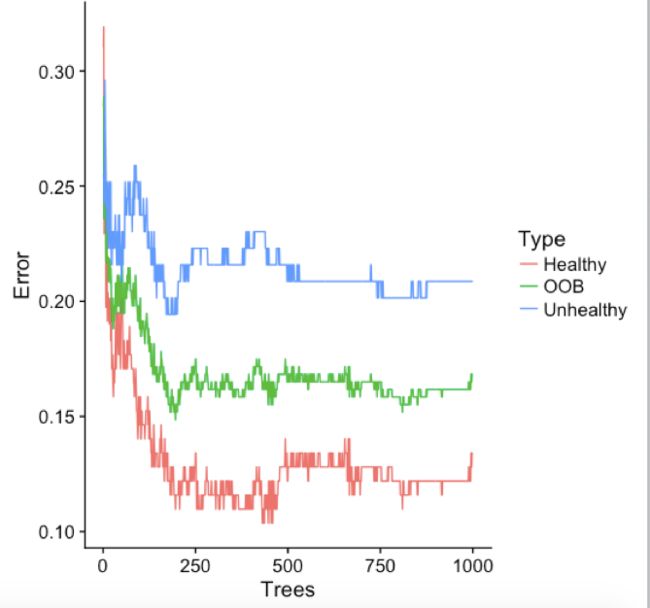
- 那么种1000颗树会怎样呢?
model <- randomForest(hd ~ ., data=data.imputed, ntree=1000, proximity=TRUE)
model
oob.error.data <- data.frame(
Trees=rep(1:nrow(model$err.rate), times=3),
Type=rep(c("OOB", "Healthy", "Unhealthy"), each=nrow(model$err.rate)),
Error=c(model$err.rate[,"OOB"],
model$err.rate[,"Healthy"],
model$err.rate[,"Unhealthy"]))
ggplot(data=oob.error.data, aes(x=Trees, y=Error)) +
geom_line(aes(color=Type))
500-1000之间,误差基本不变,因此选500颗树就好
- 更换mtry值,找到误差最低的mtry值
oob.values <- vector(length=10)
for(i in 1:10) {
temp.model <- randomForest(hd ~ ., data=data.imputed, mtry=i, ntree=1000)
oob.values[i] <- temp.model$err.rate[nrow(temp.model$err.rate),1]
}
oob.value
可以看出, mtry在3左右就很好,再低容易引起过拟合
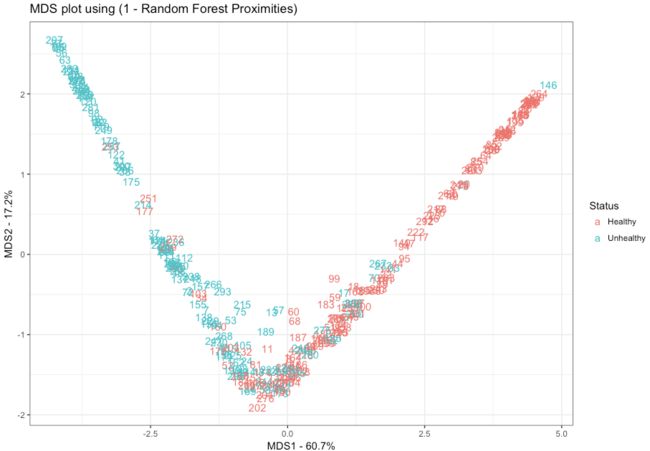
4. 应用多维尺度变换(MDS)查看样本之间的距离,即拟合效果
具体原理参照我之前的帖子
- 将proximity matrix转化成distance matrix
distance.matrix <- dist(1-model$proximity)
mds.stuff <- cmdscale(distance.matrix, eig=TRUE, x.ret=TRUE)
- 计算每个MDS轴占据的百分比
mds.var.per <- round(mds.stuff$eig/sum(mds.stuff$eig)*100, 1)
- 画图
mds.values <- mds.stuff$points
mds.data <- data.frame(Sample=rownames(mds.values),
X=mds.values[,1],
Y=mds.values[,2],
Status=data.imputed$hd)
ggplot(data=mds.data, aes(x=X, y=Y, label=Sample)) +
geom_text(aes(color=Status)) +
theme_bw() +
xlab(paste("MDS1 - ", mds.var.per[1], "%", sep="")) +
ylab(paste("MDS2 - ", mds.var.per[2], "%", sep="")) +
ggtitle("MDS plot using (1 - Random Forest Proximities)")
可见我们的随机森林效果不错, healthy分成一类,unhealthy分成一类
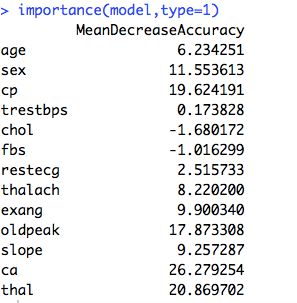
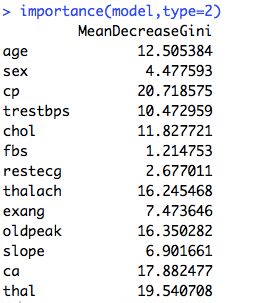
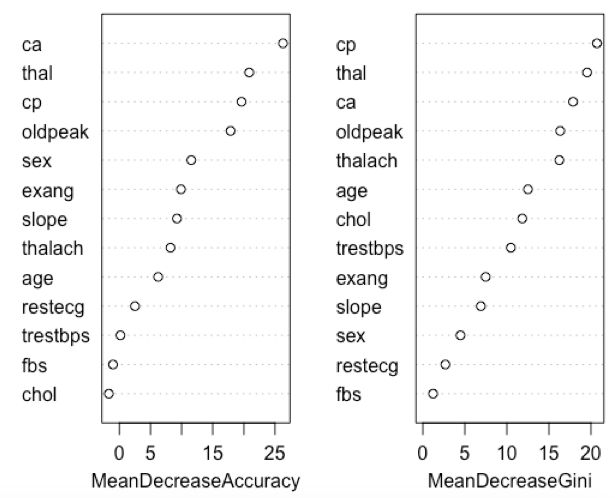
5. 查看每个变量的重要性评分
注意:importance=TRUE必须得打开,否则没法进行重要性评分
model <- randomForest(hd ~ ., data=data.imputed, proximity=TRUE,importance=TRUE)
importance(model,type=1)
importance(model,type=2)
varImpPlot(model)
- type=1 重要性评分 袋外数据自变量值发生轻微扰动后的分类正确率与扰动前分类正确率的平均减少量。即计算精度下降,平均精度的减少值。越大说明这个变量越重要
- type=2 gini指数 节点纯度,越大纯度越低