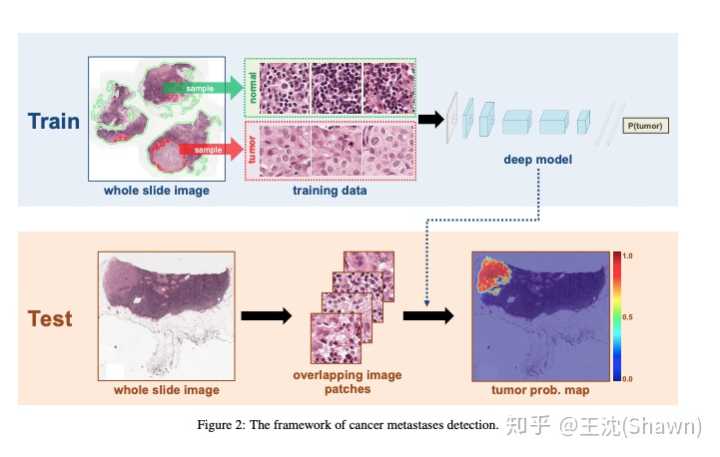
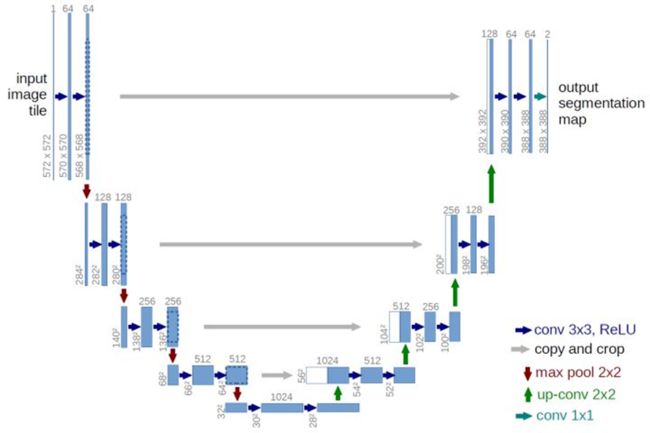
- 探秘3D UNet-PyTorch:高效三维图像分割利器
鲍凯印Fox
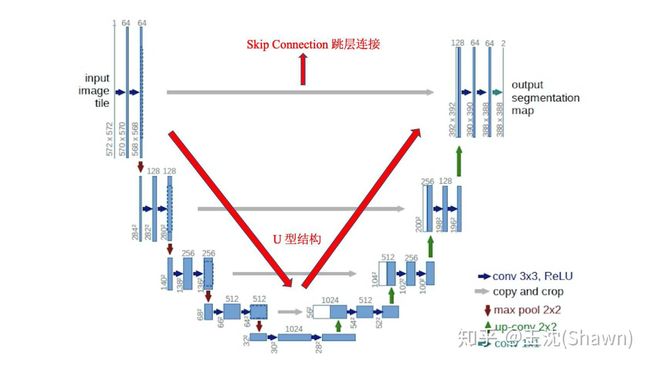
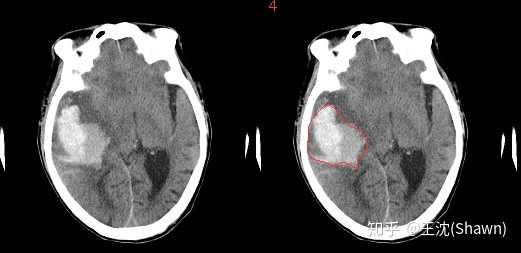
探秘3DUNet-PyTorch:高效三维图像分割利器在医学影像处理、计算机视觉和自动驾驶等领域,三维图像的理解与分析至关重要。而是一个基于PyTorch实现的深度学习模型,专为三维图像分割任务设计。本文将深入剖析该项目的技术细节,应用场景及特性,以期吸引更多的开发者和研究人员参与其中。项目简介3DUNet是2DUNet的三维扩展,其结构保持了卷积神经网络的对称性,采用跳跃连接的方式保留了不同尺度
- 使用3DUNet训练自己的数据集(pytorch)— 医疗影像分割
编程日记✧
智能医疗pytorch人工智能python计算机视觉图像处理深度学习健康医疗
代码:lee-zq/3DUNet-Pytorch:3DUNetimplementedwithpytorch(github.com)文章<cicek16miccai.pdf(uni-freiburg.de)3DU-Net:LearningDenseVolumetricSegmentation
- 中文车牌识别系统 `End-to-end-for-Chinese-Plate-Recognition` 教程
皮静滢Annette
中文车牌识别系统End-to-end-for-Chinese-Plate-Recognition教程End-to-end-for-chinese-plate-recognition基于u-net,cv2以及cnn的中文车牌定位,矫正和端到端识别软件,其中unet和cv2用于车牌定位和矫正,cnn进行车牌识别,unet和cnn都是基于tensorflow的keras实现项目地址:https://gi
- 【论文阅读33】Deep learning optoacoustic tomography with sparse data
弹伦琴的雷登
【论文阅读系列】人工智能深度学习论文阅读图像处理
Deeplearningoptoacoustictomographywithsparsedata论文题目:基于稀疏数据的深度学习光声断层扫描论文链接:Deeplearningoptoacoustictomographywithsparsedata|NatureMachineIntelligence代码链接:GitHub-ndavoudi/sparse_artefact_unet数据链接:Data发
- unet各模块内容的理解(包含注意力机制、残差、以及数据维度的变化)
云梦之上
#扩散模型系统性学习人工智能神经网络pytorch
文章目录attention机制Unet的各个模块的设计①残差块②下块做一次残差,做一次自注意力③上块:这里做了skipconnect,做一次残差,做一次注意力④中块:做两次残差和一次自注意力⑤上采样:通道数不变,长宽翻两倍⑥下采样:通道数不变,长宽缩小到原来的一半整个unet模块unet模块的示意图参考的unet代码unet代码attention机制参考内容:超详细图解Self-Attention
- Unet改进10:在不同位置添加CPCA||通道先验卷积注意力机制
AICurator
Unet改进专栏深度学习神经网络unet语义分割
本文内容:在不同位置添加CPCA注意力机制目录论文简介1.步骤一2.步骤二3.步骤三4.步骤四论文简介低对比度和显著的器官形状变化等特征经常出现在医学图像中。现有注意机制的自适应能力普遍不足,限制了医学影像分割性能的提高。本文提出了一种有效的通道先验卷积注意(CPCA)方法,该方法支持通道和空间维度上注意权重的动态分布。通过采用多尺度深度卷积模块,有效地提取空间关系,同时保留先验通道。CPCA具有
- 图像算法实习生--面经1
小豆包的小朋友0217
算法
系列文章目录文章目录系列文章目录前言一、为什么torch里面要用optimizer.zero_grad()进行梯度置0二、Unet神经网络为什么会在医学图像分割表现好?三、transformer相关问题四、介绍一下胶囊网络的动态路由五、yolo系列出到v9了,介绍一下你最熟悉的yolo算法六、一阶段目标检测算法和二阶段目标检测算法有什么区别?七、讲一下剪枝八、讲一下PTQandQAT量化的区别九、
- ubuntu 14.04.4 install
xuyss
ubuntuubuntu14.04.4
Tool:unetbootin-windows-608.exehttps://unetbootin.github.io/ISO:ubuntu-14.04.4-server-amd64.isohttp://mirrors.yun-idc.com/ubuntu-releases/14.04.4/FAQ:Whenyougettheerror,Alt+F2toasecondconsole.Findoutt
- 千卡利用率超98%,详解JuiceFS在权威AI测试中的实现策略
机器学习人工智能运维
2023年9月,AI领域的权威基准评测MLPerf推出了StorageBenchmark。该基准测试通过模拟机器学习I/O负载的方法,在不需要GPU的情况下就能进行大规模的性能压测,用以评估存储系统的在AI模型训练场景的适用性。目前支持两种模型训练:BERT(自然语言模型)和Unet3D(3D医学成像)。虽然目前不支持大语言模型如GPT、LLaMA,但BERT与大语言模型同为多层transform
- Stable Diffusion算法、结构全流程概述
lanlinbuaa
stablediffusionpython
StableDiffusion能力强、功能多、插件广,本文拟概述SD的全流程,方便梳理算法各结构的关系SD发展的重点论文DenoisingDiffusionProbabilisticModels(首次提出去噪扩散模型DDPM)DiffusionModelsBeatGANsonImageSynthesis(OpenAI改进UNet,DM超越GAN,ClassifierGuidance)High-Re
- 检查两个文件是否一样,可以通过比较两个文件的md5值
早起早起早起up
1.计算文件的md5,SHA1查看传输过程中有没有修改检查两个文件是否一样,可以通过比较两个文件的md5值(后续可以用这个方法来检验kevin.sql文件是否被修改)。(1).windows查看方式certutil-hashfileD:\BaiduNetdiskDownload\coding-265-master.zipMD5压缩文件查看(2)linux方式catkevin.sql.md5
- Unet 高阶分割网络实战、多类别分割、迁移学习(deeplab、resnet101等等)
听风吹等浪起
图像分割计算机视觉人工智能
1、前言Unet图像分割之前介绍了不少,具体可以参考图像分割专栏为了实现多类别的自适应分割,前段时间利用numpy的unique函数实现了一个项目。通过numpy函数将mask的灰度值提取出来,保存在txt文本里,这样txt里面就会有类似012...等等的灰度值。而有几个灰度值,就代表分割要分出几个类别。具体可以参考:Unet实战分割项目、多尺度训练、多类别分割将vgg换成resnet的unet参
- 对视频进行分块,断点续传
爱笑的人、
java
分块测试//分块测试@TestpublicvoidtestChunk()throwsIOException{//源路径FilesourceFile=newFile("D:\\BaiduNetdiskDownload\\Day1-00.项目导学.mp4");//分块文件存储路径StringchunkFilePath="D:\\develop\\chunk\\";//分块文件大小intchunkSiz
- OpenAI Sora视频模型技术原理报告解读
AI周红伟
人工智能sora技术原理Sora技术原理
▌01.OpenAISora视频生成模型技术报告总结•不管是在视频的保真度、长度、稳定性、一致性、分辨率、文字理解等方面。•技术细节写得比较泛(防止别人模仿)大概就是用视觉块编码(visualpatch)的方式,把不同格式的视频统一编码成了用transformer架构能够训练的embeding,然后引入类似diffusion的unet的方式做在降维和升维的过程中做加噪和去噪,然后把模型做得足够大,
- OpenAI Sora视频生成模型技术报告中英全文+总结+影响分析
龙腾亚太
音视频人工智能
01.OpenAISora视频生成模型技术报告总结不管是在视频的保真度、长度、稳定性、一致性、分辨率、文字理解等方面,Sora都做到了SOTA(当前最优)。技术细节写得比较泛(防止别人模仿)大概就是用视觉块编码(visualpatch)的方式,把不同格式的视频统一编码成了用transformer架构能够训练的embeding,然后引入类似diffusion的unet的方式做在降维和升维的过程中做加
- Sora专辑|OpenAI Sora视频生成模型技术报告中英全文+总结+影响分析
明矛顿了
音视频人工智能chatgpt
▌01.OpenAISora视频生成模型技术报告总结•不管是在视频的保真度、长度、稳定性、一致性、分辨率、文字理解等方面,Sora都做到了SOTA(当前最优)。•技术细节写得比较泛(防止别人模仿)大概就是用视觉块编码(visualpatch)的方式,把不同格式的视频统一编码成了用transformer架构能够训练的embeding,然后引入类似diffusion的unet的方式做在降维和升维的过程
- AcuAutomate:一款基于Acunetix的大规模自动化渗透测试与漏洞扫描工具
FreeBuf_
自动化elasticsearch运维
关于AcuAutomateAcuAutomate是一款基于Acunetix的大规模自动化渗透测试与漏洞扫描工具,该工具旨在辅助研究人员执行大规模的渗透测试任务。在大规模的安全测试活动中,AcuAutomate可以帮助我们同时启动或停止多个Acunetix扫描任务。除此之外,我们还可以将其功能无缝集成到枚举封装器或One-Liner中,并通过管道功能实现更高效的控制。工具安装由于该工具基于Pytho
- OpenAI视频生成模型Sora背后的技术及其深远的影响
知来者逆
SoraSora文字生成视频视频生成OpenAI
前言Sora的视频生成技术在保真度、长度、稳定性、一致性、分辨率和文字理解等方面都达到了当前最优水平。其核心技术包括使用视觉块编码将不同格式的视频统一编码成Transformer可训练的嵌入向量,以及类似于扩散过程的UNet方法进行降维和升维的加噪与去噪操作。通过构建足够大的模型,使其具备了智能的涌现能力,例如在一定程度上理解真实世界的物理影响和因果关系。与其他视频生成模型不同,OpenAI采用了
- Mamba-UNet:用于医学图像分割的类似UNet的纯视觉Mamba网络
AI浩
高质量人类CV论文翻译深度学习人工智能计算机视觉
摘要在医学图像分析的最新进展中,卷积神经网络(CNN)和视觉转换器(ViT)都取得了显著的基准成绩。前者通过其卷积操作在捕获局部特征方面表现出色,而后者则通过利用自注意力机制实现了出色的全局上下文理解。然而,这两种架构在有效建模医学图像中的长距离依赖关系时都存在局限,这对于精确分割至关重要。受到Mamba架构的启发,该架构因其处理长序列和全局上下文信息的能力以及作为国家空间模型(SSM)的增强计算
- 机器学习案例3:从科学论文图片中提取标题、作者和摘要
suoge223
机器学习实用指南人工智能
在这个项目中,我的目标是从科学论文图片中提取某些部分(标题、作者和摘要)。预期提取部分是科学论文中常见的部分,例如标题、摘要和作者。输入与最终结果。我的输入是将第一页纸转换成图像。最终结果是一个txt文件,其中包含标题、作者和摘要部分,如下图1和图2所示。我将使用UNet来了解在哪里可以找到这些部分,然后将训练学到的信息传递到OCR中。完整的项目可以在这里找到。图1要提取的论文首页(图片格式)图2
- 跟《世界报》Le monde学法语新闻2020-6-2 l’exode des villes vers les campagnes
木星外语
对应音频讲解,可前往木星外语网站:http://muxingwaiyu.com«Opposervilleetcampagnenenousferapasavancer»(1)Dansunetribuneau«Monde»,MagaliTalandier,professeureenétudesurbainesetrégionalesàl’universitéGrenoble-Alpes,décritl
- Go ahead
AngelaTong
Howmanytimesareyouhopeless?Howmanytimesareyouhelpless?And,howmanytimesareyoufearless?WhenIwasyoung,mymomaskedtheforunetelleraboutmyfaith"Thisgirlcouldn'tbetoohappy,otherwisebadluckwillcome"男儿命女儿身,劳苦坎坷
- Unet 实战分割项目、多尺度训练、多类别分割
听风吹等浪起
图像分割深度学习人工智能机器学习
1.介绍之前写了篇二值图像分割的项目,支持多尺度训练,网络采用backbone为vgg的unet网络。缺点就是没法实现多类别的分割,具体可以参考:二值图像分割统一项目本章只对增加的代码进行介绍,其余的参考上述链接博文本章实现的unet网络的多类别分割,也就是分割可以是两个类别,也可以是多个类别。训练过程仍然采用多尺度训练,即网络会随机将图片缩放到设定尺寸的0.5-1.5倍之间文件目录如下:2.实现
- Unet+ResNet 实战分割项目、多尺度训练、多类别分割
听风吹等浪起
图像分割人工智能计算机视觉
1.介绍传统的Unet网络,特征提取的backbone采用的是vgg模型,vgg的相关介绍和实战参考以前的博文:pytorch搭建VGG网络VGG的特征提取能力其实是不弱的,但网络较为臃肿,容易产生梯度消失或者梯度爆炸的问题。而Resnet可以解决这一问题,参考:ResNet训练CIFAR10数据集,并做图片分类本章在之前文章的基础上,只是将Unet的backbone进行替换,将vgg换成了res
- NansException: A tensor with all NaNs was produced in Unet. This could be either because there‘s not
mhack5200
python开发语言人工智能作画windows
最近下载个XL模型,用图生图时提示NansException:AtensorwithallNaNswasproducedinUnet.Thiscouldbeeitherbecausethere'snotenoughprecisiontorepresentthepicture,orbecauseyourvideocarddoesnotsupporthalftype,找了一堆方法,最终有个是成功,在次
- Stable Diffusion ubuntu 部署,问题记录
丿|一口亅皓桀
StableDiffusionstablediffusion
问题一在使用图生图时,报错NansException:AtensorwithallNaNswasproducedinUnet.Thiscouldbeeitherbecausethere'snotenoughprecisiontorepresentthepicture,orbecauseyourvideocarddoesnotsupporthalftype.Trysettingthe"Upcastc
- BaiduNetdiskPlugin Mac版百度网盘超级会员插件
产品U理
这次Mac毒为大家带来一款百度网盘客户端的超级会员插件,可以开启无限免费试用功能,速度杠杠的,需要的小伙伴可以试试,安装方法也很简单。由于本教程是基于百度网盘官方客户端而进行破解的,所以小伙伴们请务必先下载官方版的客户端哦,官方下载地址「https://pan.baidu.com/download」。安装教程1.下载并安装完成百度网盘客户端后,打开Mac的「终端」应用。不知道在哪里的可以按快捷键「
- 登录功能渗透测试
软件测试很重要
安全性测试
登录功能渗透测试登录功能的渗透测试是一种安全评估方法,用于检测Web应用程序中的登录系统是否存在安全漏洞。进行登录功能渗透测试时,测试者会尝试通过各种手段来模拟攻击者的行为,以发现和利用可能的安全弱点。以下是进行登录功能渗透测试时可以采取的几种常见方法:自动化扫描工具:使用AWVS(AcunetixWebVulnerabilityScanner)和Nessus等工具进行自动化扫描,这些工具可以帮助
- 公开 学生课堂行为数据集 SCB-Dataset 2 Student Classroom Behavior dataset
计算机视觉-杨帆
数据集学生行为
公开学生课堂行为数据集SCB-Dataset2StudentClassroomBehaviordatasetb站:https://www.bilibili.com/video/BV1cW4y197Ah/arxiv:https://arxiv.org/pdf/2306.03318.pdfgithub:https://github.com/Whiffe/SCB-dataset百度云:BaiduNetd
- Swin-UMamba:结合基于ImageNet的预训练和基于Mamba的UNet模型
AI浩
人工智能计算机视觉
摘要https://arxiv.org/pdf/2402.03302v1.pdf准确的医学图像分割需要整合从局部特征到全局依赖的多尺度信息。然而,现有方法在建模长距离全局信息方面面临挑战,其中卷积神经网络(CNNs)受限于其局部感受野,而视觉转换器(ViTs)则受到其注意力机制高二次复杂度的困扰。最近,基于Mamba的模型因其在长序列建模中的出色能力而备受关注。多项研究表明,这些模型在各种任务中可
- 多线程编程之卫生间
周凡杨
java并发卫生间线程厕所
如大家所知,火车上车厢的卫生间很小,每次只能容纳一个人,一个车厢只有一个卫生间,这个卫生间会被多个人同时使用,在实际使用时,当一个人进入卫生间时则会把卫生间锁上,等出来时打开门,下一个人进去把门锁上,如果有一个人在卫生间内部则别人的人发现门是锁的则只能在外面等待。问题分析:首先问题中有两个实体,一个是人,一个是厕所,所以设计程序时就可以设计两个类。人是多数的,厕所只有一个(暂且模拟的是一个车厢)。
- How to Install GUI to Centos Minimal
sunjing
linuxInstallDesktopGUI
http://www.namhuy.net/475/how-to-install-gui-to-centos-minimal.html
I have centos 6.3 minimal running as web server. I’m looking to install gui to my server to vnc to my server. You can insta
- Shell 函数
daizj
shell函数
Shell 函数
linux shell 可以用户定义函数,然后在shell脚本中可以随便调用。
shell中函数的定义格式如下:
[function] funname [()]{
action;
[return int;]
}
说明:
1、可以带function fun() 定义,也可以直接fun() 定义,不带任何参数。
2、参数返回
- Linux服务器新手操作之一
周凡杨
Linux 简单 操作
1.whoami
当一个用户登录Linux系统之后,也许他想知道自己是发哪个用户登录的。
此时可以使用whoami命令。
[ecuser@HA5-DZ05 ~]$ whoami
e
- 浅谈Socket通信(一)
朱辉辉33
socket
在java中ServerSocket用于服务器端,用来监听端口。通过服务器监听,客户端发送请求,双方建立链接后才能通信。当服务器和客户端建立链接后,两边都会产生一个Socket实例,我们可以通过操作Socket来建立通信。
首先我建立一个ServerSocket对象。当然要导入java.net.ServerSocket包
ServerSock
- 关于框架的简单认识
西蜀石兰
框架
入职两个月多,依然是一个不会写代码的小白,每天的工作就是看代码,写wiki。
前端接触CSS、HTML、JS等语言,一直在用的CS模型,自然免不了数据库的链接及使用,真心涉及框架,项目中用到的BootStrap算一个吧,哦,JQuery只能算半个框架吧,我更觉得它是另外一种语言。
后台一直是纯Java代码,涉及的框架是Quzrtz和log4j。
都说学前端的要知道三大框架,目前node.
- You have an error in your SQL syntax; check the manual that corresponds to your
林鹤霄
You have an error in your SQL syntax; check the manual that corresponds to your MySQL server version for the right syntax to use near 'option,changed_ids ) values('0ac91f167f754c8cbac00e9e3dc372
- MySQL5.6的my.ini配置
aigo
mysql
注意:以下配置的服务器硬件是:8核16G内存
[client]
port=3306
[mysql]
default-character-set=utf8
[mysqld]
port=3306
basedir=D:/mysql-5.6.21-win
- mysql 全文模糊查找 便捷解决方案
alxw4616
mysql
mysql 全文模糊查找 便捷解决方案
2013/6/14 by 半仙
[email protected]
目的: 项目需求实现模糊查找.
原则: 查询不能超过 1秒.
问题: 目标表中有超过1千万条记录. 使用like '%str%' 进行模糊查询无法达到性能需求.
解决方案: 使用mysql全文索引.
1.全文索引 : MySQL支持全文索引和搜索功能。MySQL中的全文索
- 自定义数据结构 链表(单项 ,双向,环形)
百合不是茶
单项链表双向链表
链表与动态数组的实现方式差不多, 数组适合快速删除某个元素 链表则可以快速的保存数组并且可以是不连续的
单项链表;数据从第一个指向最后一个
实现代码:
//定义动态链表
clas
- threadLocal实例
bijian1013
javathreadjava多线程threadLocal
实例1:
package com.bijian.thread;
public class MyThread extends Thread {
private static ThreadLocal tl = new ThreadLocal() {
protected synchronized Object initialValue() {
return new Inte
- activemq安全设置—设置admin的用户名和密码
bijian1013
javaactivemq
ActiveMQ使用的是jetty服务器, 打开conf/jetty.xml文件,找到
<bean id="adminSecurityConstraint" class="org.eclipse.jetty.util.security.Constraint">
<p
- 【Java范型一】Java范型详解之范型集合和自定义范型类
bit1129
java
本文详细介绍Java的范型,写一篇关于范型的博客原因有两个,前几天要写个范型方法(返回值根据传入的类型而定),竟然想了半天,最后还是从网上找了个范型方法的写法;再者,前一段时间在看Gson, Gson这个JSON包的精华就在于对范型的优雅简单的处理,看它的源代码就比较迷糊,只其然不知其所以然。所以,还是花点时间系统的整理总结下范型吧。
范型内容
范型集合类
范型类
- 【HBase十二】HFile存储的是一个列族的数据
bit1129
hbase
在HBase中,每个HFile存储的是一个表中一个列族的数据,也就是说,当一个表中有多个列簇时,针对每个列簇插入数据,最后产生的数据是多个HFile,每个对应一个列族,通过如下操作验证
1. 建立一个有两个列族的表
create 'members','colfam1','colfam2'
2. 在members表中的colfam1中插入50*5
- Nginx 官方一个配置实例
ronin47
nginx 配置实例
user www www;
worker_processes 5;
error_log logs/error.log;
pid logs/nginx.pid;
worker_rlimit_nofile 8192;
events {
worker_connections 4096;}
http {
include conf/mim
- java-15.输入一颗二元查找树,将该树转换为它的镜像, 即在转换后的二元查找树中,左子树的结点都大于右子树的结点。 用递归和循环
bylijinnan
java
//use recursion
public static void mirrorHelp1(Node node){
if(node==null)return;
swapChild(node);
mirrorHelp1(node.getLeft());
mirrorHelp1(node.getRight());
}
//use no recursion bu
- 返回null还是empty
bylijinnan
javaapachespring编程
第一个问题,函数是应当返回null还是长度为0的数组(或集合)?
第二个问题,函数输入参数不当时,是异常还是返回null?
先看第一个问题
有两个约定我觉得应当遵守:
1.返回零长度的数组或集合而不是null(详见《Effective Java》)
理由就是,如果返回empty,就可以少了很多not-null判断:
List<Person> list
- [科技与项目]工作流厂商的战略机遇期
comsci
工作流
在新的战略平衡形成之前,这里有一个短暂的战略机遇期,只有大概最短6年,最长14年的时间,这段时间就好像我们森林里面的小动物,在秋天中,必须抓紧一切时间存储坚果一样,否则无法熬过漫长的冬季。。。。
在微软,甲骨文,谷歌,IBM,SONY
- 过度设计-举例
cuityang
过度设计
过度设计,需要更多设计时间和测试成本,如无必要,还是尽量简洁一些好。
未来的事情,比如 访问量,比如数据库的容量,比如是否需要改成分布式 都是无法预料的
再举一个例子,对闰年的判断逻辑:
1、 if($Year%4==0) return True; else return Fasle;
2、if ( ($Year%4==0 &am
- java进阶,《Java性能优化权威指南》试读
darkblue086
java性能优化
记得当年随意读了微软出版社的.NET 2.0应用程序调试,才发现调试器如此强大,应用程序开发调试其实真的简单了很多,不仅仅是因为里面介绍了很多调试器工具的使用,更是因为里面寻找问题并重现问题的思想让我震撼,时隔多年,Java已经如日中天,成为许多大型企业应用的首选,而今天,这本《Java性能优化权威指南》让我再次找到了这种感觉,从不经意的开发过程让我刮目相看,原来性能调优不是简单地看看热点在哪里,
- 网络学习笔记初识OSI七层模型与TCP协议
dcj3sjt126com
学习笔记
协议:在计算机网络中通信各方面所达成的、共同遵守和执行的一系列约定 计算机网络的体系结构:计算机网络的层次结构和各层协议的集合。 两类服务: 面向连接的服务通信双方在通信之前先建立某种状态,并在通信过程中维持这种状态的变化,同时为服务对象预先分配一定的资源。这种服务叫做面向连接的服务。 面向无连接的服务通信双方在通信前后不建立和维持状态,不为服务对象
- mac中用命令行运行mysql
dcj3sjt126com
mysqllinuxmac
参考这篇博客:http://www.cnblogs.com/macro-cheng/archive/2011/10/25/mysql-001.html 感觉workbench不好用(有点先入为主了)。
1,安装mysql
在mysql的官方网站下载 mysql 5.5.23 http://www.mysql.com/downloads/mysql/,根据我的机器的配置情况选择了64
- MongDB查询(1)——基本查询[五]
eksliang
mongodbmongodb 查询mongodb find
MongDB查询
转载请出自出处:http://eksliang.iteye.com/blog/2174452 一、find简介
MongoDB中使用find来进行查询。
API:如下
function ( query , fields , limit , skip, batchSize, options ){.....}
参数含义:
query:查询参数
fie
- base64,加密解密 经融加密,对接
y806839048
经融加密对接
String data0 = new String(Base64.encode(bo.getPaymentResult().getBytes(("GBK"))));
String data1 = new String(Base64.decode(data0.toCharArray()),"GBK");
// 注意编码格式,注意用于加密,解密的要是同
- JavaWeb之JSP概述
ihuning
javaweb
什么是JSP?为什么使用JSP?
JSP表示Java Server Page,即嵌有Java代码的HTML页面。使用JSP是因为在HTML中嵌入Java代码比在Java代码中拼接字符串更容易、更方便和更高效。
JSP起源
在很多动态网页中,绝大部分内容都是固定不变的,只有局部内容需要动态产生和改变。
如果使用Servl
- apple watch 指南
啸笑天
apple
1. 文档
WatchKit Programming Guide(中译在线版 By @CocoaChina) 译文 译者 原文 概览 - 开始为 Apple Watch 进行开发 @星夜暮晨 Overview - Developing for Apple Watch 概览 - 配置 Xcode 项目 - Overview - Configuring Yo
- java经典的基础题目
macroli
java编程
1.列举出 10个JAVA语言的优势 a:免费,开源,跨平台(平台独立性),简单易用,功能完善,面向对象,健壮性,多线程,结构中立,企业应用的成熟平台, 无线应用 2.列举出JAVA中10个面向对象编程的术语 a:包,类,接口,对象,属性,方法,构造器,继承,封装,多态,抽象,范型 3.列举出JAVA中6个比较常用的包 Java.lang;java.util;java.io;java.sql;ja
- 你所不知道神奇的js replace正则表达式
qiaolevip
每天进步一点点学习永无止境纵观千象regex
var v = 'C9CFBAA3CAD0';
console.log(v);
var arr = v.split('');
for (var i = 0; i < arr.length; i ++) {
if (i % 2 == 0) arr[i] = '%' + arr[i];
}
console.log(arr.join(''));
console.log(v.r
- [一起学Hive]之十五-分析Hive表和分区的统计信息(Statistics)
superlxw1234
hivehive分析表hive统计信息hive Statistics
关键字:Hive统计信息、分析Hive表、Hive Statistics
类似于Oracle的分析表,Hive中也提供了分析表和分区的功能,通过自动和手动分析Hive表,将Hive表的一些统计信息存储到元数据中。
表和分区的统计信息主要包括:行数、文件数、原始数据大小、所占存储大小、最后一次操作时间等;
14.1 新表的统计信息
对于一个新创建
- Spring Boot 1.2.5 发布
wiselyman
spring boot
Spring Boot 1.2.5已在7月2日发布,现在可以从spring的maven库和maven中心库下载。
这个版本是一个维护的发布版,主要是一些修复以及将Spring的依赖提升至4.1.7(包含重要的安全修复)。
官方建议所有的Spring Boot用户升级这个版本。
项目首页 | 源