文献阅读:深度学习模型利用生物医学文本上下文关系进行命名实体识别
文献阅读:利用深度学习模型在生物医学文本的上下文关系中进行命名实体识别
- 题目
- 1 背景
- 2 材料和方法
- 2.1 GRAM-CNN方法
- 2.1.1 嵌入方法
- 2.1.2 GRAM-CNN
- 2.1.3 CRF
- 2.2 实施细节
- 2.3 数据集
- 2.4 评价标准
- 3 结果
- 4 讨论
题目
GRAM-CNN: a deep learning approach with local context for named entity recognition in biomedical text
1 背景
生物医学命名实体识别要比一般命名实体识别问题更复杂,原因为:
1、已经发现的实体数以万计,且数量还在不断增加;
2、同一个实体有许多不同的名字;
3、许多实体名字都很长;
4、生物医学文本中句子都很长。
目前存在的方法有基于词典的方法,基于规则的方法,机器学习的方法,深度学习的方法。机器学习中表现最好的是CRF(条件随机场),但它的性能很大程度上依赖于特征的选择,如正字法、形态学、基于语言学、连接词和基于词典的特征。
文章中说到目前已有的将LSTM运用到NER的方法考虑整个句子的信息,而对于一些句子中包含了和实体识别无关信息的情况,这种方法有些多余,不如利用CNN只考虑上下文几个词的信息。(但我认为考虑整个句子是由道理的,很多医学信息及其关系分布得都很分散,有时候跨句存在)
文章中提出的GRAM-CNN方法,在输入中加入了基于语言学的特征(POS tags词类),使用到了n-gram方法。
2 材料和方法
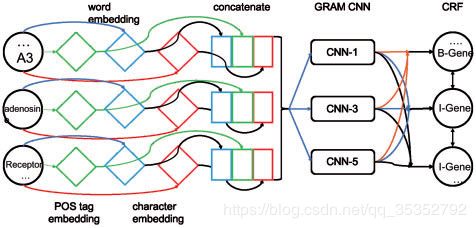
2.1 GRAM-CNN方法
1、得到词,词类标记集和字符嵌入结果。
2、将一个词的每个字母的字符嵌入结果和它对应的词嵌入结果以及词类嵌入结果聚合起来,得到一个连接向量。
3、将该连接向量输入到GRAM-CNN模型中,该模型有三个卷积核,尺寸分别为1,3,5,输出结果为每个词的局部特征。
4、上一步的输出作为CRF模型的输入,最终输出对应词的标签。
下图为该方法的整体框架。
2.1.1 嵌入方法
词嵌入的预训练模型来自2016年的一篇处理生物医学文本的文献,POS tag使用NLTK工具提取。
原文:During training,we fixed the word embeddings and trained POS tag embedding together with the whole system.(彷佛是词嵌入和词类标记一起训练)
词嵌入方法有一些缺陷,对于不在训练集词典中的词,将无法进行词嵌入表示。为了解决这个问题,文章加入了字符水平的嵌入方法。具体如下图所示,采用CNN结构。
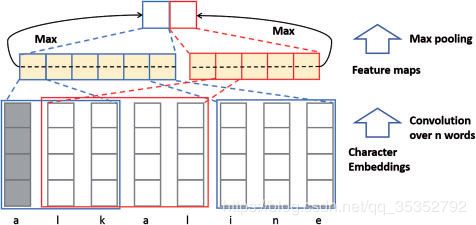
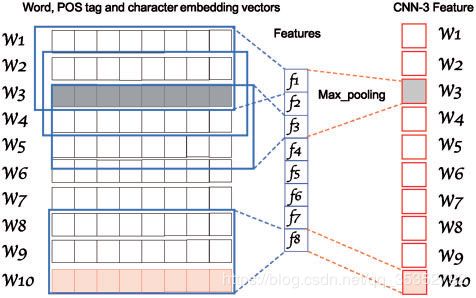
2.1.2 GRAM-CNN
此图为一个示例结构,在该示例中,输入为10个单词,卷积核尺寸为3。为了得到每个单词的局部信息,文章采用和每个单词相关的特征图的信息进行计算(最大池化操作),例如和 w 3 w_3 w3相关的特征图为 f 1 , f 2 , f 3 f_1,f_2,f_3 f1,f2,f3,和 w 10 w_{10} w10相关的特征图为 f 8 f_8 f8。
每个单词对应的GRAM-CNN结果再经过一个两层的全连接层,得到一个对应的label。
2.1.3 CRF
若一个句子包含n个单词,用 x x x={ x 1 , . . . , x n x_1,...,x_n x1,...,xn}表示该句子, y y y={ y 1 , . . . , y n y_1,...,y_n y1,...,yn}表示该句子的label。文章使用了CRF的一个变体,它被分解成单个标签的一元势和输出标签之间的每次转换的二元势,具体公式如下(这一部分不是很懂)。
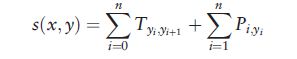
2.2 实施细节
这一部分讲述了各个方法的参数选择和初始化的方法。
1、word embedding时,若某单词无对应的嵌入结果,则用‘UNK’代替;
2、Character embedding和word embedding参数用了dropout
layer;
3、character embedding维度是25,POS tag embedding维度是15;
4、优化器选择SGD,学习率为0.002,衰减率0.95。
2.3 数据集
the BioCreative II Gene Mention task (BC2)
the NCBI disease corpus (NCBI)
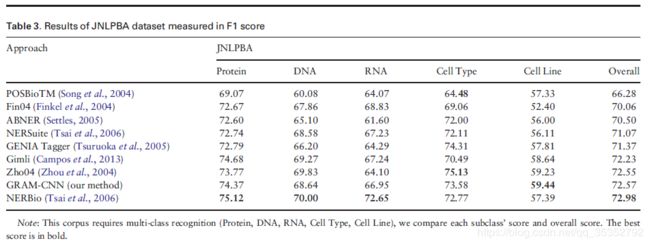
the JNLPBA corpus
各个数据集包含的命名实体类型、数据量以及训练集、测试集、验证集划分情况如下图所示。

2.4 评价标准
准确率、召回率、F1 score
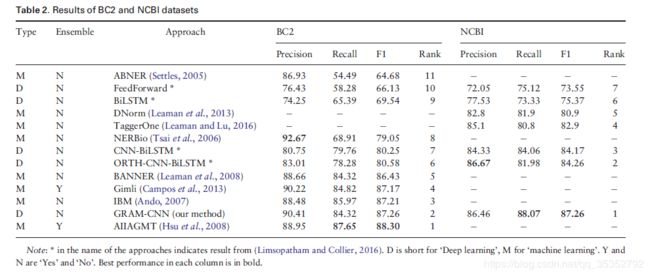
3 结果
4 讨论
1、GRAM-CNN适用于由一个词或一组连续的词组成的实体的识别,并且对拼写错误也有很强的鲁棒性。但该方法不适用于考虑重叠或不连贯的实体的识别,也不考虑表中出现的实体的识别(因为其只考虑局部相关信息)。
2、该方法耗时较长,对于JNLPBA和BC2数据集,训练过程大概需要五天,对于NCBI数据集,训练过程大约1.5天。
3、该方法对于一些复杂的情况,比如包含连词和标点符号的实体,将无法掌控。