文献阅读:神经网络提取生物医学文本中的关系
文献阅读:神经网络提取生物医学文本中的关系
- 题目
- 1 背景
- 2 相关工作
- 3 方法
- 3.1 生物医学关系提取
- 3.2 Dependency graphs and SDPs
- 3.3 句子嵌入表示
- 3.4 CNN和RNN的结合
- 4 结果和讨论
- 4.1 数据集
- 4.2 实验设置
- 4.3 PPI extraction的实验结果
- 4.4 PPI extraction的实验结果比较(与其他文献的方法)
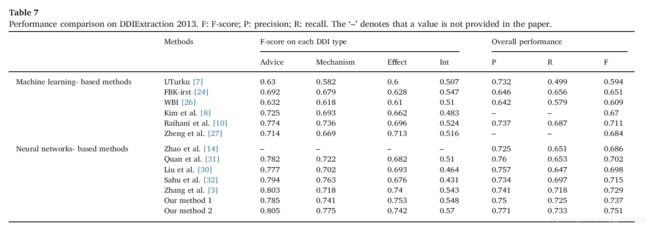
- 4.5 DDI extraction的实验结果
- 4.6 DDI extraction的实验结果比较(与其他文献的方法)
- 4.6 实验结果分析和未来的工作
题目
A hybrid model based on neural networks for biomedical relation extraction
1 背景
生物医学文献中包含的知识正以指数倍的速度增长,如果以人工的方式将这些关系提取出来,则要耗费大量的时间和精力。传统的方法包括pattern-based methods基于模式的方法、feature-based methods基于特征的方法和kernel-based methods基于核函数的方法。目前自动地将生物医学关系提取出来的方法结合了自然语言处理、机器学习和深度学习的多种模型。
CNN能够捕捉句子中的局部特征,RNN能够捕捉长期依赖的特征,但这两种方法都无法从句子本身学到足够多的信息,在关系抽取任务中,依赖图和语法树是很有价值和有用的。从依赖图或句法树中获取重要的词汇和句法信息将有助于生物医学关系的分类,特别是长句和复杂句的分类。
为了提高生物医学关系提取的性能,文章不仅使用了句子序列,而且整合了最短依赖路径(SDP)。SDP由dependency graph生成,包含了两个生物医学实体最重要的语义信息。CNN和RNN分别从句子序列和SDP中提取句法和语法特征,提取到的特征经过多层感知机进行整合,最后通过一层softmax层输出关系分类。
2 相关工作
现状(略掉)
3 方法
3.1 生物医学关系提取
怎么确定实体对??
简单来说,分为两步
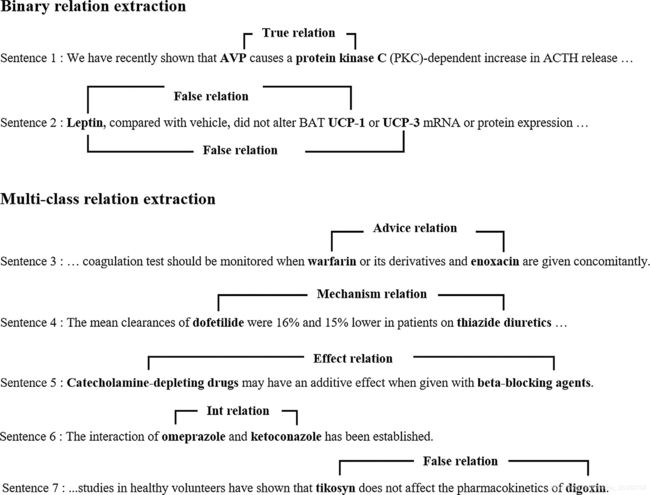
(1)二分类:确定一个实体对有无语义上的关系;
(2)多分类:确定由关系的实体对的具体关系。
3.2 Dependency graphs and SDPs
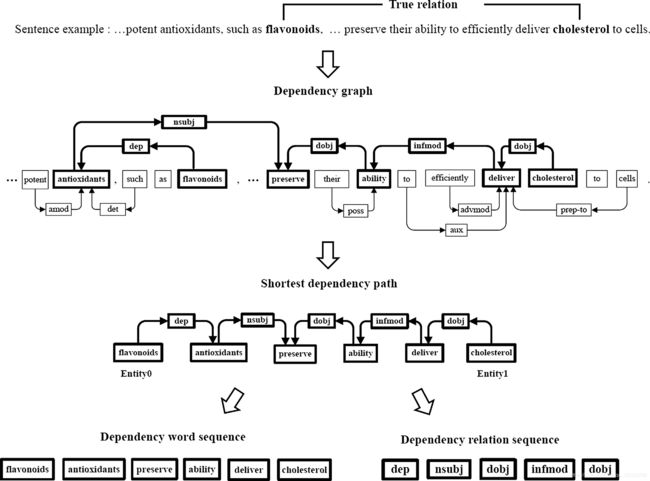
上图是一个例子,SDP能够将这个有多个分句的句子中的和“flavonoids” 、“cholesterol”有关的词以及关系都提取出来,之后神经网络可以对dependency word sequence和dependency relation sequence进行学习。
3.3 句子嵌入表示
{ w 1 , w 2 , . . . , w n w_1,w_2,...,w_n w1,w2,...,wn},{ d 1 , d 2 , . . . , d m d_1,d_2,...,d_m d1,d2,...,dm}以及{ r 1 , r 2 , . . . , r l r_1,r_2,...,r_l r1,r2,...,rl}分别代表the raw
sentence sequence,dependency word sequence和dependency relation
sequence。
W w o r d , W d e p , W d i s W_{word},W_{dep},W_{dis} Wword,Wdep,Wdis分别表示word embedding matrix,dependency
relation embedding matrix和position embedding matrix。
the raw sentence sequence和dependency word sequence可以用 W w o r d , W d i s W_{word},W_{dis} Wword,Wdis表示,dependency relation sequence可以用 W d e p W_{dep} Wdep表示。
3.4 CNN和RNN的结合
CNN用来学习dependency word sequence和dependency relation sequence的特征,因为这两个序列较短(最长的也不超过20个word),RNN用来学习the raw sentence sequence的特征,因为原生的句子一般非常长长长。整个结构如下图所示。
4 结果和讨论
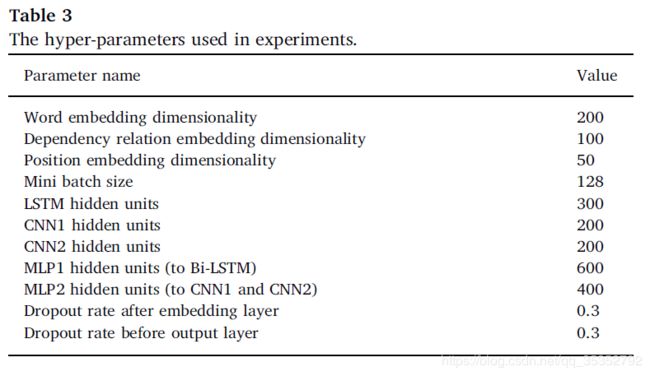
4.1 数据集
PPI extraction task(protein-protein interactions):五个公开的PPI语料库AIMed, BioInfer , IEPA ,HPRD50 和 LLL,该任务为二分类(判断语义关系是否存在)
DDI extraction tasks(drug-drug interactions):DDIExtraction 2013,多分类,四个类型(Advice, Mechanism, Effect, and Int)

使用Stanford parser来生成句子的dependency syntactic information,而SDP基于dependency syntactic information得到。用Dijkstra算法得到dependency graph中两个实体的SDP。
4.2 实验设置
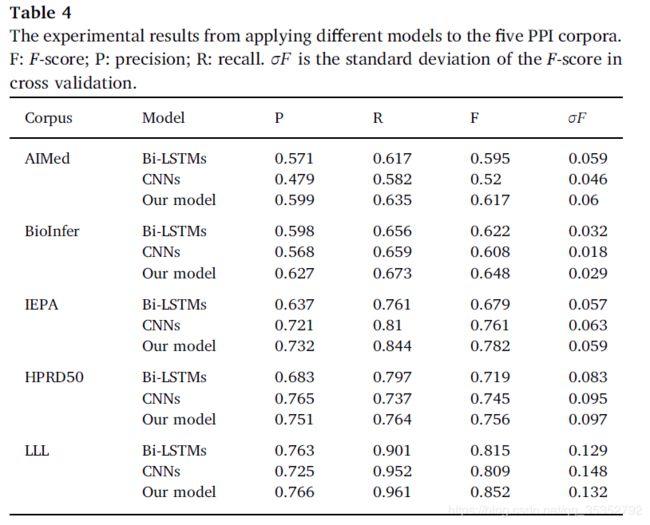
4.3 PPI extraction的实验结果
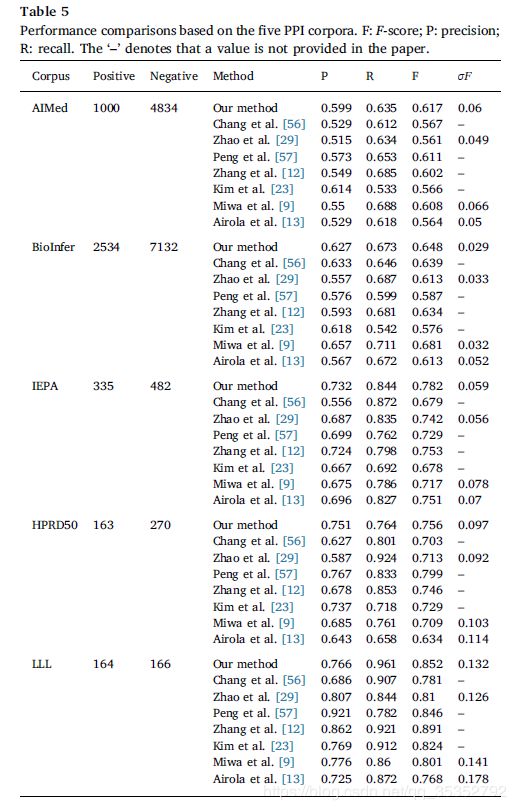
4.4 PPI extraction的实验结果比较(与其他文献的方法)
4.5 DDI extraction的实验结果
4.6 DDI extraction的实验结果比较(与其他文献的方法)
4.6 实验结果分析和未来的工作
1、混合模型让RNN和CNN进行互补,从而实现了最好的实验结果。
2、false result的原因:一个是句子语义太复杂,分句较多;另一个是句子存在否定表达以及结构复杂。
3、有监督的方法非常依赖于训练集,而得到大量有标签的训练集很困难,所以可以考虑加入无监督的方法(考虑生物医学文献和领域知识)