数据挖掘十大算法(二):K-Means、二分K-均值 python和sklearn实现
早在刚接触数据挖掘算法时就已经看过,以及使用过简单的K-均值算法来做聚类,现在为了进一步的掌握该知识,通过机器学习实战又看了一遍,由于相对于其它算法较简单,所以看的也比较快,同时也学习了一下更为强大的二分K-均值算法,该算法建立在K-Means算法上,但难度不大,理论知识也很好理解,所以这里对两者的思路都记录一下。本篇文章主要内容(K-Means原理、二分K-Means原理、基础代码实现、sklearn实现)等。
K-Means原理:
聚类是一种无监督的学习,它将相似的对象归到同一个簇中。有点像全自动分类,聚类方法几乎可以应用于所有对象,簇内的对象越相似,聚类的效果越好。K-均值(K-Means)属于聚类算法,之所以称为K-均值是因为它可以发现k个不同的簇,且每个簇的中心采用簇中所含值得均值计算而成。
实现聚类的思想:
- 随机选择K个中心值
- 根据样本数据各点与中心点的距离来进行归簇
- 通过整个簇的均值,重置每个簇的中心值
- 重复2、3,直至达到最大的迭代次数(例如:我这里的条件设置为本次与上次的平方误差相等)
看了这个步骤,感觉不会算法得人,都应该有了大概的思路。下面通过作者的代码来演示具体的操作:
代码:
样例数据(来自第十章):
from numpy import *
import matplotlib.pyplot as plt
# 加载本地数据
def loadDataSet(fileName):
dataMat = []
fr = open(fileName)
for line in fr.readlines():
curLine = line.strip().split('\t')
fltLine = list(map(float,curLine)) # 映射所有数据为浮点数
dataMat.append(fltLine)
return dataMat
# 欧式距离计算
def distEclud(vecA, vecB):
return sqrt(sum(power(vecA - vecB, 2))) # 格式相同的两个向量做运算
# 中心点生成 随机生成最小到最大值之间的值
def randCent(dataSet, k):
n = shape(dataSet)[1]
centroids = mat(zeros((k,n))) # 创建中心点,由于需要与数据向量做运算,所以每个中心点与数据得格式应该一致(特征列)
for j in range(n): # 循环所有特征列,获得每个中心点该列的随机值
minJ = min(dataSet[:,j])
rangeJ = float(max(dataSet[:,j]) - minJ)
centroids[:,j] = mat(minJ + rangeJ * random.rand(k,1)) # 获得每列的随机值 一列一列生成
return centroids
# 返回 中心点矩阵和聚类信息
def kMeans(dataSet, k, distMeas=distEclud, createCent=randCent):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2))) # 创建一个矩阵用于记录该样本 (所属中心点 与该点距离)
centroids = createCent(dataSet, k)
clusterChanged = True
while clusterChanged:
clusterChanged = False # 如果没有点更新则为退出
for i in range(m):
minDist = inf; minIndex = -1
for j in range(k): # 每个样本点需要与 所有 的中心点作比较
distJI = distMeas(centroids[j,:],dataSet[i,:]) # 距离计算
if distJI < minDist:
minDist = distJI; minIndex = j
if clusterAssment[i,0] != minIndex: # 若记录矩阵的i样本的所属中心点更新,则为True,while下次继续循环更新
clusterChanged = True
clusterAssment[i,:] = minIndex,minDist**2 # 记录该点的两个信息
# print(centroids)
for cent in range(k): # 重新计算中心点
# print(dataSet[nonzero(clusterAssment[:,0] == cent)[0]]) # nonzero返回True样本的下标
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]] # 得到属于该中心点的所有样本数据
centroids[cent,:] = mean(ptsInClust, axis=0) # 求每列的均值替换原来的中心点
return centroids, clusterAssment
datMat = mat(loadDataSet('testSet.txt'))
myCentroids,clustAssing = kMeans(datMat,4)
print(myCentroids)
print(clustAssing)
fig = plt.figure()
ax = fig.add_subplot(111)
ax.scatter(myCentroids[:,0].flatten().A[0],myCentroids[:,1].flatten().A[0],color='r',s=60)
ax.scatter(datMat[:,0].flatten().A[0],datMat[:,1].flatten().A[0])
plt.show()中心点:
每个点的所属簇、及平方和误差:
K-Means算法很简单,实现起来也不困难,这是它的优势,但有时你可能会发现K-均值收敛,但聚类效果并不是很好,这是因为K-均值收敛到了局部最小值,而非全局最小值(局部最小值表示结果还可以,但还可以更好,全局最小值表示结果可能是最好的了)。
由此我们需要使用后处理来提高聚类性能,一种度量聚类效果的指标是SSE(误差平方和),就是上面clusterAssment的第一列之和。SSE越小表示越接近质心点,由于取了平方,所以更重视距离远的点。增加簇可以减少SSE,但这可能不符合要求,另一种方法是将具有较大SSE的簇进行二分,具体实现时可以将最大簇包含的点过滤出来并在这些点上运用K-均值算法,K设为2。
基于划分簇的思想下面介绍二分K-均值
二分K-均值:
首先将所有点作为一个簇,然后将该簇一分为二。之后选择一个簇继续进行划分,选择哪一个簇进行划分取决于对其划分是否可以最大程度降低SSE的值。而划分就是上面提到的K-均值的思想了,利用上面的函数k设为2来划分。通过不断重复的操作,直到达到需要的簇数量。
代码:
样例数据:
# 二分均值聚类 SSE误差平方和 nonzero判断非0或给定条件,返回两个数组,[0]为True的下标组
def biKmeans(dataSet, k, distMeas=distEclud):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2))) # 保存数据点的信息(所属类、误差)
centroid0 = mean(dataSet, axis=0).tolist()[0] # 根据数据集均值获得第一个簇中心点
centList =[centroid0] # 创建一个带有质心的 [列表],因为后面还会添加至k个质心
for j in range(m):
clusterAssment[j,1] = distMeas(mat(centroid0), dataSet[j,:])**2 # 求得dataSet点与质心点的SSE
while (len(centList) < k):
lowestSSE = inf
for i in range(len(centList)):
ptsInCurrCluster = dataSet[nonzero(clusterAssment[:,0].A==i)[0],:] # 与上面kmeans一样获得属于该质心点的所有样本数据
# 二分类
centroidMat, splitClustAss = kMeans(ptsInCurrCluster, 2, distMeas) # 返回中心点信息、该数据集聚类信息
sseSplit = sum(splitClustAss[:,1]) # 这是划分数据的SSE 加上未划分的 作为本次划分的总误差
sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:,0].A!=i)[0],1]) # 这是未划分的数据集的SSE
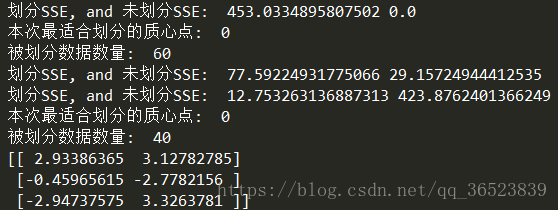
print("划分SSE, and 未划分SSE: ",sseSplit,sseNotSplit)
if (sseSplit + sseNotSplit) < lowestSSE: # 将划分与未划分的SSE求和与最小SSE相比较 确定是否划分
bestCentToSplit = i # 得出当前最适合做划分的中心点
bestNewCents = centroidMat # 划分后的两个新中心点
bestClustAss = splitClustAss.copy() # 划分点的聚类信息
lowestSSE = sseSplit + sseNotSplit
bestClustAss[nonzero(bestClustAss[:,0].A == 1)[0],0] = len(centList) # 由于是二分,所有只有0,1两个簇编号,将属于1的所属信息转为下一个中心点
bestClustAss[nonzero(bestClustAss[:,0].A == 0)[0],0] = bestCentToSplit # 将属于0的所属信息替换用来聚类的中心点
print('本次最适合划分的质心点: ',bestCentToSplit)
print('被划分数据数量: ', len(bestClustAss))
centList[bestCentToSplit] = bestNewCents[0,:].tolist()[0] # 与上面两条替换信息相类似,这里是替换中心点信息,上面是替换数据点所属信息
centList.append(bestNewCents[1,:].tolist()[0])
clusterAssment[nonzero(clusterAssment[:,0].A == bestCentToSplit)[0],:] = bestClustAss # 替换部分用来聚类的数据的所属中心点与误差平方和 为新的数据
return mat(centList), clusterAssment
datMat3 = mat(loadDataSet('testSet2.txt'))
centList,myNewAssments = biKmeans(datMat3,3)
print(centList)
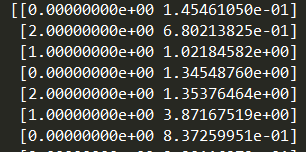
print(myNewAssments)
fig = plt.figure()
ax = fig.add_subplot(111)
ax.scatter(centList[:,0].flatten().A[0],centList[:,1].flatten().A[0],color='r',s=60)
ax.scatter(datMat3[:,0].flatten().A[0],datMat3[:,1].flatten().A[0])
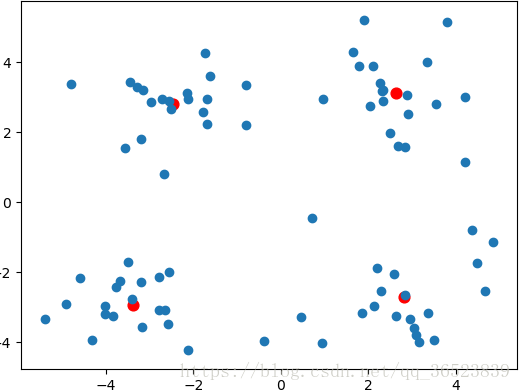
plt.show()
中心值:
每个点的所属簇、及SSE值:
大部分的解释都在代码上,这里就不再累述了。二分K-均值相比于K-均值效果更好,克服了K-Means算法收敛于局部最小值得问题。
sklearn实现:
由于篇幅问题,这里简单说一下。通过sklearn的cluster可以使用KMeans算法,或者使用另一种方法Mini Batch K-Means。
Mini Batch K-Means算法是K-Means算法的变种,采用小批量的数据子集减小计算时间,同时仍试图优化目标函数,这里所谓的小批量是指每次训练算法时所随机抽取的数据子集,采用这些随机产生的子集进行训练算法,大大减小了计算时间,与其他算法相比,减少了k-均值的收敛时间,小批量k-均值产生的结果,一般只略差于标准算法。
官方文档的连接:http://scikit-learn.org/stable/modules/clustering.html#mini-batch-kmeans
可以参考的博客:
https://blog.csdn.net/sinat_26917383/article/details/70240628 参数含义
https://blog.csdn.net/gamer_gyt/article/details/51244850
当然如果可以的话,最好使用官方的文档进行深入的学习。
参考书籍:《机器学习实战》