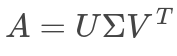![]()
Spark分布式机器学习源码分析:奇异值分解(SVD)与主成分分析(PCA)
原理 Spark是一个极为优秀的大数据框架,在大数据批处理上基本无人能敌,流处理上也有一席之地,机器学习则是当前正火热AI人工智能的驱动引擎,在大数据场景下如何发挥AI技术成为优秀的大数据挖掘工程师必备技能。本文结合机器学习思想与Spark框架代码结构来实现分布式机器学习过程,希望与大家一起学习进步~
本文采用的组件版本为:Ubuntu 19.10、Jdk 1.8.0_241、Scala 2.11.12、Hadoop 3.2.1、Spark 2.4.5,老规矩先开启一系列Hadoop、Spark服务与Spark-shell窗口:
![]()
降维是减少所考虑变量数量的过程。它可用于从原始和嘈杂的特征中提取潜在特征,或者在保持结构的同时压缩数据。spark.mllib为RowMatrix类提供降维支持。
1
SVD介绍
奇异值分解(SVD)将矩阵分解为三个矩阵:U,Σ和V,使得:
这里U是一个正交矩阵,其列称为左奇异向量,Σ是对角矩阵,其中非对角线按降序排列,其对角线称为奇异值,V是一个正交矩阵,其列称为右奇异向量。
对于大型矩阵,通常不需要完整的因式分解,而仅需要顶部奇异值及其关联的奇异矢量。这样可以节省存储空间,降低噪声并恢复矩阵的低阶结构。如果我们保留前k个奇异值,那么所得的低秩矩阵的维将为:
U:m*kΣ:k*k
V:n*k
我们假设n小于m。奇异值和右奇异向量是从Gramian矩阵ATA的特征值和特征向量得出的。如果用户通过computeU参数请求,则通过矩阵乘法将存储左奇异矢量Ui的矩阵计算为U = A(VS-1)。实际使用的方法是根据计算成本自动确定的:
如果n小(n <100)或k与n(k> n / 2)相比较大,我们首先计算Gramian矩阵,然后在驱动程序上局部计算其最高特征值和特征向量。这需要在每个执行器和驱动程序上进行一次O(n2)存储操作,并在驱动程序上进行O(n2k)时间处理。
否则,我们将以分布式方式计算(ATA)v并将其发送到ARPACK,以计算驱动程序节点上(ATA)的最高特征值和特征向量。这需要O(k)次通过,每个执行程序上的O(n)存储以及驱动程序上的O(nk)存储。
2
SVD实例
spark.mllib为RowMatrix类中提供的面向行的矩阵提供SVD功能。
import org.apache.spark.mllib.linalg.Matrix
import org.apache.spark.mllib.linalg.SingularValueDecomposition
import org.apache.spark.mllib.linalg.Vector
import org.apache.spark.mllib.linalg.Vectors
import org.apache.spark.mllib.linalg.distributed.RowMatrix
// 定义数组
val data = Array(
Vectors.sparse(5, Seq((1, 1.0), (3, 7.0))),
Vectors.dense(2.0, 0.0, 3.0, 4.0, 5.0),
Vectors.dense(4.0, 0.0, 0.0, 6.0, 7.0))
val rows = sc.parallelize(data)
val mat: RowMatrix = new RowMatrix(rows)
// 计算前5个奇异值和相应的奇异向量。
val svd: SingularValueDecomposition[RowMatrix, Matrix] = mat.computeSVD(5, computeU = true)
val U: RowMatrix = svd.U // U因子
val s: Vector = svd.s // 奇异值存储在一个本地dense向量中
val V: Matrix = svd.V // V因子
3
SVD源码分析
计算SVD的源码如下:
def computeSVD(
k: Int,
computeU: Boolean = false,
rCond: Double = 1e-9): SingularValueDecomposition[RowMatrix, Matrix] = {
// 迭代次数
val maxIter = math.max(300, k * 3)
// 阈值
val tol = 1e-10
computeSVD(k, computeU, rCond, maxIter, tol, "auto")
}
computeSVD(k, computeU, rCond, maxIter, tol, "auto")的实现分为三步。分别是选择计算模式,特征值分解,计算V,U,Sigma。下面分别介绍这三步。首先是选择计算模式:
val computeMode = mode match {
case "auto" =>
if (k > 5000) {
logWarning(s"computing svd with k=$k and n=$n, please check necessity")
}
if (n < 100 || (k > n / 2 && n <= 15000)) {
// 满足上述条件,首先计算方阵,然后本地计算特征值,避免数据传递
if (k < n / 3) {
SVDMode.LocalARPACK
} else {
SVDMode.LocalLAPACK
}
} else {
// 分布式实现
SVDMode.DistARPACK
}
case "local-svd" => SVDMode.LocalLAPACK
case "local-eigs" => SVDMode.LocalARPACK
case "dist-eigs" => SVDMode.DistARPACK
}
特征值分解:
val (sigmaSquares: BDV[Double], u: BDM[Double]) = computeMode match {
case SVDMode.LocalARPACK =>
val G = computeGramianMatrix().toBreeze.asInstanceOf[BDM[Double]]
EigenValueDecomposition.symmetricEigs(v => G * v, n, k, tol, maxIter)
case SVDMode.LocalLAPACK =>
// breeze (v0.10) svd latent constraint, 7 * n * n + 4 * n < Int.MaxValue
val G = computeGramianMatrix().toBreeze.asInstanceOf[BDM[Double]]
val brzSvd.SVD(uFull: BDM[Double], sigmaSquaresFull: BDV[Double], _) = brzSvd(G)
(sigmaSquaresFull, uFull)
case SVDMode.DistARPACK =>
if (rows.getStorageLevel == StorageLevel.NONE) {
logWarning("The input data is not directly cached, which may hurt performance if its"
+ " parent RDDs are also uncached.")
}
EigenValueDecomposition.symmetricEigs(multiplyGramianMatrixBy, n, k, tol, maxIter)
}
计算U,V以及Sigma:
//获取特征值向量
val sigmas: BDV[Double] = brzSqrt(sigmaSquares)
val sigma0 = sigmas(0)
val threshold = rCond * sigma0
var i = 0
// sigmas的长度可能会小于k
// 所以使用 i < min(k, sigmas.length) 代替 i < k.
if (sigmas.length < k) {
logWarning(s"Requested $k singular values but only found ${sigmas.length} converged.")
}
while (i < math.min(k, sigmas.length) && sigmas(i) >= threshold) {
i += 1
}
val sk = i
if (sk < k) {
logWarning(s"Requested $k singular values but only found $sk nonzeros.")
}
//计算s,也即sigma
val s = Vectors.dense(Arrays.copyOfRange(sigmas.data, 0, sk))
//计算V
val V = Matrices.dense(n, sk, Arrays.copyOfRange(u.data, 0, n * sk))
//计算U
// N = Vk * Sk^{-1}
val N = new BDM[Double](n, sk, Arrays.copyOfRange(u.data, 0, n * sk))
var i = 0
var j = 0
while (j < sk) {
i = 0
val sigma = sigmas(j)
while (i < n) {
//对角矩阵的逆即为倒数
N(i, j) /= sigma
i += 1
}
j += 1
}
//U=A * N
val U = this.multiply(Matrices.fromBreeze(N))
4
PCA介绍
主成分分析是最常用的一种降维方法。我们首先考虑一个问题:对于正交矩阵空间中的样本点,如何用一个超平面对所有样本进行恰当的表达。容易想到,如果这样的超平面存在,那么他大概应该具有下面的性质。 基于最近重构性和最大可分性,能分别得到主成分分析的两种等价推导。
最近重构性:样本点到超平面的距离都足够近
最大可分性:样本点在这个超平面上的投影尽可能分开
主成分分析(PCA)是一种统计方法,用于查找旋转,以使第一个坐标具有最大的方差,而每个后续坐标又具有最大的方差。旋转矩阵的列称为主成分。PCA被广泛用于降维。spark.mllib支持将PCA用于以行格式和任何Vector存储的高而瘦的矩阵。
5
PCA实例
以下代码演示了如何在RowMatrix上计算主成分并将其用于将向量投影到低维空间中。
import org.apache.spark.mllib.linalg.Matrix
import org.apache.spark.mllib.linalg.Vectors
import org.apache.spark.mllib.linalg.distributed.RowMatrix
val data = Array(
Vectors.sparse(5, Seq((1, 1.0), (3, 7.0))),
Vectors.dense(2.0, 0.0, 3.0, 4.0, 5.0),
Vectors.dense(4.0, 0.0, 0.0, 6.0, 7.0))
val rows = sc.parallelize(data)
val mat: RowMatrix = new RowMatrix(rows)
// 计算4个主成分
// 主成分存储在本地dense矩阵中
val pc: Matrix = mat.computePrincipalComponents(4)
// 将行投影到前4个主要成分所跨越的线性空间
val projected: RowMatrix = mat.multiply(pc)
6
PCA源码分析
主成分分析的实现代码在RowMatrix中实现。源码如下:
def computePrincipalComponents(k: Int): Matrix = {
val n = numCols().toInt
//计算协方差矩阵
val Cov = computeCovariance().toBreeze.asInstanceOf[BDM[Double]]
//特征值分解
val brzSvd.SVD(u: BDM[Double], _, _) = brzSvd(Cov)
if (k == n) {
Matrices.dense(n, k, u.data)
} else {
Matrices.dense(n, k, Arrays.copyOfRange(u.data, 0, n * k))
}
}
这段代码首先会计算样本的协方差矩阵,然后在通过breeze的svd方法进行奇异值分解。这里由于协方差矩阵是方阵,所以奇异值分解等价于特征值分解。下面是计算协方差的代码:
def computeCovariance(): Matrix = {
val n = numCols().toInt
checkNumColumns(n)
val (m, mean) = rows.treeAggregate[(Long, BDV[Double])]((0L, BDV.zeros[Double](n)))(
seqOp = (s: (Long, BDV[Double]), v: Vector) => (s._1 + 1L, s._2 += v.toBreeze),
combOp = (s1: (Long, BDV[Double]), s2: (Long, BDV[Double])) =>
(s1._1 + s2._1, s1._2 += s2._2)
)
updateNumRows(m)
mean :/= m.toDouble
// We use the formula Cov(X, Y) = E[X * Y] - E[X] E[Y], which is not accurate if E[X * Y] is
// large but Cov(X, Y) is small, but it is good for sparse computation.
// TODO: find a fast and stable way for sparse data.
val G = computeGramianMatrix().toBreeze.asInstanceOf[BDM[Double]]
var i = 0
var j = 0
val m1 = m - 1.0
var alpha = 0.0
while (i < n) {
alpha = m / m1 * mean(i)
j = i
while (j < n) {
val Gij = G(i, j) / m1 - alpha * mean(j)
G(i, j) = Gij
G(j, i) = Gij
j += 1
}
i += 1
}
Matrices.fromBreeze(G)
}
Spark 降维算法的内容至此结束,有关Spark的基础文章可参考前文:
Spark分布式机器学习源码分析:矩阵向量
Spark分布式机器学习源码分析:基本统计
Spark分布式机器学习源码分析:线性模型
Spark分布式机器学习源码分析:朴素贝叶斯
Spark分布式机器学习源码分析:决策树模型
Spark分布式机器学习源码分析:集成树模型
Spark分布式机器学习源码分析:协同过滤
Spark分布式机器学习源码分析:K-means
Spark分布式机器学习源码分析:隐式狄利克雷分布(LDA)
参考链接:
http://spark.apache.org/docs/latest/mllib-clustering.html
https://github.com/endymecy/spark-ml-source-analysis
![]()
历史推荐
“高频面经”之数据分析篇
“高频面经”之数据结构与算法篇
“高频面经”之大数据研发篇
“高频面经”之机器学习篇
“高频面经”之深度学习篇
爬虫实战:Selenium爬取京东商品
爬虫实战:豆瓣电影top250爬取
爬虫实战:Scrapy框架爬取QQ音乐
![]()
数据分析与挖掘
数据结构与算法
机器学习与大数据组件
欢迎关注,感谢“在看”,随缘稀罕~
![]()
一个赞,晚餐加鸡腿