机器学习笔记(14)——sklearn降维方法举例(RandomProjection,TSVD,t-SNE)
sklearn降维方法举例
以datasets.digits数据为例导入相关包
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import time
from sklearn.datasets import load_digits大样本数据的可视化是一个相对比较麻烦的事情,
一般情况下我们都要用到降维的方法先处理特征。我们找一个例子来看看,可以怎么做,比如我们数据集取经典的『手写数字集』
Each datapoint is a 8x8 image of a digit.
Classes->10
Samples per class->180
Samples total->1797
Dimensionality->64
Features integers->0-16
导入数据,0,1,2,3,4,5,6这7个数字的手写图像
digits = load_digits(n_class=7)#integer
X = digits.data
y = digits.target
n_samples,n_features = X.shape输出图像
from matplotlib import offsetbox
def plot_embedding(X, title=None):
x_min, x_max = np.min(X, 0), np.max(X, 0)
X = (X - x_min) / (x_max - x_min)#正则化
print X
plt.figure(figsize=(10, 10))
ax = plt.subplot(111)
for i in range(X.shape[0]):
plt.text(X[i, 0], X[i, 1], str(digits.target[i]),
color=plt.cm.Set1(y[i] / 10.),
fontdict={'weight': 'bold', 'size': 12})
#打印彩色字体
if hasattr(offsetbox, 'AnnotationBbox'):
# only print thumbnails with matplotlib > 1.0
shown_images = np.array([[1., 1.]]) # just something big
for i in range(digits.data.shape[0]):
dist = np.sum((X[i] - shown_images) ** 2, 1)
if np.min(dist) < 4e-3:
# don't show points that are too close
continue
shown_images = np.r_[shown_images, [X[i]]]
imagebox = offsetbox.AnnotationBbox(
offsetbox.OffsetImage(digits.images[i], cmap=plt.cm.gray_r),
X[i])
ax.add_artist(imagebox)#输出图上输出图片
plt.xticks([]), plt.yticks([])
if title is not None:
plt.title(title)
1.随机投射(Random-Projection)
首先我们来看一个问题, 如果你手头有一组数据 X ∈ R n X \in R^n X∈Rn, 它的维数太高, 从而不得不进行降维至 R k R^k Rk, 你会怎么办?
相信不少人反射性地求它的 x ’ x x’x x’x,然后转化为延最大方差展开的问题。 当然这个方法需要求解固有值和固有向量。对于测试用的小数据一切似乎都很好, 但如果我给你的是50G大小csv格式的Twitter发言呢?如果需要对全部单词降维, 恐怕半个月吧。 因此我们需要个更加快速的方法。
那么现在再来想, 既然要快, 那我们就用个最简单的方法: 随机在高维空间里选几个单位向量 e i e_i ei, 但注意, 这里我们仅仅要求是单位向量, 并没有要求它们之间必须正交 < e i , e j > = 0 <e_i,e_j>=0 <ei,ej>=0, 因此你可以随便选。 最后, 我们把高维数据投影到选择的这一组基上。也就完成了降维。
随便选的轴如何能保证降维效果? 但它是有数学依据的, 叫做Johnson-Lindenstrauss Lemma。这个定理保证了任何降维方法的精度上下限。
#####Johnson-Lindenstrauss Lemma
假设我们有数据 x ∈ R n x \in R^n x∈Rn, 而我们通过一种方法 f ( x ) f(x) f(x)将其降维成 y ∈ R k y \in R^k y∈Rk, 那么, 将为前后任意两点 a , b a,b a,b之间的距离有不等式保证:
( 1 − ϵ ) ∥ a − b ∥ 2 ≤ ∥ f ( a ) − f ( b ) ∥ 2 ≤ ( 1 + ϵ ) ∥ a − b ∥ 2 (1-\epsilon )\| a-b \| ^2 \le \| f(a)-f(b) \| ^2 \le (1+ \epsilon ) \| a-b \|^2 (1−ϵ)∥a−b∥2≤∥f(a)−f(b)∥2≤(1+ϵ)∥a−b∥2
对于随机映射来说, 只要注意到下面几点就行了:
- 1.不定式中的精度仅仅受制于维数、数据大小等因素, 与将为方法无关。
- 2.在维数差不是很大时, 总可以保证一个相对较高的精度, 不论用什么方法。
- 3.到这里一切就很明显了, 既然精度有上界, 那我们也就不必担心轴的选取,那么最简单的方法自然就是随机挑选了, 这也就产生的Random Projection这一方法。
Random Projection
简单来说,Random Projection流程就是
选择影射矩阵 R ∈ R K × N R \in \mathcal{R}^{K \times N} R∈RK×N。
用随机数填充影射矩阵。 可以选择uniform或者gaussian。
归一化每一个新的轴(影射矩阵中的每一行)。
对数据降维 y = R X y=R\mathcal{X} y=RX。
上个小节里面的JL-lemma保证的降维的效果不会太差。
从直观上来看看。
首先假设我们有数据 X = { x ∣ f i ( θ ) + σ } \mathcal{X}=\{ x | f^{i}(\theta)+\sigma \} X={x∣fi(θ)+σ}, 其中 θ \theta θ是一组参数, σ \sigma σ则是高斯噪声。 回想PCA方法, 我们很自然的认为所有的特征都是正交的, 我们既没有考虑它是不是有多个中心, 也没有考虑是不是有特殊结构, 然而对于实际数据, 很多时候并不是这样。 比如我们把取 f ( θ ) = S i ( θ i ) N ( A , σ i ) ∈ R 3 f(\theta)=S^{i}(\theta _i)N(A, \sigma^i) \in R^3 f(θ)=Si(θi)N(A,σi)∈R3, 其中 S i ∈ S O ( 3 ) S^{i} \in SO(3) Si∈SO(3), 这样我们得到的数据可能会像 × \times ×或者 ∗ * ∗一样, 显然用PCA并不能得到好的结果。
在这种情况下, 考虑在非常高维度的空间, 可以想象random projection这种撞大运的方法总会选到一些超平面能够接近数据的特征, 同时也能够想象如果目标维数过低, 那么命中的概率就会大大降低。
实现:
from sklearn import random_projection
rp = random_projection.SparseRandomProjection(n_components=2, random_state=42)
#随机映射算法
start_time = time.time()
X_projected = rp.fit_transform(X)#随机投射
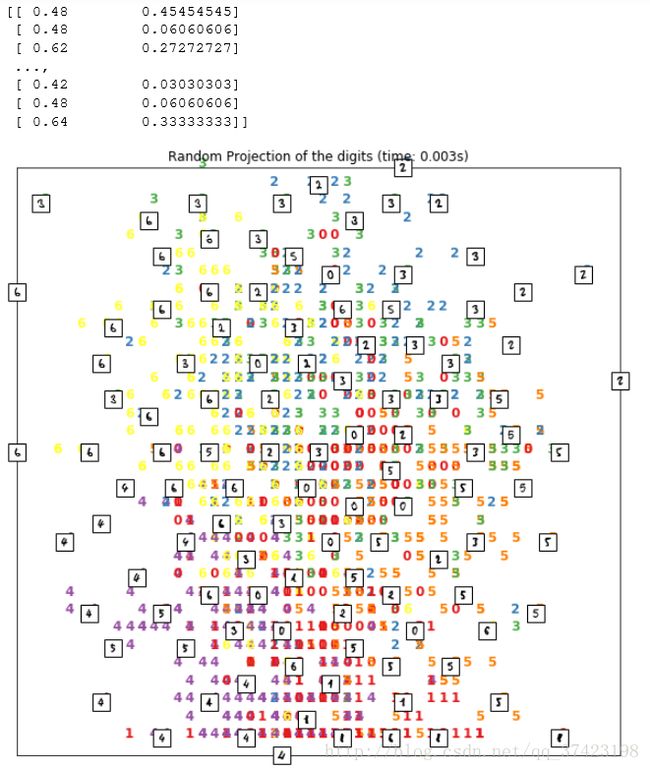
plot_embedding(X_projected, "Random Projection of the digits (time: %.3fs)" % (time.time() - start_time))
plt.show()时间很短但效果一般
官方描述:
def sparse_random_matrix(n_components, n_features, density='auto',
random_state=None):
"""Generalized Achlioptas random sparse matrix for random projection
Setting density to 1 / 3 will yield the original matrix by Dimitris
Achlioptas while setting a lower value will yield the generalization
by Ping Li et al.
If we note :math:`s = 1 / density`, the components of the random matrix are
drawn from:
- -sqrt(s) / sqrt(n_components) with probability 1 / 2s
- 0 with probability 1 - 1 / s
- +sqrt(s) / sqrt(n_components) with probability 1 / 2s
Read more in the :ref:`User Guide `.
Parameters
----------
n_components : int,
Dimensionality of the target projection space.
n_features : int,
Dimensionality of the original source space.
density : float in range ]0, 1] or 'auto', optional (default='auto')
Ratio of non-zero component in the random projection matrix.
If density = 'auto', the value is set to the minimum density
as recommended by Ping Li et al.: 1 / sqrt(n_features).
Use density = 1 / 3.0 if you want to reproduce the results from
Achlioptas, 2001.
random_state : integer, RandomState instance or None (default=None)
Control the pseudo random number generator used to generate the
matrix at fit time.
Returns
-------
components: numpy array or CSR matrix with shape [n_components, n_features]
The generated Gaussian random matrix.
"""
_check_input_size(n_components, n_features)
density = _check_density(density, n_features)
rng = check_random_state(random_state)
if density == 1:
# skip index generation if totally dense
components = rng.binomial(1, 0.5, (n_components, n_features)) * 2 - 1
return 1 / np.sqrt(n_components) * components
else:
# Generate location of non zero elements
indices = []
offset = 0
indptr = [offset]
for i in xrange(n_components):
# find the indices of the non-zero components for row i
n_nonzero_i = rng.binomial(n_features, density)
indices_i = sample_without_replacement(n_features, n_nonzero_i,
random_state=rng)
indices.append(indices_i)
offset += n_nonzero_i
indptr.append(offset)
indices = np.concatenate(indices)
# Among non zero components the probability of the sign is 50%/50%
data = rng.binomial(1, 0.5, size=np.size(indices)) * 2 - 1
# build the CSR structure by concatenating the rows
components = sp.csr_matrix((data, indices, indptr),
shape=(n_components, n_features))
return np.sqrt(1 / density) / np.sqrt(n_components) * components
2.TSVD(截断奇异值分解, TruncatedSVD,PCA的一种实现)
截断奇异值分解(Truncated singular value decomposition,TSVD)是一种矩阵因式分解(factorization)技术,将矩阵 M 分解成 U , Σ 和 V 。它与PCA很像,只是SVD分解是在数据矩阵上进行,而PCA是在数据的协方差矩阵上进行。通常,SVD用于发现矩阵的主成份。对于病态矩阵,目前主要的处理办法有预调节矩阵方法、区域分解法、正则化方法等,截断奇异值分解技术TSVD就是一种正则化方法,它牺牲部分精度换去解的稳定性,使得结果具有更高的泛化能力。
对于原始数据矩阵A(N*M) ,N代表样本个数,M代表维度,对其进行SVD分解:
A = U Σ V T , Σ = d i a g ( δ 1 , δ 2 , … , δ n ) = [ δ 1 0 0 . . 0 0 δ 2 0 . . 0 0 0 δ 3 . . 0 . . . . . . . . . . . . 0 0 0 . . δ n ] A = U\Sigma V^T, \Sigma=diag(\delta_1,\delta_2,…,\delta_n)=\left[ \begin{matrix} \delta_1&0&0&.&.&0\\ 0&\delta_2&0&.&.&0\\ 0&0&\delta_3&.&.&0\\ .&.&.&.&.&.\\ .&.&.&.&.&.\\ 0&0&0&.&.&\delta_n \end{matrix} \right] A=UΣVT,Σ=diag(δ1,δ2,…,δn)=⎣⎢⎢⎢⎢⎢⎢⎡δ100..00δ20..000δ3..0............000..δn⎦⎥⎥⎥⎥⎥⎥⎤
上式中的 δ \delta δ就是数据的奇异值,且 δ 1 \delta_1 δ1> δ 2 \delta_2 δ2> δ 3 \delta_3 δ3…,通常如果A非常病态,delta的后面就越趋向于0, δ 1 δ n \frac{\delta_1}{\delta_n} δnδ1就是数据的病态程度,越大说明病态程度越高,无用的特征越多,通常会截取前p个最大的奇异值,相应的U截取前p列,V截取前p列,这样A依然是N*M的矩阵,用这样的计算出来的A代替原始的A,就会比原始的A更稳定。
TSVD与一般SVD不同的是它可以产生一个指定维度的分解矩阵。例如,有一个 n×n 矩阵,通过SVD分解后仍然是一个 n×n 矩阵,而TSVD可以生成指定维度的矩阵。这样就可以实现降维了。
演示一下TSVD工作原理
In[1]:
import numpy as np
from scipy.linalg import svd
D = np.array([[1, 2], [1, 3], [1, 4]])
DOut[1]:
array([[1, 2],
[1, 3],
[1, 4]])In[2]:
U, S, V = svd(D, full_matrices=False)
#我们可以根据SVD的定义,用 UU , SS 和 VV 还原矩阵 DD
U.shape, S.shape, V.shapeOut[2]:
((3, 2), (2, 1), (2, 2))In[3]:
np.dot(U.dot(np.diag(S)), V)#还原Out[3]:
array([[ 1., 2.],
[ 1., 3.],
[ 1., 4.]])TruncatedSVD返回的矩阵是 U 和 S 的点积。如果我们想模拟TSVD,我们就去掉最新奇异值和对于 U 的列向量。例如,我们想要一个主成份,可以这样:
In[4]:
new_S = S[0]
new_U = U[:, 0]
new_U.dot(new_S)Out[4]:
array([-2.20719466, -3.16170819, -4.11622173])TruncatedSVD有个“陷阱”。随着随机数生成器状态的变化,TruncatedSVD连续地拟合会改变输出的符合。为了避免这个问题,建议只用TruncatedSVD拟合一次,然后用其他变换。这正是管线命令的另一个用处。
实现:
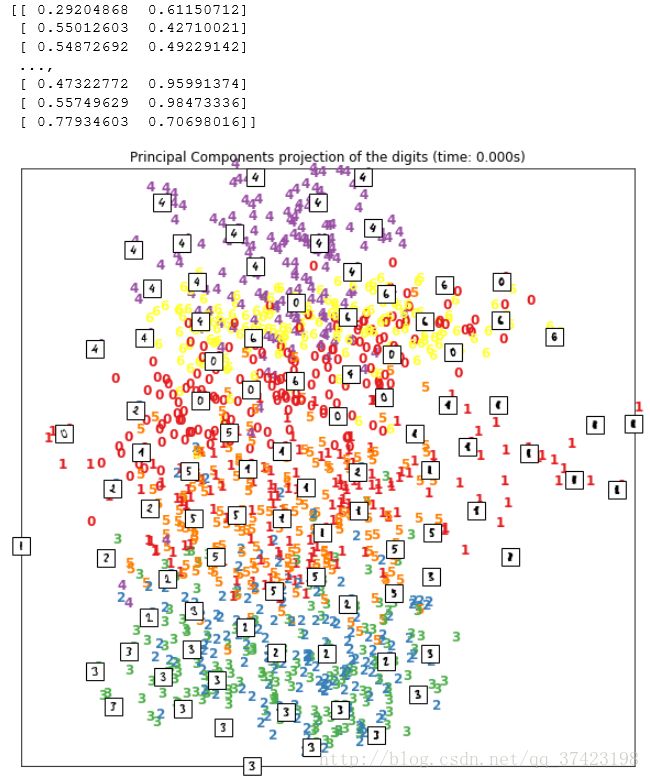
from sklearn import decomposition
X_pca = decomposition.TruncatedSVD(n_components=2).fit_transform(X)
start_time = time.time()
plot_embedding(X_pca,"Principal Components projection of the digits (time: %.3fs)" % (time.time() - start_time))
plt.show()3.t-SNE(t-分布邻域嵌入算法)
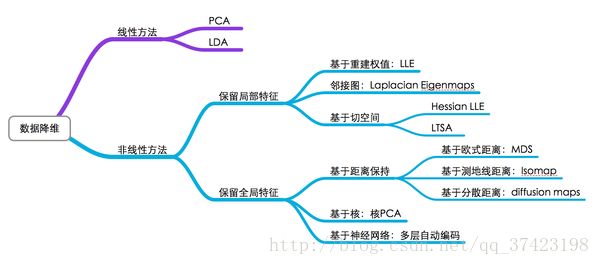
流形学习方法(Manifold Learning),简称流形学习,可以将流形学习方法分为线性的和非线性的两种,线性的流形学习方法如我们熟知的主成份分析(PCA),非线性的流形学习方法如等距映射(Isomap)、拉普拉斯特征映射(Laplacian eigenmaps,LE)、局部线性嵌入(Locally-linear embedding,LLE)。
t-SNE详细介绍:http://lvdmaaten.github.io/tsne/
from sklearn import manifold
#降维
tsne = manifold.TSNE(n_components=2, init='pca', random_state=0)
start_time = time.time()
X_tsne = tsne.fit_transform(X)
#绘图
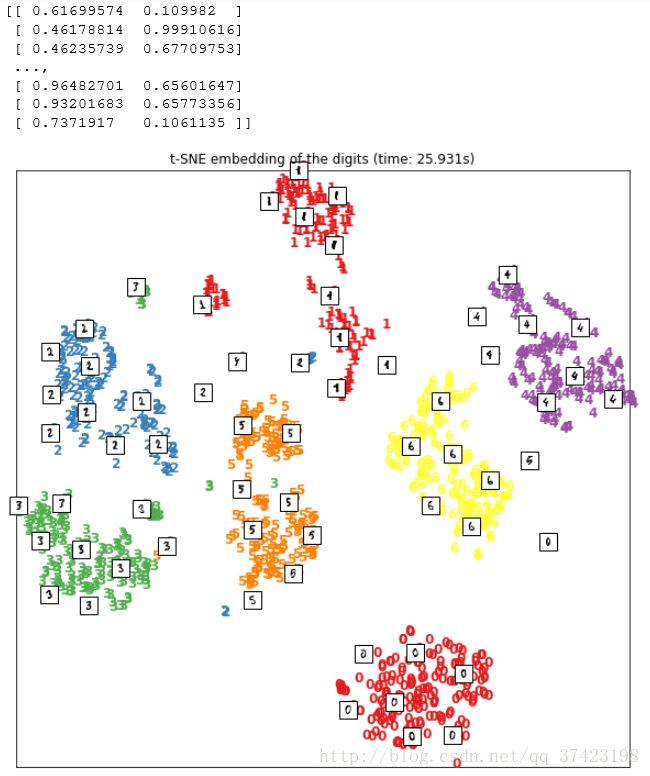
plot_embedding(X_tsne,
"t-SNE embedding of the digits (time: %.3fs)" % (time.time() - start_time))
plt.show()
#这个非线性变换降维过后,仅仅2维的特征,就可以将原始数据的不同类别,在平面上很好地划分开。
#不过t-SNE也有它的缺点,一般说来,相对于线性变换的降维,它需要更多的计算时间。