RDKit | 化学信息学与AI
RDKit | 化学信息学与AI
介绍RDKit相关知识点和运用以及RDKit作为处理化学、生物、药学和材料学科中分子数据作为可输入机器学习和深度学习模型的重要工具应用。内容涵盖了基于RDKit的Python3的分子的读写、化合物的分子指纹和分子描述符计算、化合物的2D/2D比对、化合物相似性搜索、化合物骨架分析和亚结构搜索、RMSD计算与构象生成优化、分子相似图与聚类分析、化学反应处理、可视化与化学空间探索及RDkit相关的机器学习、深度学习应用过程详解
1
RDKit简介
开源化学信息学与机器学习工具包
RDKit在2000-2006年期间在Rational Discovery开发和使用,用于构建吸收、分布、代谢、代谢、毒性和生物活性的预测模型。
2006年6月Rational Discovery被关闭,但该工具包在BSD许可证下作为开源发布。
目前,RDKit的开源开发由诺华积极贡献,其中包括诺华捐赠的源代码。
2
RDKit特点
商业友好型BSD许可证
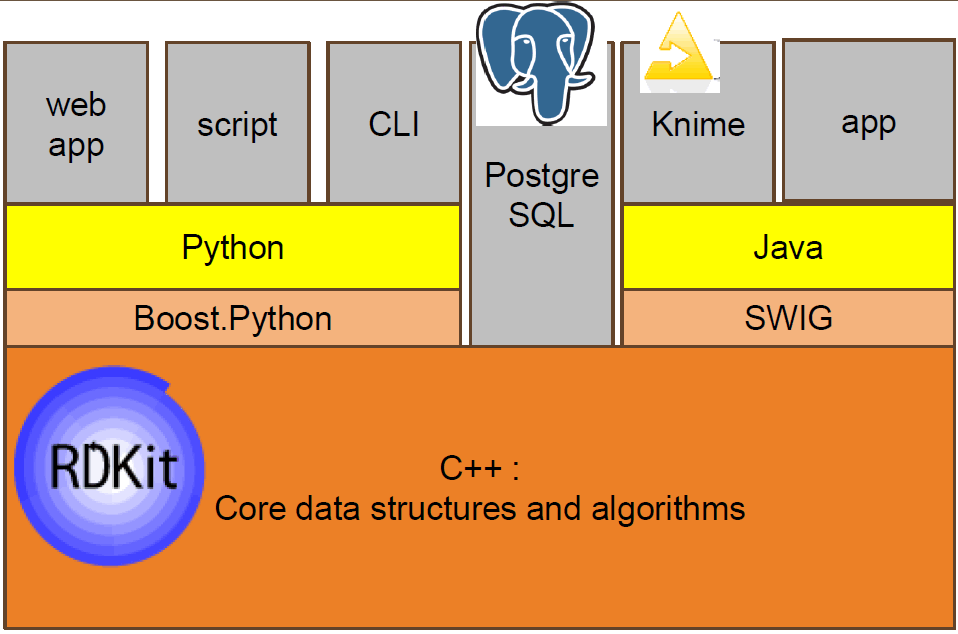
核心数据结构和算法由C ++编写
使用Boost.Python生成的Python 3.x包装器
用SWIG生成的Java和C#包装器
2D和3D分子操作
机器学习与深度学习的分子指纹和分子描述符生成
PostgreSQL分子数据库集成
KNIME的化学信息学Nod
RDKit提供各种功能,如不同的化学I/O格式,包括SMILES/SMARTS,结构数据格式(SDF),Thor数据树(TDT),Sybyl线符号(SLN),mol2和蛋白质结构文件(PDB);子结构搜索; 标准SMILES; 手性支持;化学转化;化学反应;分子序列化;相似性/多样性选择;二维药效团;三维维药效团;分层子图/片段分析; Bemis和Murcko骨架;逆合成组合分析及分子碎裂(RECAP); 多分子最大共同亚结构;功能图;基于形状的相似性;基于RMSD的分子比对;基于形状的对齐;使用Open3-DALIGN算法的无监督分子-分子比对;与PyMOL进行3D可视化集成;功能基团过滤;分子描述符库;相似图;机器学习等。
3
‘’RDKit | 化学信息学与AI”专栏介分类整理
RDKit安装
Linux(CentOS 7_x64位)系统下安装RDkit
https://blog.csdn.net/u012325865/article/details/78046781
创建独立的Python化学信息学环境
https://blog.csdn.net/u012325865/article/details/80419755
基于RDKit的分子读写及入门
RDKit:基于分子文件输出分子结构
https://blog.csdn.net/u012325865/article/details/82318084
RDKit | 分子处理入门
https://blog.csdn.net/u012325865/article/details/100808051
RDKit | 可视化分子来自于xyz文件
https://blog.csdn.net/u012325865/article/details/102589553
RDKit | 统计分子库中某种元素出现的次数
https://blog.csdn.net/u012325865/article/details/102590698
RDKit | 生物大分子的HELM表示法
https://blog.csdn.net/u012325865/article/details/102037029
基于RDKit的分子指纹与描述符计算
分子指纹
RDKit:化学指纹(Chemical Fingerprinting)
https://blog.csdn.net/u012325865/article/details/81784517
RDKit | 基于SMILES查找化合物的MACCS密钥
https://blog.csdn.net/u012325865/article/details/101697880
RDKit | 基于分子指纹的相似性图
https://blog.csdn.net/u012325865/article/details/102595442
分子描述符
RDKit 描述符计算及可视化
https://blog.csdn.net/u012325865/article/details/78578046
RDKit | 计算化合物描述符
https://blog.csdn.net/u012325865/article/details/101682612
RDKit | 删除方差低的描述符
https://blog.csdn.net/u012325865/article/details/101103957
RDKit | 计算拓扑极性表面积TPSA
https://blog.csdn.net/u012325865/article/details/102552770
RDKit | 基于RDKit描述三维分子形状(3D描述符)
https://blog.csdn.net/u012325865/article/details/102627024
RDKit | 化合物芳香度分子描述符的计算与相关性比较
https://blog.csdn.net/u012325865/article/details/102524904
RDKit | 定量评估类药性(QED)
基于RDKit的构象与RMSD计算
RDKit | 分子的多种构象
https://blog.csdn.net/u012325865/article/details/100832810
RDKit | 分子的力场优化
https://blog.csdn.net/u012325865/article/details/100826445
RDKit | 小分子构象的生成和比对
https://blog.csdn.net/u012325865/article/details/102515273
RDKit:计算不同分子或构象之间的RMSD
https://blog.csdn.net/u012325865/article/details/78578658
基于RDKit的相似性与分子图
RDKit | 基于分子指纹的分子相似性
https://blog.csdn.net/u012325865/article/details/102543725
RDKit | 基于相似图可视化原子贡献
https://blog.csdn.net/u012325865/article/details/102505583
RDKit | 子结构搜索和MCS算法
https://blog.csdn.net/u012325865/article/details/102626236
RDKit | 基于Fraggle算法评估化合物的相似性
https://blog.csdn.net/u012325865/article/details/102795069
RDKit | 化合物亚结构搜索与结果输出
https://blog.csdn.net/u012325865/article/details/102464673
RDKit | 化合物库的相似性分析
https://blog.csdn.net/u012325865/article/details/100916060
RDKit:化合物相似性搜索
https://blog.csdn.net/u012325865/article/details/82153557
RDKit:化合物亚结构(Substructure)搜索
https://blog.csdn.net/u012325865/article/details/82534661
RDKit与药效团
RDKit:可视化药效团(Pharmacophore)
https://blog.csdn.net/u012325865/article/details/92669296
RDKit | 聚类分析与可视化
RDKit | 比较化合物并通过PCA可视化化学空间
https://blog.csdn.net/u012325865/article/details/101693464
RDKit | 基于Ward方法对化合物进行分层聚类
https://blog.csdn.net/u012325865/article/details/101108342
RDKit | 基于PCA探索化学空间
https://blog.csdn.net/u012325865/article/details/100727712
Python脚本:聚类小分子数据集
https://blog.csdn.net/u012325865/article/details/81202123
RDKit与化学反应
RDKit | 基于RDKit绘制化学反应
https://blog.csdn.net/u012325865/article/details/103687862
RDKit | 基于RDKit和SMARTS的化学反应处理
https://blog.csdn.net/u012325865/article/details/103680157
RDKit | 基于RDKit通过SMARTS定义反应模式来生成反应产物
https://blog.csdn.net/u012325865/article/details/103679476
RDKit | 基于RDKit处理化学信息学中的反应方程式
https://blog.csdn.net/u012325865/article/details/103686941
基于RDKit与Python的应用
RDKit | 基于Lipinski规则过滤化合物库
https://blog.csdn.net/u012325865/article/details/102485055
RDKit | 通过评估合成难度(SA Score)筛选化合物
https://blog.csdn.net/u012325865/article/details/102488056
RDKit | 天然产物的相似度评分(NP-likeness)
https://blog.csdn.net/u012325865/article/details/102593918
RDKit | RDKit 中的RECAP进行分子裂解
https://blog.csdn.net/u012325865/article/details/102524938
基于RDKit探索DrugBank
https://blog.csdn.net/u012325865/article/details/82954600
RDKit:化合物骨架分析
https://blog.csdn.net/u012325865/article/details/82082317
RDKit与AI、深度学习和机器学习
RDKit | 基于随机森林的化合物活性二分类模型
https://blog.csdn.net/u012325865/article/details/83830763RDKit | 基于Pytorch和RDKit建立QSAR模型
https://blog.csdn.net/u012325865/article/details/81585844
RDKit | 基于神经网络的溶解度预测和回归分析
https://blog.csdn.net/u012325865/article/details/82725777
RDKit | 基于机器学习的化合物活性预测模型
https://blog.csdn.net/u012325865/article/details/82467904
RDKit:基于RDKit的溶解度预测的机器学习模型
https://blog.csdn.net/u012325865/article/details/82318276
RDKit:基于支持向量回归预测logP
https://blog.csdn.net/u012325865/article/details/88085254
RDkit&mol2vec :基于逻辑回归的靶标抑制剂活性二分类对比
https://blog.csdn.net/u012325865/article/details/92801149
RDKit | 基于随机森林(RF)的机器学习模型预测hERG阻断剂活性
https://blog.csdn.net/u012325865/article/details/89857801
RDKit | 基于Smiles2vec预测化合物物理性质
https://blog.csdn.net/u012325865/article/details/100879193
DeepChem | DeepChem的图卷积特征化器
https://blog.csdn.net/u012325865/article/details/102461491
RDKit | 化合物活性数据的不平衡学习
https://blog.csdn.net/u012325865/article/details/102466647
RDKit | 基于不同描述符和指纹的机器学习模型预测logP
https://blog.csdn.net/u012325865/article/details/102475761
RDKit | 基于RDKit和scikit-learn的KNN模型预测Ames的致突变性
https://blog.csdn.net/u012325865/article/details/102706596
DGL | 基于深度学习框架DGL的分子图初探
DGL | 基于深度图学习框架DGL的分子图生成
DGL | 基于JTNN可视化给定分子的邻居分子
作者&编辑
王建民
DrugAI
长按识别二维码关注我们获取最新消息!
本文为DrugAI原创编译整理,如需转载,请在公众号后台留言。
点击阅读原文查看更多