【学习】 R语言与机器学习学习笔记(1)K-近邻算法
前言
最近在学习数据挖掘,对数据挖掘中的算法比较感兴趣,打算整理分享一下学习情况,顺便利用R来实现一下数据挖掘算法。
数据挖掘里我打算整理的内容有:分类,聚类分析,关联分析,异常检测四大部分。其中分类算法主要介绍:K-近邻算法,决策树算法,朴素贝叶斯算法,支持向量机,神经网络,logistic回归。
写这份学习笔记主要以学校data mining课程的课件为主,会参考一堆的baidu,一堆的google,一堆的blog,一堆的book以及一堆乱七八糟的资料,由于精力有限,恕不能一一列出,如果有认为有侵权行为欢迎与我联系,保证及时删除。
这篇文章是我博客数据挖掘系列的第一篇文章,介绍分类算法中最基本的算法——k近邻算法。
算法一:K-近邻算法
原理及举例
工作原理:我们知道样本集中每一个数据与所属分类的对应关系,输入没有标签的新数据后,将新数据与训练集的数据对应特征进行比较,找出“距离”最近的k(通常k<20)数据,选择这k个数据中出现最多的分类作为新数据的分类。
算法描述:
(1) 计算已知类别数据及中的点与当前点的距离;
(2) 按距离递增次序排序
(3) 选取与当前点距离最小的k个点
(4) 确定前K个点所在类别出现的频率
(5) 返回频率最高的类别作为当前类别的预测
距离计算方法有”euclidean”(欧氏距离),”minkowski”(明科夫斯基距离), “maximum”(切比雪夫距离), “manhattan”(绝对值距离),”canberra”(兰式距离), 或 “minkowski”(马氏距离)等.
分析学的知识告诉我们Rn上范数之间是等价的,所以我们也没必要太过纠结选谁,毕竟范数之间都是可以相互控制的。
这里我们使用最常见欧氏距离作为衡量标准,以鸢尾花数据集为例来说明K-近邻算法:
鸢尾花数据集包含150个数据,测量变量为花瓣,花萼的长度与宽度,分类变量为setosa, versicolor, 和 virginica。
准备数据:
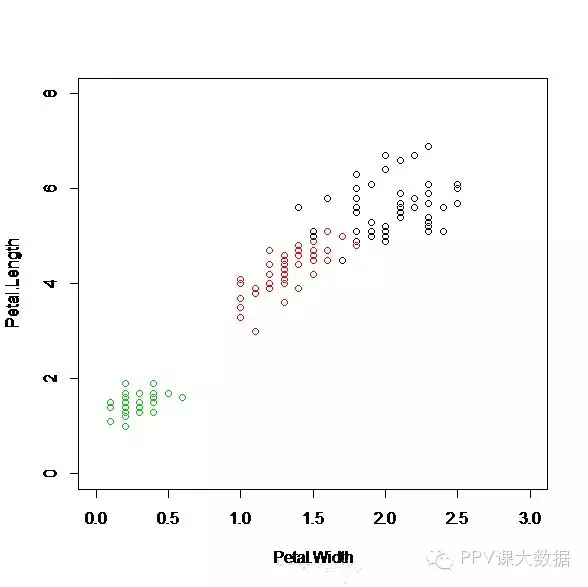
为了了解数据,我们先通过作图分析,相关分析来看看数据分类指标的合理性,这一点十分重要,有助于减少分类指标中的噪声。
从上图可以看出,我们通过这2个变量大致是可以把鸢尾花分类的,也就是说分类的特征变量选择是合理的,(同理可以分析另外2个,分类效果不如这两个,但大致上还是能区分的)当然我们也可以选择计算相关系数来看特征变量的合理性。
我们很容易发现,数值差最大的属性对距离的影响最大,所以在特征值等权重的假定下,我们先得归一化特征值,计算公式为:
Newvalue=(oldvalue-min)/(max-min)
(注:网友@ reus123指出归一化的提法不太合适,也的确如此,我们这里将本文的“归一化”理解为一种“标准化”就好,感谢@ reus123的指正)
R代码:
[plain] view plaincopyprint?![]()
![]()
-
autonorm<-function(data){
-
min<-min(data)
-
max<-max(data)
-
for(i in 1:length(data))
-
data[i]<-(data[i]-min)/(max-min)
-
return(data)
-
}
-
data<-apply(as.matrix(iris[,1:4]),2,autonorm)
(之前的程序有误,已更正,感谢@ reus123的指正)
得到了归一化后的数据集,下面计算距离。我们在这里取三个数据作为验证集来看看分类的效果,首先将验证集归一化:
[plain] view plaincopyprint?![]()
![]()
-
x<-iris[13,1:4]
-
y<-iris[79,1:4]
-
z<-iris[100,1:4]
-
x<-(x-apply(iris[c(-13,-79,-100),1:4],2,min))/(apply(iris[c(-13,-79,-100),1:4],2,max)-apply(iris[c(-13,-79,-100),1:4],2,min))
-
y<-(y-apply(iris[c(-13,-79,-100),1:4],2,min))/(apply(iris[c(-13,-79,-100),1:4],2,max)-apply(iris[c(-13,-79,-100),1:4],2,min))
-
z<-(z-apply(iris[c(-13,-79,-100),1:4],2,min))/(apply(iris[c(-13,-79,-100),1:4],2,max)-apply(iris[c(-13,-79,-100),1:4],2,min))
计算距离,仅以Z为例,运行代码:(k取5)
[plain] view plaincopyprint?![]()
![]()
-
dis<-rep(0,length(data[,1]))
-
for(iin 1:length(data[,1]))
-
dis[i]<-sqrt(sum((z-data[i,1:4])^2))
-
table(data[order(dis)[1:5],5])
从x,y,z的输出结果可以看到,分类完全正确,没有错误分类。
值得一提的是,我们用同样的办法计算K=3时的情形,会发现没有出现误分类。这也就引出了一个值得思考的问题:k应该如何选取?k过小,噪声对分类的影响就会变得非常大,K过大,那么包含错误就理所当然,误分类也不足为奇。虽然这里我们对K的取值并未进行讨论,但在实际中,我们应该通过交叉验证的办法来确定k值。
R语言内置函数kknn简介
R语言里的kknn包也可以实现最邻近算法——使用kknn函数。
kknn(formula = formula(train),train, test, na.action = na.omit(),
k= 7, distance = 2, kernel = “optimal”, ykernel = NULL, scale=TRUE,
contrasts= c(‘unordered’ = “contr.dummy”, ordered =”contr.ordinal”))
参数解释:
formula 一个回归模型,具体为:分类变量~特征变量
train 训练集
test 测试集
na.action 缺失值处理,默认为去掉缺失值
k k值选择,默认为7
distance 这个是明科夫斯基距离,p=2时为欧氏距离
其他参数 略
上面的鸢尾花例子使用kknn包可以实现(k=5):
[plain] view plaincopyprint?![]()
![]()
-
library(kknn)
-
-
data(iris)
-
m <- dim(iris)[1]
-
val <- sample(1:m, size =round(m/3), replace = FALSE,
-
prob= rep(1/m, m))
-
iris.learn <- iris[-val,]
-
iris.valid <- iris[val,]
-
iris.kknn <- kknn(Species~.,iris.learn, iris.valid, distance = 5,
-
kernel= “triangular”)
-
summary(iris.kknn)
-
fit <- fitted(iris.kknn)
-
table(iris.valid$Species, fit)
这里我们的训练集选取更随机化,得到结果是:
fit
setosa versicolor virginica
setosa 12 0 0
versicolor 0 22 0
virginica 0 0 16
分类完全正确。
应用举例:手写数字识别
下面我们来做一个规模大一些的数据处理,利用k-近邻实现一下数字的模式识别。这个例子来自《机器学习实战》,具体数据集已上传至百度云盘(点击这里下载)。数据为了简单起见,仅提供0~9,10个数字的识别。需要识别的数字你可以看做是被图像处理软件处理为了32*32的黑白图像。尽管文本格式储存图片不能够有效地利用存储空间,但是为了方便理解还是提供了这个文本版的图片数据。至于图像版本的数据,你可以找到《手写数字的光学识别》一文(登载于2010年的UCI机器学习资料库中)的数据集合,并下载它。
完整的R实现:
[plain] view plaincopyprint?![]()
![]()
-
setwd(“D:/R/data/digits/trainingDigits”)
-
names<-list.files(“D:/R/data/digits/trainingDigits”)
-
data<-paste(“train”,1:1934,sep=””)
-
for(i in 1:length(names))
-
assign(data[i],as.matrix(read.fwf(names[i],widths=rep(1,32))))
-
-
-
dis<-function(datatest,datatrain,len){
-
distance<-rep(0,len)
-
for(i in 1:len)
-
distance[i]<-sqrt(sum((get(datatest)-get(datatrain[i]))^2))
-
return((distance))
-
}
-
-
judge<-function(test,data,names){
-
index<-rep(0:9,c(189,198,195,199,186,187,195,201,180,204))
-
di<-rep(0,1934)
-
di[1:1934]<-dis(test,data,length(names))
-
return(names(which.max(table(index[order(di)[1:5]]))))
-
}
-
-
setwd(“D:/R/data/digits/testDigits”)
-
name<-list.files(“D:/R/data/digits/testDigits”)
-
test<-paste(“test”,1:946,sep=””)
-
for(i in 1:length(name))
-
assign(test[i],as.matrix(read.fwf(name[i],widths=rep(1,32))))
-
index1<-rep(0:9,c(87,97,92,85,114,108,87,96,91,89))
-
-
error<-0
-
for(i in 1:946){
-
if(judge(test[i],data,names)!=index1[i])
-
error<-error+1
-
}
运行结果:
>error
[1]19
>19/946
[1]0.02008457
也就是说,使用5-近邻算法,误差率为2%,属于一个可以接受的范围。
这里由于本人没有找到较好的批量导入数据的办法,所以代码有些复杂,也出现了hardcode和magicnumber的毛病,但是泛化也不是那么的复杂,所以也没再做更进一步的改进。希望读者告诉我如何解决R里导入批量数据的方法。
其中有两个函数是我在之前的博客中没有使用过的,现在简单介绍如下:
赋值函数assign:
assign(“x”, c(10.4, 5.6, 3.1, 6.4, 21.7)) 与x <- c(10.4,5.6, 3.1, 6.4, 21.7)等价
读取赋值函数的函数get:
a<- 1:4
assign(“a[1]“,2)
a[1]== 2 #FALSE
get(“a[1]“) == 2 #TRUE
在R中,我没有找到求众数的函数,简单编写了一个names(which.max(table(index[order(di)[1:5]]))),这个函数有两个众数时会输出两个,所以K近邻为了保证多数投票法有用,麻烦仔细选择合理的k值。
这里我在做训练集时并没有选择k值得过程(因为这个算法实在是太慢了,没有那个耐心)
实际使用这个算法,执行效率相当的低下,每个距离的计算包含了1024个维度的浮点运算,总计900多次,还要为测试向量准备2M的存储空间。所以k决策树是你需要进一步了解的。
K决策树的种类也有不少,比如kd树,但是他们的问题就是k的选取总是一个麻烦的过程,kd树找最近邻是十分高效的,但是找k近邻,删除结点重新建树还是比较麻烦的。
Further reading:
JULY大神的《从K近邻算法、距离度量谈到KD树、SIFT+BBF算法》