去东方,最好用的在线GO富集分析工具
微信公众号:http://blog.genesino.com
GeneOntology富集分析是是高通量数据分析的标配,不管是转录组、甲基化、ChIP-seq还是重测序,都会用到对一个或多个集合的基因进行功能富集分析。分析结果可以指示这个集合的基因具有什么样的功能偏好性,进而据此判断相应的生物学意义。
GOEAST,去东方
今天向大家推荐一款,中科院遗传所王秀杰老师组2008年发表的在线工具GOEAST,访问网站是http://omicslab.genetics.ac.cn/GOEAST/index.php。GOEAST自发表以来一直在更新维护,并且每周与Gene Ontology网站同步GO注释数据库,保证分析结果的时效性和准确性。文章从发表至今,被引用504次,总访问量近140万次,总使用量近87万次。
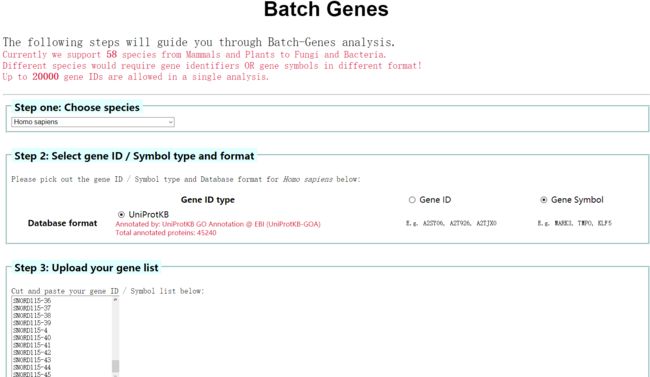
这款工具最初是为芯片研究开发的,有一些芯片分析特有的功能。但这次我们用到的是GOEAST的Advance-Btach-Genes功能。同时GOEAST还支持多个GO富集分析结果的比较和自定义背景数据集。
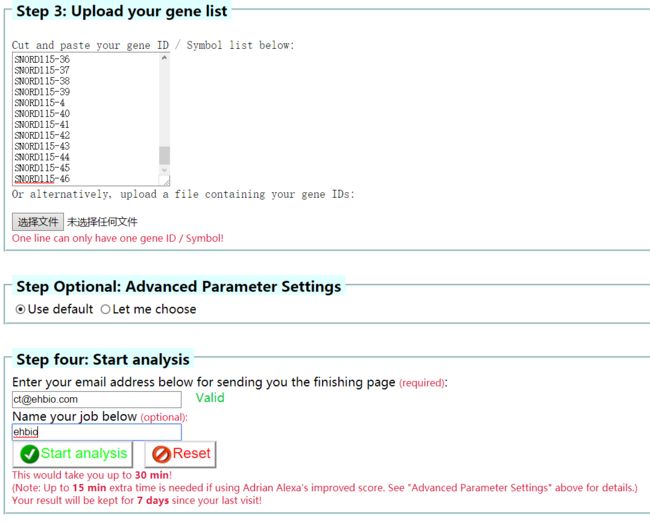
GOEAST使用很简单,用户只需提交常见的一组Gene Symbol或Gene ID,选择对应的物种 (支持动植物真菌细菌等59个物种),等待平均10分钟即可获得结果。也可以输入邮箱,给自己的提交取一个名字,把多组基因分别上传,等着去邮箱查收结果就好了。
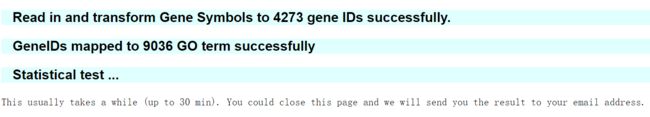
点击提交后,出现如下跳转页面。这页会自动更新程序运行信息,通常10分钟左右即可获得结果。如果你填写了邮箱,也可以关掉此页面,在邮件中打开即可。
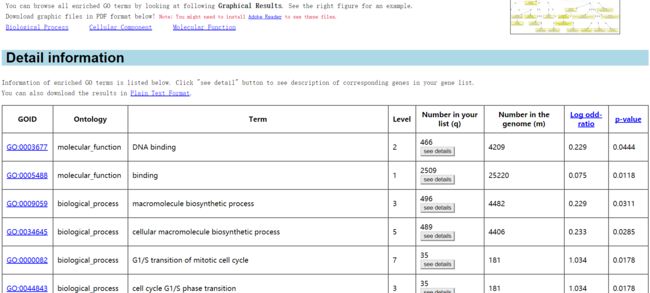
结果页如下所示,包含3个PDF图(蓝色带下划线的Biological Process, Cellular Component, Molecular Function)和一个富集分析表格。
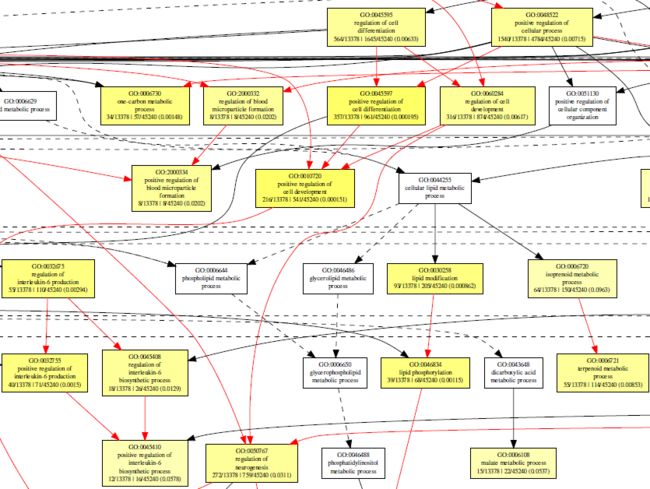
展示的富集分析图为常见的树形图样式,每个方格代表一个GO条目,连线代表它们之间的层级关系,颜色越深表示富集越显著。
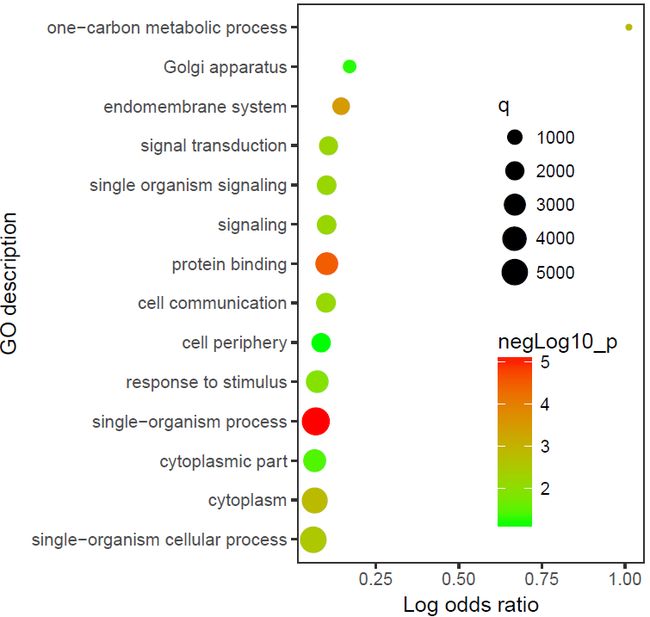
上述的树图,能反应富集的GO条目之间的层级关系,但图会较大,看着不太方便。通常会使用泡泡图来展示其中一部分最为富集的结果。这时就需要用到刚才生成的表格数据,点击Plain Text Format下载表格数据,格式如下:
选择前面15行存储为文件goeast.txt作为测试 (注意是tab键分割的txt文件),用R语言学习 - 富集分析泡泡图 (文末有彩蛋)和富集分析DotPlot,可以服中提到的方法绘图 (两篇文章结合着看,效果更佳)。
sp_enrichmentPlot.sh -f goeast.txt -o log_odds_ratio -T numeric -v Term -c p -s q -l p -a 12 -x "Log odds ratio" -y "GO description" -w 12 -P "c(0.75,0.45)"支持的物种列表
-- Primates --
Homo sapiens
-- Mammals --
Bos taurus
Canis lupus familiaris
Sus scrofa
Mus musculus
Rattus norvegicus
-- Other Vertebrates --
Danio rerio
Gallus gallus
Xenopus laevis
-- Invertebrates --
Caenorhabditis elegans
Dictyostelium discoideum
Drosophila melanogaster
Plasmodium falciparum
Plasmodium falciparum 3D7
Trypanosoma brucei TREU927
-- Plants & Fungi --
Arabidopsis thaliana
Aspergillus fumigatus
Aspergillus niger
Aspergillus oryzae
Candida albicans
Candida albicans SC5314
Candida dubliniensis
Candida glabrata
Candida glabrata CBS 138
Candida parapsilosis
Candida parapsilosis CDC317
Emericella nidulans
Magnaporthe grisea
Magnaporthe oryzae 70-15
Oryza sativa
Oryza sativa Indica Group
Oryza sativa Japonica Group
Saccharomyces cerevisiae
Schizosaccharomyces pombe
-- Bacteria --
Anaplasma phagocytophilum HZ
Bacillus anthracis
Bacillus anthracis str. Ames
Campylobacter jejuni RM1221
Carboxydothermus hydrogenoformans Z-2901
Clostridium perfringens ATCC 13124
Colwellia psychrerythraea 34H
Coxiella burnetii RSA 493
Dehalococcoides ethenogenes 195
Ehrlichia chaffeensis str. Arkansas
Escherichia coli K-12
Geobacter sulfurreducens PCA
Hyphomonas neptunium ATCC 15444
Listeria monocytogenes serotype 4b str. F2365
Methylococcus capsulatus str. Bath
Mycobacterium tuberculosis
Pseudomonas aeruginosa PAO1
Pseudomonas protegens Pf-5
Pseudomonas syringae pv. phaseolicola 1448A
Pseudomonas syringae pv. tomato str. DC3000
Ruegeria pomeroyi DSS-3
Shewanella oneidensis MR-1
Vibrio cholerae O1 biovar El Tor
Vibrio cholerae O1 biovar El Tor str. N16961命令行下的基因富集分析
如果你是在命令行下做富集分析,出门左拐找clusterprofiler。