异常检测——Anomaly Detection
目录
- 1.问题来源
- 2.应用
- 3.分类
- 3.1 method1:With Classifier
- 3.2 method 2:Gaussian distribution
- 3.2.1 问题阐述
- 实现代码
- 3.3 method 3: Auto-Encoder
- 3.4 method 4: PCA
- 3.5 method5:Isolation Forest
- 3.6 GAN anomaly detection
- 4.异常侦测系统(模型)的评价指标
- 自己定义
- AUC
- 5.遇到检测错误的点
1.问题来源
2.应用
难点:异常点难以收集,无法穷举。我只能收集到正常情况的训练数据。
3.分类
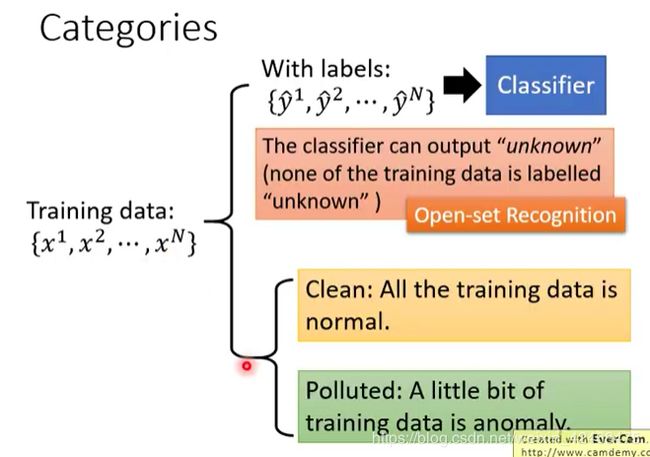
3.1 method1:With Classifier
不能被分类器 高置信地分类就当作异常点。
1.对每个非异常成员打标训练。
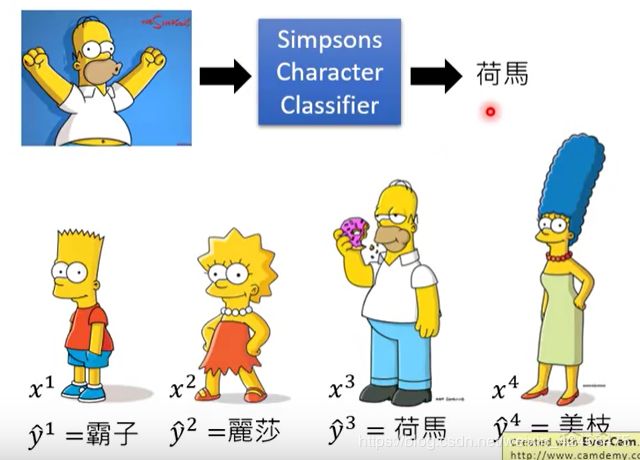
2.查看每一条数据 预测矢量 的最大值 c(x),比较异常点和非异常点的c(x)值有没有明显的分界点。

但是总有一些异常点会被当作正常点
3.用 偏离集(dev set) 评估“异常检测”模型的 “异常阈值”λ ,要求偏离集有很多异常点和非异常点。

3.2 method 2:Gaussian distribution
这种情况下没有标签,从数据每一维特征的分布处理。
3.2.1 问题阐述
还是要寻找一个阈值,分布大于某一阈值为正常点,小于某一阈值为异常点。

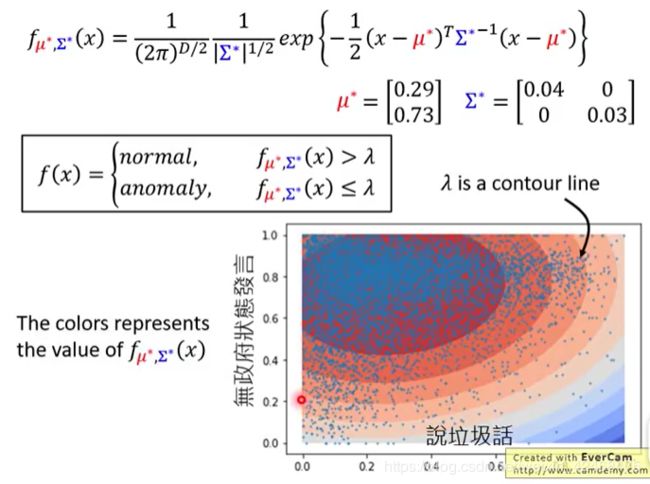
计算每一个样本的分布概率,累乘,寻找能让 L ( θ ) L(\theta) L(θ)达到最大值的参数 θ \theta θ。

f θ ( x ) f_\theta(x) fθ(x)具体展开:
实际上我们是要找 一组 μ \mu μ 和 Σ \Sigma Σ,使得 L ( θ ) L(\theta) L(θ)达到最大值。
为什么用高斯分布? :常见~
找到最佳参数后,把具体要检测的点带入,即可得到 f(x) 。D表述数据维度。
实现代码
1.首先要得到两个参数 μ \mu μ 和 Σ \Sigma Σ:

def getGaussianParams(X, useMultivariate):
"""
The input X is the dataset with each n-dimensional data point in one row
The output is an n-dimensional vector mu, the mean of the data set
the variances sigma^2, an n x 1 vector 或者是(n,n)矩阵,if你使用了多元高斯函数
作业这里求样本方差除的是 m 而不是 m - 1,实际上效果差不了多少。
"""
mu = X.mean(axis=0)
if useMultivariate:
sigma2 = ((X-mu).T @ (X-mu)) / len(X)
else:
sigma2 = X.var(axis=0, ddof=0) # 样本方差
return mu, sigma2
2.计算每条数据的可能性
def gaussian(X, mu, sigma2):
'''
mu, sigma2参数已经决定了一个高斯分布模型
因为原始模型就是多元高斯模型在sigma2上是对角矩阵而已,所以如下:
If Sigma2 is a matrix, it is treated as the covariance matrix.
If Sigma2 is a vector, it is treated as the sigma^2 values of the variances
in each dimension (a diagonal covariance matrix)
output:
一个(m, )维向量,包含每个样本的概率值。
'''
# 如果想用矩阵相乘求解exp()中的项,一定要注意维度的变换。
# 事实上我们只需要取对角线上的元素即可。(类似于方差而不是想要协方差)
# 最后得到一个(m,)的向量,包含每个样本的概率,而不是想要一个(m,m)的矩阵
# 注意这里,当矩阵过大时,numpy矩阵相乘会出现内存错误。例如9万维的矩阵。所以画图时不能生成太多数据~!
# n = len(mu)
# if np.ndim(sigma2) == 1:
# sigma2 = np.diag(sigma2)
# X = X - mu
# p1 = np.power(2 * np.pi, -n/2)*np.sqrt(np.linalg.det(sigma2))
# e = np.diag([email protected](sigma2)@X.T) # 取对角元素,类似与方差,而不要协方差
# p2 = np.exp(-.5*e)
# return p1 * p2
# 下面是不利用矩阵的解法,相当于把每行数据输入进去,不会出现内存错误。
m, n = X.shape
if np.ndim(sigma2) == 1:
sigma2 = np.diag(sigma2)
norm = 1./(np.power((2*np.pi), n/2)*np.sqrt(np.linalg.det(sigma2)))
exp = np.zeros((m,1))
for row in range(m):
xrow = X[row]
exp[row] = np.exp(-0.5*((xrow-mu).T).dot(np.linalg.inv(sigma2)).dot(xrow-mu))
return norm*exp
3.3 method 3: Auto-Encoder
只有正常的点会被较好地还原,异常点不会被很好地还原。
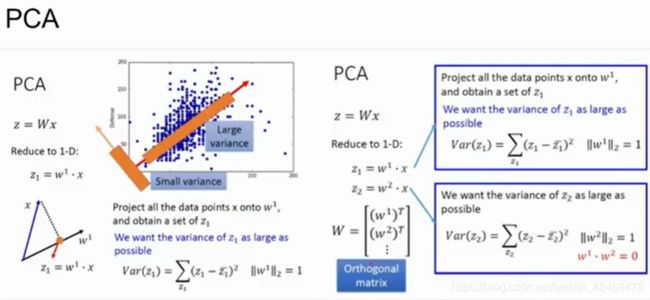
3.4 method 4: PCA
把多维数据(正常点+异常点)投影成低维,然后再把低维逆投影成原来的维度,正常点会和异常点还原的程度会不一样。
降到几维比较好?——用precision-recall曲线,这里来看是27比较好,因为阴影面积更加大,用roc曲线也可以判断~

3.5 method5:Isolation Forest
https://blog.csdn.net/bbbeoy/article/details/80300941?ops_request_misc=%257B%2522request%255Fid%2522%253A%2522159179723219724843318563%2522%252C%2522scm%2522%253A%252220140713.130102334…%2522%257D&request_id=159179723219724843318563&biz_id=0&utm_medium=distribute.pc_search_result.none-task-blog-2allsobaiduend~default-3-80300941.first_rank_ecpm_v3_pc_rank_v2&utm_term=%E5%BC%82%E5%B8%B8%E6%A3%80%E6%B5%8B%E7%AE%97%E6%B3%95
- F. T. Liu, K. M. Ting and Z. H. Zhou,Isolation-based Anomaly Detection,TKDD,2011
实现代码:
https://scikit-learn.org/dev/modules/generated/sklearn.ensemble.IsolationForest.html
3.6 GAN anomaly detection
4.异常侦测系统(模型)的评价指标
自己定义
因为样本不平衡,所以不建议用accuracy,建议对照precision-recall表,比较假阳性和假阴性的重要程度,分别给予不同的权重,计算新的损失函数。
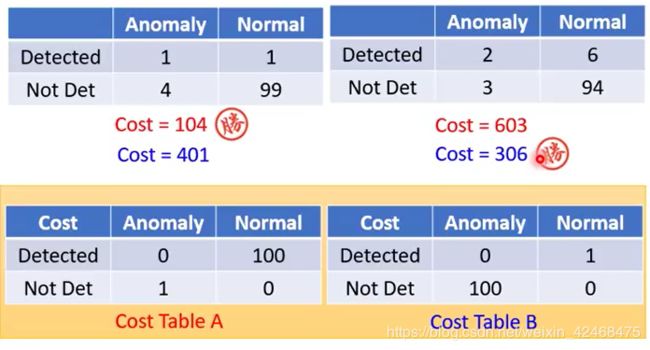
AUC
可以避免测试样本分布不平均的情况,使得假阳性和假阴性一样重要。
5.遇到检测错误的点
容易出现“意外”情况:

如何解决:
多找些异常点(通过generative model),让模型对异常点产出很低的置信度。