如何从 PDB 文件中提取氨基酸序列
蛋白质数据库Protein Data Bank(PDB)是一个包含蛋白质、核酸等生物大分子的结构数据的数据库,网址是http://www.rcsb.org。PDB可以经由网络免费访问,是结构生物学研究中的重要资源。为了确保PDB资料的完备与权威,各个主要的科学杂志、基金组织会要求科学家将自己的研究成果提交给PDB。PDB数据库存储结构数据的文件是PDB文件,每一个蛋白质或核酸都对应着一个编号,即PDBID, 文件的扩展名为.pdb。PDB文件可以由各种3D结构显示软件打开,比如pymol,Swiss-PDB viewer,VMD等。PDB文件里面的信息是有严格的格式的。各行数据,如标识,原子名,原子序号,残基名称,残基序号等,不仅要按照严格的顺序书写,而且各项所占的空符串长度,及其所处的各行的位置都是严格规定。更多信息请查看PDB文件中信息的格式。
写在前面的话:
本人是一枚生物学的学生,由于对生物信息学特别感兴趣,于是想自学生物信息学(新手莫怪)。了解到生物信息学要有编程基础,尤其是要会一门编程语言,例如:R语言、Python、Perl等,还要熟悉Linux系统,作为生信小白,听说Python挺简单的,于是就自学了Python,花了两天时间了解了Python的基础语法后,今天想做个练习题试试手(实践是检验真理的唯一标准)。
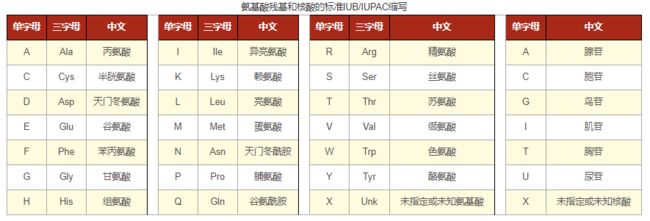
氨基酸残基与核酸缩写
众所周知氨基酸标准缩写是三个字母,而这次我们所用的PDB文件里面也是用三个字母表示的氨基酸,我们需要把它转化为常见的fasta格式的氨基酸序列,所以我们首先应该用字典将三个字母的氨基酸转换为单字母代码。
字典的键是氨基酸三字母代码, 值是对应的氨基酸的单字母代码。使程序用该字典从 PDB 文件的 SEQRES 行读取残基名称 (以三字母代码的形式) (文件 1TDL 中) , 然后将它们转换为一个字母代码,连接这些字母,就得到了蛋白质序列。最后,将该序列以 FASTA 的格式打印出来。
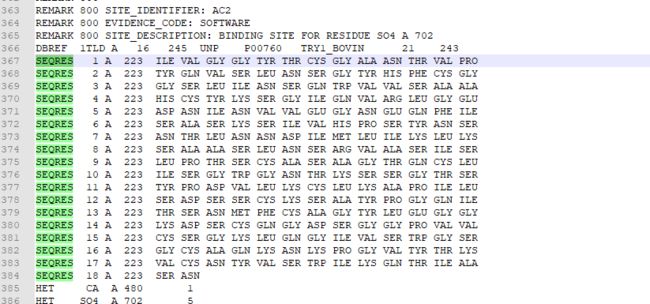
1TLD. pdb文件如图所示:
要使用该程序,需要找到 PDB文档 ,下载并保存 PDB文件。 此例使用了 PDB文件 1TLD. pdb,其中有牛自膜蛋白酶在1. 5A分辨率下的晶体结构,该 PDB文件 的 SEQ阻S行列出了实验用到的蛋白质序列。每个 SEQRES行可分为 17 列。 第一列包含关键 字"SEQRES"; 第二列有序列的行号(从 1 开始) ;第三列是链 ID; 第四列是组成序列的残基数; 从第五列到最后一列是(三字母代码形式的)残基。列之间由一个空格进行分隔。
第一步:定义字典
aa_codes = {
'ALA':'A','CYS':'C','ASP':'D','GLU':'E',
'PHE':'F','GLY':'G','HIS':'H','LYS':'K',
'ILE':'I','LEU':'L','MET':'M','ASN':'N',
'PRO':'P','GLN':'Q','ARG':'R','SER':'S',
'THR':'T','VAL':'V','TYR':'Y','TRP':'W'}
第二步:读取文件
首先读取文件,以"SEQRES"定位到位置,并.split()去掉空格
seq = ''
for line in open("C:/Desktop/1tld.pdb"):
if line[0:6] =="SEQRES":
columns = line.split()
for resname in columns[4:]:
seq = seq + aa_codes[resname]
第三步:打印输出*
i = 0
print(">1TLD")
while i < len(seq) :
print(seq[i:i+64])
i =i+64
最后附上完整代码:
aa_codes = {
'ALA':'A','CYS':'C','ASP':'D','GLU':'E',
'PHE':'F','GLY':'G','HIS':'H','LYS':'K',
'ILE':'I','LEU':'L','MET':'M','ASN':'N',
'PRO':'P','GLN':'Q','ARG':'R','SER':'S',
'THR':'T','VAL':'V','TYR':'Y','TRP':'W'
}
seq = ''
for line in open("C:/Desktop/1tld.pdb"):
if line[0:6] =="SEQRES":
columns = line.split()
for resname in columns[4:]:
seq = seq + aa_codes[resname]
i = 0
print(">1TLD")
while i < len(seq) :
print(seq[i:i+64])
i =i+64
日常结尾:
虽然这是个小小的计算程序,但对于初学者的我来说每一次对原代码的升级改造,哪怕是读懂后的注释都感觉是一次进步提升,总之代码虽小,动手最重要!希望更多学习Python的爱好者不要像我一样眼高手低,学习编程就是要,思考,敲码,思考,敲码,敲码,再敲码!