写在写在前面的前面
或许,没有人知道TBtools到底是什么?能干啥。
但是看完这个推文,或许你就知道了其中的一部分。
写在前面
TBtools对外开放两年多,不时会有熟悉的不熟悉的人与我聊到TBtools。TBtools在每个人的认知上,或许都不一样。
有的人觉得TBtools就是耽误了他们所谓的生物信息学习。
有的人说,TBtools保了他们毕业。
也有的人说,TBtools帮助了他发了文章
更或者,TBtools。。。
TBtools是一个序列提取小工具?
三年前开始写TBtools。功能很简单,主要是做做序列提取,也做了BlastWrapper(当时并不稳健)。目的很纯粹,课题组的人不会找我提取序列,也可以直接Blast到转录组找序列。那时候接触TBtools的朋友,或许都这么认为。
当然,后来我对这方面进行了各种增强,也保证了其现在的稳健性。一个输入窗口,支持不同的输入,无论是提取序列全长,还是提取序列区段;不仅支持ID提取,还支持ID子串匹配...
此外,也增加了基于gff3进行序列提取的功能,比如提取所有序列的全长CDS,全长EXON,甚至是可以批量一次性提取一个物种的所有启动子序列。
除了Blast Wrapper,可能还需要调用外部程序的,那么是muscle(主要是我实现的NW算法运行效率一般,用到多序列比对,就不提了)
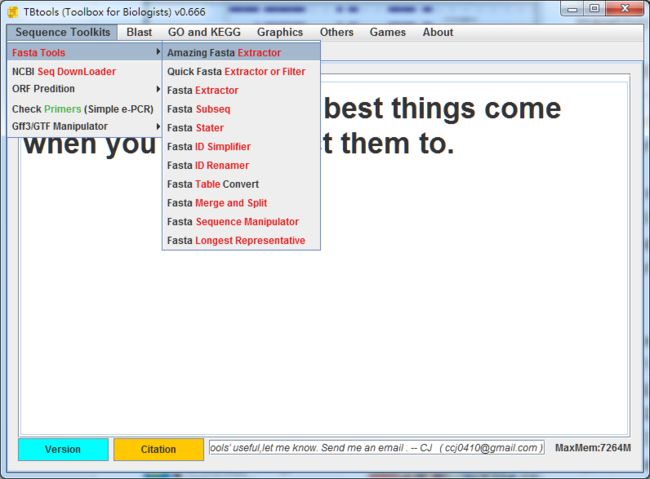
可以从一个子菜单看到
功能相对丰富,包括
- 序列提取
- 序列统计
- ID简化
- 序列重命名
- Fasta格式与Table格式相互转换
- Fasta文件的合并与分隔
- 序列的反向互补,DNA/RNA转换,大小写,格式化
- 基于ID模式的基因最长代表转录本提取
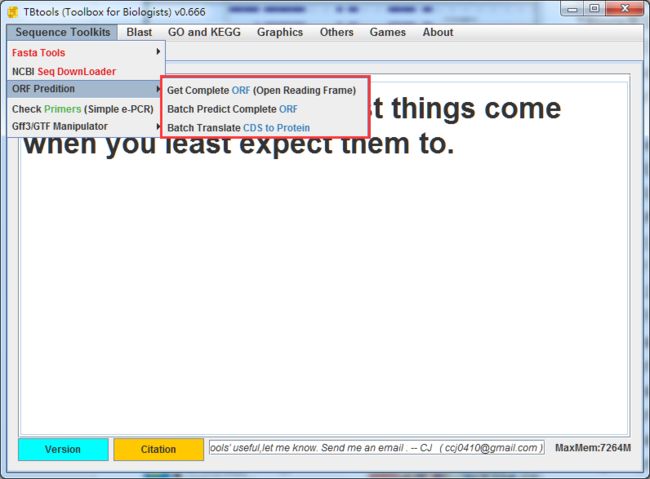
TBtools做核酸到蛋白序列的翻译?
- 从某一条序列中预测全长ORF
- 批量从一个数据集合中预测最长全长ORF
- CDS序列批量转换为蛋白序列
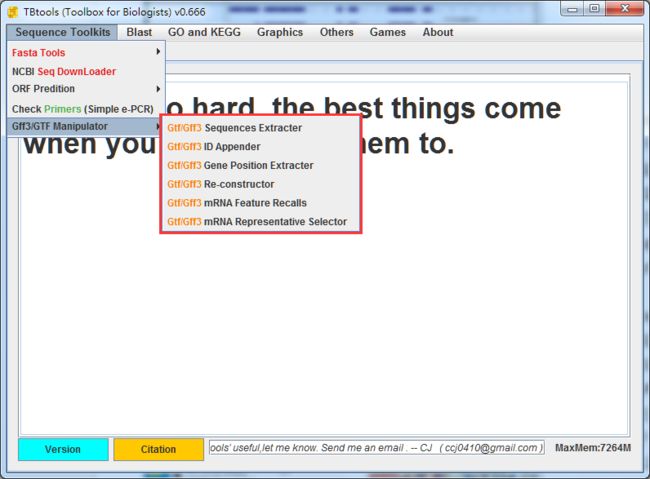
TBtools是一个Gff3/GTF基因结构注释文件操作工具?
- 基于GFF/GTF文件,从基因组提取序列
- 批量修改基因和染色体ID,使不同物种的合并分析不会有ID冲突
- 提取所有基因信息
- 基于mRNA序列和基因组序列,直接重构基因结构信息,输出gff3文件
- 基于不完全的gff和gtf文件,重构mRNA feature
- 基于gff3/gtf文件,提取所有基因的最长代表转录本序列
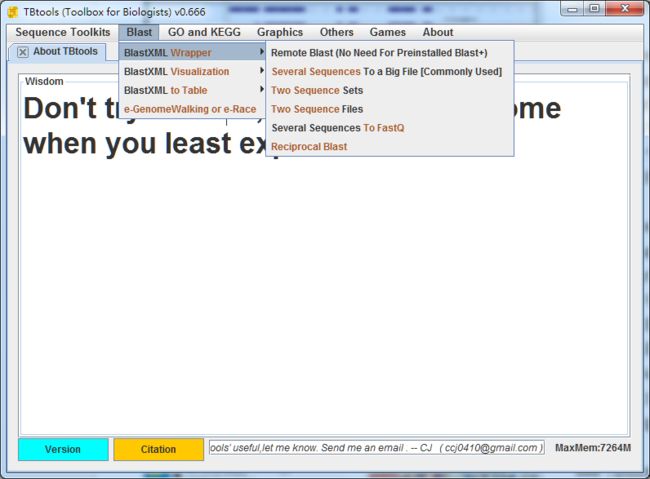
TBtools降低本地Blast操作的时间损耗?
无论是两条序列的直接比较,还是两个序列文件,甚至是两个基因组的两个指定区间的Blast,TBtools中都已经提供了GUI;不仅于此,四种可视化方式,常常能满足大多数人的需求。
比如
TBtools是一个GO和KEGG分析(富集)小工具?
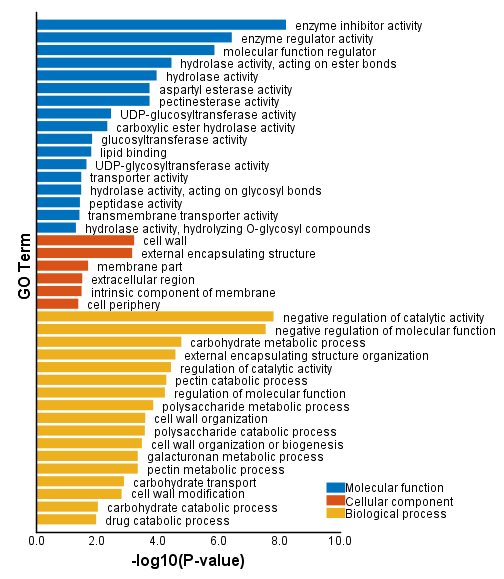
后来,由于Blast2GO太慢了。基于IDmapping的逻辑,我大体写了一个GO注释的功能。当然,更重要的或许是直接写了GO和KEGG富集分析的功能。所以后来,也有不少做非模式生物的朋友认为,TBtools事实就做这个事情。其中也包括一些可视化,比如GO Level2的可视化。后来,我也写了一些富集结果的可视化。
TBtools是一个画图的小工具?
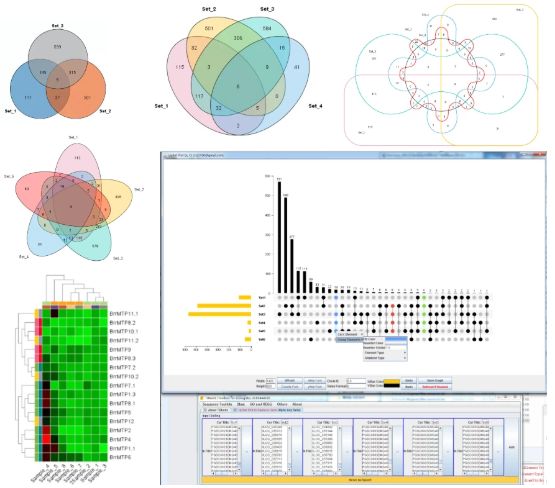
慢慢地,我发现,网页版工具,如Venny,明明是很小的韦恩图绘制功能,网络太差,等待缓冲总是占用了我太多的时间。应该本地化。所以我索性写了一个最高支持六组的Venn图工具。当然,也有后来的UpsetPlot工具。
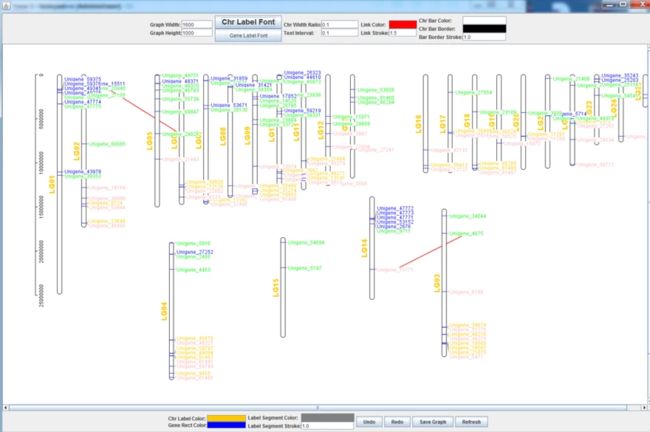
基因展示在染色体上的,类似MapChart的工具等。
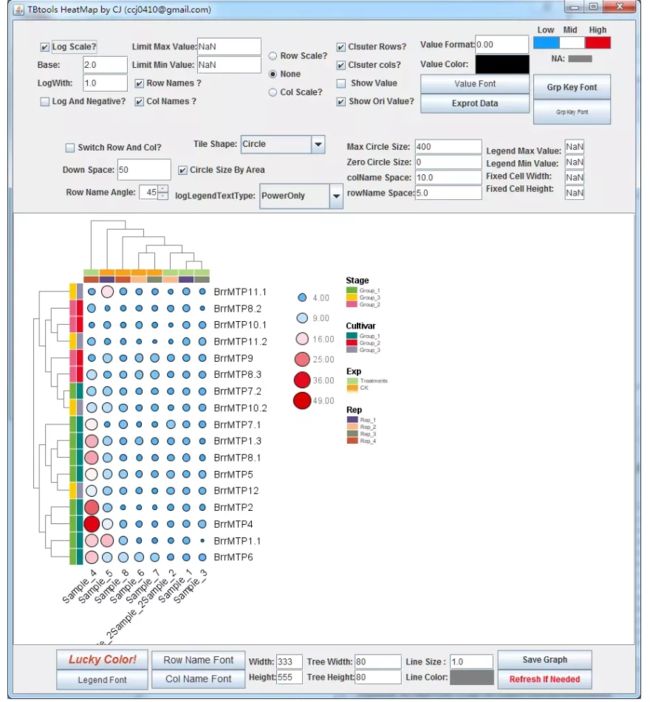
此外也由于一些工具,如热图绘制上,我觉得用起来真的不顺手。或者参数太少,或者不容易调整各种细节。所以我也写了热图工具。
所以,或许确实有的朋友就觉得,你这工具,就是一个画图工具包。
Venn图与UpSetPlot
热图及其变型
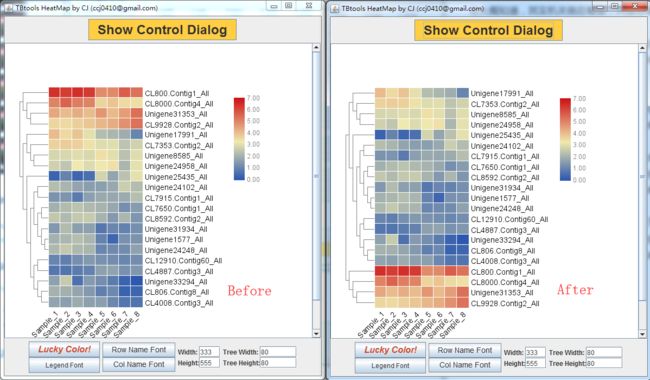
甚至是,掰弯的热图,可以一次展示大量数据的热图
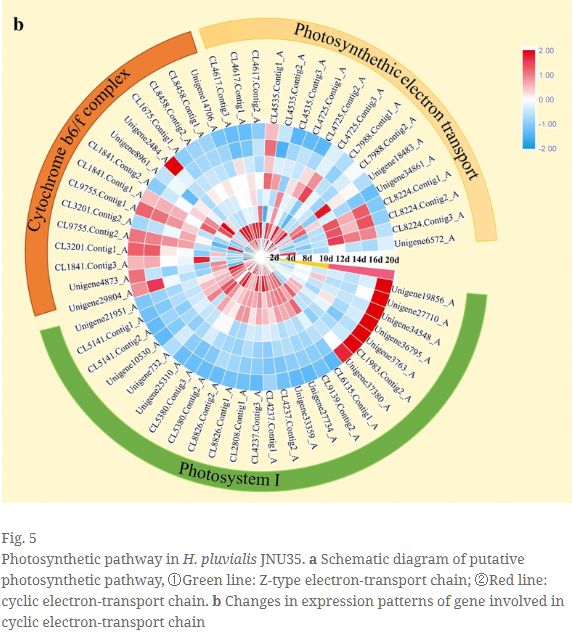
图片来自黄博士的文章( https://biotechnologyforbiofuels.biomedcentral.com/articles/10.1186/s13068-019-1355-5)
不仅可以掰弯,你还可以调整聚类位置
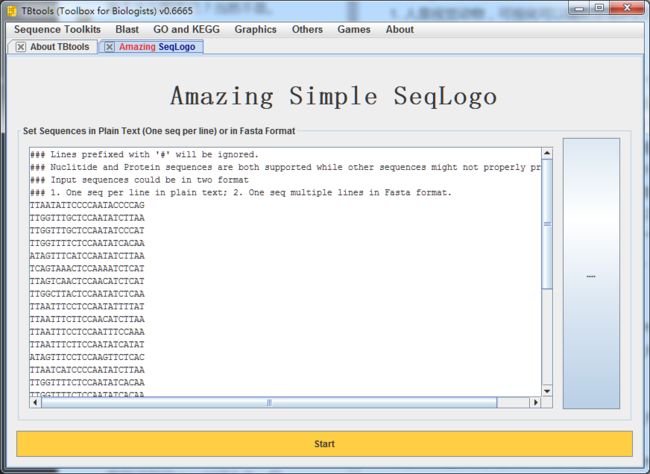
当然,你还可以直接绘制SeqLogo
TBtools是一个基因家族分析工具?
基于前面我发送过的推文,总的来说,有了TBtools,所有人无需任何一行命令,也不需要Linux或者虚拟机操作,可以完成常见的基因家族分析。
大体包括的工具有:
- 基于GFF3的序列提取
- Blast Wrapper
- MEME结果的可视化
- 结构域分析信息的可视化
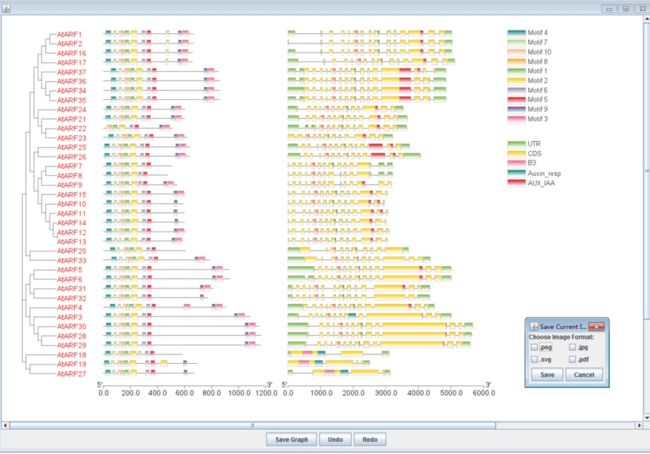
- 基因结构的可视化
- 以上三者和进化树的一次性可视化
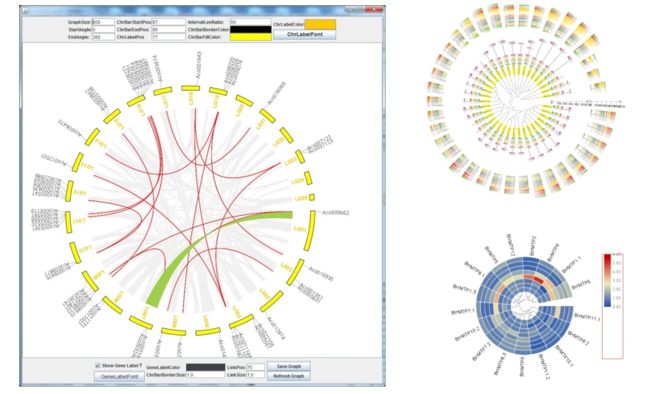
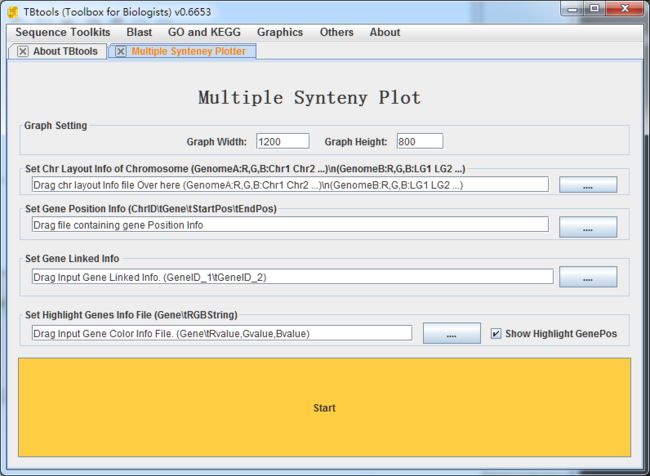
- 多基因组-共线性分析及可视化
似乎,有一些培训机构提供的线上线下的基因家族分析培训,需要使用的各种虚拟机,Linux,命令,脚本,统统都可以扔掉。常见的基因家族分析项目,可能只需要TBtools就完全足够了。
为此,慢慢地开始有朋友给TBtools下了定义:TBtools是一个基因家族分析工具包。在我看来,事实上,这些朋友对TBtools有很大的误解。我从来就没想过写一个基因家族分析工具。不是因为TBtools要做一个基因家族分析工具,而是因为基因家族分析本身就是所有人都需要,都懂的生物数据分析的基本技能。我只是简化这些技能的实现。
正如下面,我所写的TBtools中,或许很多人想想不到功能一样。
与其说TBtools是你以为的基因家族分析工具,你不如说他是比较基因组分析工具,那么还显得高大上一些。
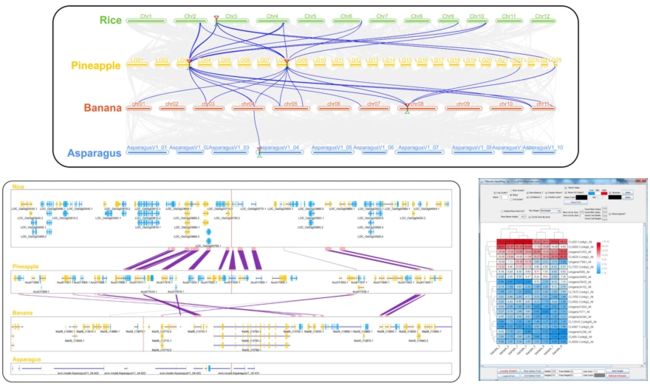
TBtools是一个比较基因组分析工具?
基因组-点图?KaKs计算?共线性分析?多基因组共线性可视化?
这半个月以来,课题组的安排下,我参与了一些基因组分析相关的工作;
基因组分析,本身确实是一个耗费智力和体力的活。分析过程中,也发现了一些或许可以让所有人都从中获取生物信息的分析手段。
于是我花了两个晚上的时间,写了几个工具。
加上一些以前TBtools中就有的工具,相信对做基因组分析的朋友会有一定的帮助。
但是,请注意,除非是赞助我们课题组的户外拓展活动或者合作单位,否则我并不保证这些工具的使用。
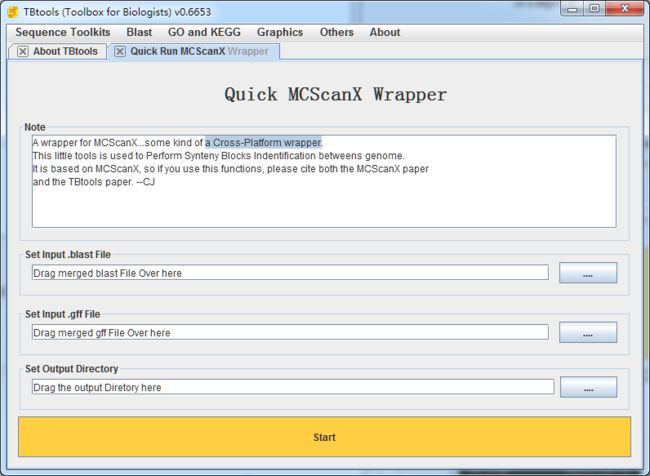
所有人都能用的MCScanX
MCScanX是比较基因组分析中常用的工具。我已经将其打包到TBtools中,所以即使是windows用户,也可以轻松进行分析。此外,也不要求用户保证gff文件和blast文件的名字一致。
多基因组共线性可视化
关于这个工具... 详细见公众号以前的推文。
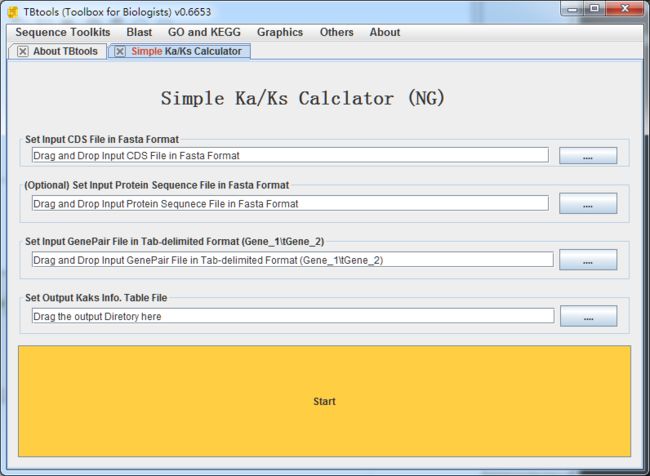
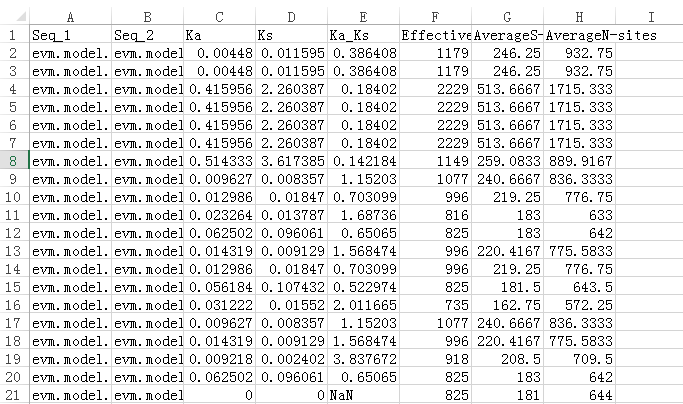
简单的KaKs批量计算工具
Ka/Ks的计算,常常会被人问题。事实上,如果只是简单的进行NG算法的计算,是非常容易实现的。目前用的广泛的,或许是KaksCalculator2和PAML。这两个软件都是大牛级的软件。在TBtools中,我终于还是开放了去年还是前年实现的NG计算逻辑,并打了非常方便的GUI。用户几乎只有有CDS序列和基因对信息就可以直接进行计算,而完全不用浪费时间在文件格式整理上。
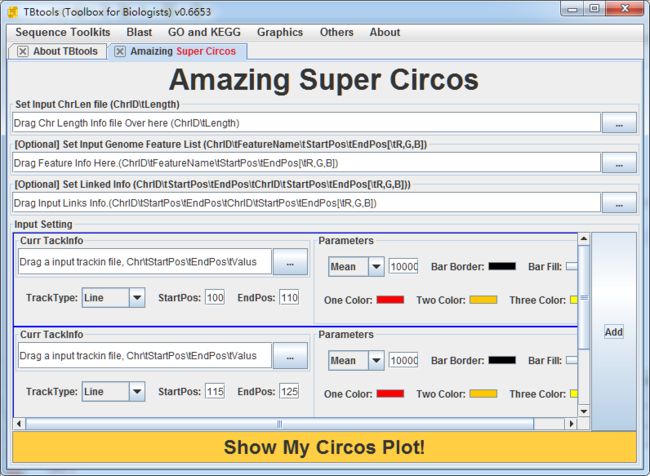
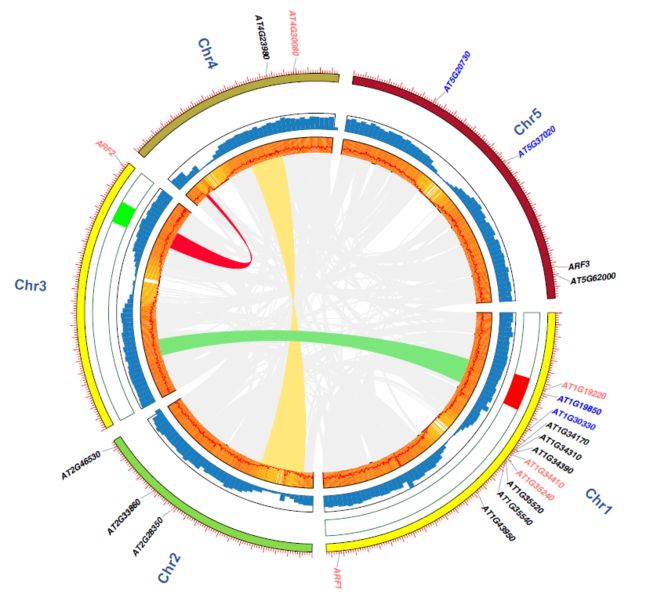
Circos图绘制工具
其实,还可以玩游戏
写在最后
工具是不断地优化和发展的。
分析门槛也是会被不断打破的。
或许让所有人,都能有开展一些分析的能力,也是推进一些事物发展的方式。
欢迎尚未加到TBtools使用交流QQ群的朋友,加入
下载最新版,与其他用户朋友交流使用经验与技巧
题外
课题组每年暑期有内部生信入门培训,主要是对实验室新生开展(以及湿实验为主的成员)培训。一直有收到其他课题组想要了解我们课题组生信数据分析的想法。故,在博导的提议和课题组的讨论后,我们近期计划,在本年度暑期(7~8月份之间)对外增设生信基础培训名额10枚(前面每年只是课题组内培训,而不对外)。具体请见https://mp.weixin.qq.com/s/OtmeTErd9f9rvjJPtBKjMw
欢迎访问本课题组网站
园艺植物小分子RNA与基因组研究-夏瑞课题组
课题组主页:http://xialab.scau.edu.cn/