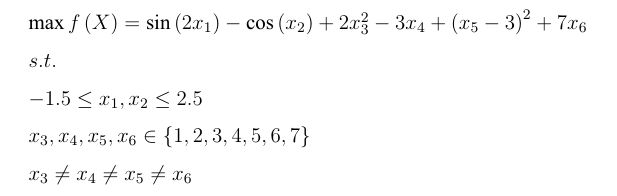遗传算法混合编码
对于大多数复杂的实际问题,单靠一种编码是很难甚至是完全无法进行求解的。例如变量中既有连续的实数,又有整数,某些变量还满足排列编码(即多个变量之间互不相等)的特征,这个时候需要混合编码才能很好地对问题进行求解。
本文采用Python高性能实用型进化算法工具箱Geatpy来进行相关的实验探究,Geatpy工具箱内置支持复杂混合编码的进化优化,并且只需把待求解的问题编写在”问题类“中,然后调用内置的进化算法模板便能求解问题,通用性很强。
Geatpy的官方教程如下:
http://geatpy.com/index.php/geatpy%E6%95%99%E7%A8%8B/
Geatpy的染色体本身有三种最基础的编码方式:'BG'(二进制/格雷编码)、'RI'(实数整数混合编码)以及'P'(排列编码),这意味着一条染色体只能是这三种编码方式的其中一种。因此当需要更加复杂的编码时,需要用多条染色体来进行协同表达。
Geatpy的四个大类中的Population种群类只支持单染色体,其Chrom属性(种群染色体矩阵)中每一行对应的是种群的一条染色体,因此只支持'BG'、'RI'或'P'中的一种编码方式。
这里引入PsyPopulation种群类,它是继承了Population类的一个新的类,它用Linds列表代替Population中的Lind来存储各染色体的长度;用Encodings代替Population中的Encoding来存储各染色体的编码方式;用Fields代替Population中的Field来存储各染色体的译码矩阵;用Chroms代替Population中的Chrom来存储各个染色体矩阵。PsyPopulation和Population的UML类图对比如下所示:
上图中的Field, Chrom, ObjV, FitnV, CV, Phen等均满足Geatpy的数据结构(详见“Geatpy数据结构”章节)。
如果在进化过程中采用的是PsyPopulation类的种群,而不采用Population,那么原有的算法模板都要作出一定的修改。首先是染色体的初始化,需要遍历各种编码的染色体进行初始化操作。然后对于重组和变异,也是需要遍历各种编码的染色体分别进行重组和变异;因此Geatpy为各个算法模板提供其对应的多染色体版本,并且用“psy”字符串加以标识。比如多目标优化NSGA2的算法模板“moea_NSGA2_templet”,其对应的多染色体版本为“moea_psy_NSGA2_templet”(算法的源码可点开超链接进行查看)。
下面以一个单目标带约束问题为例,阐述如何使用多染色体混合编码求解问题:
该问题本来可以像以往那样,直接使用实整数编码'RI'的染色体来表达所有的变量,然后加一个不等式约束使得x3 ≠ x4 ≠ x5 ≠ x6 。但这里我们采用另一种编码方法:不难发现x3, x4, x5, x6 ∈ {1, 2, 3, 4, 5, 6, 7}和x3 ≠ x4 ≠ x5 ≠ x6 正好符合排列编码的特征,即x3, x4, x5, x6 是从{1,2,3,4,5,6,7}中任意挑选出4个数字的排列。因此前两个变量用'RI'编码(实整数编码)的染色体(把对应的varTypes设为0标记这2个变量为连续型变量);后4个变量用'P'编码(排列编码)的染色体。实验代码如下:
第一步:编写问题类MyProblem,写在MyProblem.py文件中。
# -*- coding: utf-8 -*-
"""MyProblem.py"""
import numpy as np
import geatpy as ea
class MyProblem(ea.Problem): # 继承Problem父类
def __init__(self):
name = 'MyProblem' # 初始化name(函数名称,可以随意设置)
M = 1 # 初始化M(目标维数)
# 初始化maxormins(目标最小最大化标记列表,1:最小化;-1:最大化)
maxormins = [-1]
Dim = 6 # 初始化Dim(决策变量维数)
# 初始化决策变量的类型,元素为0表示变量是连续的;1为离散的
varTypes = [0,0,1,1,1,1]
lb = [-1.5,-1.5,1,1,1,1] # 决策变量下界
ub = [2.5,2.5,7,7,7,7] # 决策变量上界
lbin = [1] * Dim # 决策变量下边界
ubin = [1] * Dim # 决策变量上边界
# 调用父类构造方法完成实例化
ea.Problem.__init__(self, name, M, maxormins, Dim, varTypes, lb, ub, lbin, ubin)
def aimFunc(self, pop): # 目标函数
X = pop.Phen # 得到决策变量矩阵
x1 = X[:, [0]]
x2 = X[:, [1]]
x3 = X[:, [2]]
x4 = X[:, [3]]
x5 = X[:, [4]]
x6 = X[:, [5]]
pop.ObjV = np.sin(2*x1) - np.cos(x2) + 2*x3**2 -3*x4 + (x5-3)**2 + 7*x6 # 计算目标函数值,赋值给pop种群对象的ObjV属性第二步:编写执行脚本,写在main.py文件中。
# -*- coding: utf-8 -*-
"""main.py"""
import numpy as np
import geatpy as ea # import geatpy
from MyProblem import MyProblem # 导入自定义问题接口
"""===========================实例化问题对象========================"""
problem = MyProblem() # 生成问题对象
"""=============================种群设置==========================="""
NIND = 40 # 种群规模
# 创建区域描述器,这里需要创建两个,前2个变量用RI编码,其余用排列编码
Encodings = ['RI', 'P']
Field1 = ea.crtfld(Encodings[0], problem.varTypes[:2], problem.ranges[:,:2], problem.borders[:,:2])
Field2 = ea.crtfld(Encodings[1], problem.varTypes[2:], problem.ranges[:,2:], problem.borders[:,2:])
Fields = [Field1, Field2]
population = ea.PsyPopulation(Encodings, Fields, NIND) # 实例化种群对象(此时种群还没被初始化,仅仅是完成种群对象的实例化)
"""===========================算法参数设置=========================="""
myAlgorithm = ea.soea_psy_SEGA_templet(problem, population) # 实例化一个算法模板对象
myAlgorithm.MAXGEN = 25 # 最大进化代数
"""======================调用算法模板进行种群进化===================="""
[population, obj_trace, var_trace] = myAlgorithm.run() # 执行算法模板
population.save() # 把最后一代种群的信息保存到文件中
# 输出结果
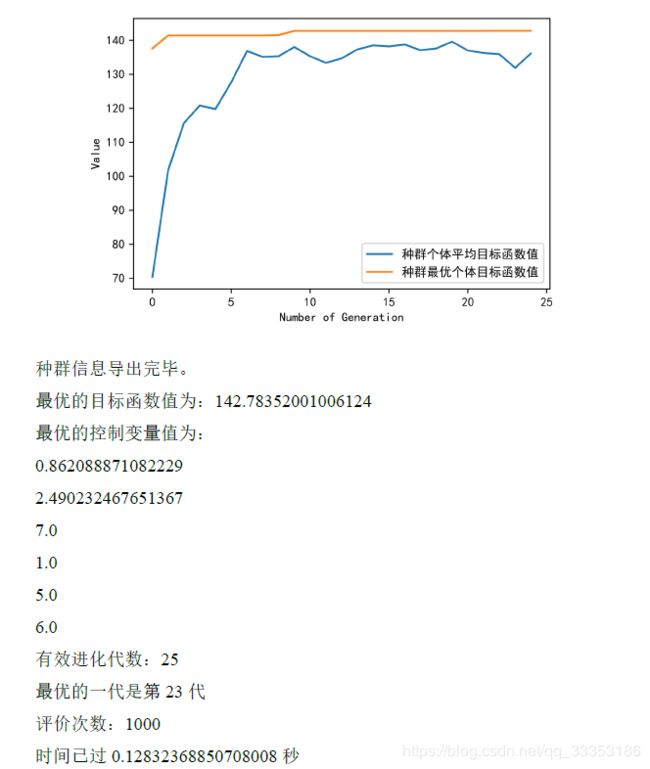
best_gen = np.argmax(obj_trace[:, 1]) # 记录最优种群是在哪一代
best_ObjV = obj_trace[best_gen, 1]
print('最优的目标函数值为:%s'%(best_ObjV))
print('最优的控制变量值为:')
for i in range(var_trace.shape[1]):
print(var_trace[best_gen, i])
print('有效进化代数:%s'%(obj_trace.shape[0]))
print('最优的一代是第 %s 代'%(best_gen + 1))
print('评价次数:%s'%(myAlgorithm.evalsNum))
print('时间已过 %s 秒'%(myAlgorithm.passTime))代码解析:
Geatpy的问题类是不用管种群用何种编码方式的,只需把问题描述清楚。上面的代码中,种群染色体采用何种编码方式是在执行脚本main.py中进行设置的;由于上述问题中前两个变量是实数,后四个变量是互不相等的整数,于是设置两个Encoding,把它存储在Encodings列表中,因此有“Encodings = ['RI', 'P']”,由于有两种编码,因此需要创建两个译码矩阵Field1和Field2,最后把它们存储到Fields列表中。在随后的实例化种群对象时,要注意用的种群类是PsyPopulation而不是Population。因为PsyPopulation是Population衍生出来的支持多染色体混合编码的种群类。最后在实例化算法模板对象时,要注意挑选的算法模板的名称要带有“psy”字符串,表示这是一个支持多染色体混合编码的算法模板。由于本例是一个单目标优化问题,因此可以采用“soea_SEGA_templet”的多染色体版本:“soea_psy_SEGA_templet”算法模板进行求解(算法的源码可点开超链接进行查看)。
运行main.py,得到如下结果:
以下是Geatpy目前内置的多染色体版本的进化算法模板,其具体代码均可在源码中查看到:
• soea_psy_EGA_templet (精英保留的多染色体遗传算法模板)
• soea_psy_SEGA_templet (增强精英保留的多染色体遗传算法模板)
• soea_psy_SGA_templet (最简单、最经典的多染色体遗传算法模板)
• soea_psy_GGAP_SGA_templet (带代沟的多染色体简单遗传算法模板)
• soea_psy_studGA_templet (多染色体种马遗传算法模板)
• soea_psy_steady_GA_templet (多染色体稳态遗传算法模板)
• moea_psy_awGA_templet (基于awGA 算法的多染色体多目标进化算法模板)
• moea_psy_NSGA2_archive_templet (带全局存档的多染色体多目标进化NSGA-II 算
法模板)
• moea_psy_NSGA2_templet (基于NSGA-II 算法的多染色体多目标进化算法模板)
• moea_psy_NSGA3_templet (基于NSGA-III 算法的多染色体多目标进化算法模板)
• moea_psy_RVEA_templet (基于RVEA 算法的多染色体多目标进化算法模板)
• moea_psy_RVEA_RES_templet (基于带参考点再生策略的多染色体RVEA 算法的多
目标进化算法模板)
更多相关介绍可以详见博客:
https://blog.csdn.net/qq_33353186/article/details/82014986
https://blog.csdn.net/qq_33353186/article/details/82020507
https://blog.csdn.net/qq_33353186/article/details/82047692
https://blog.csdn.net/qq_33353186/article/details/82082053
欢迎继续跟进!感谢!