小牛的学习笔记--乳腺癌症数据分析
二分类;损失函数精确度的图像;加深模型
一.深度学习五步骤
- 加载数据集
- 定义模型
- 编译模型
- 训练模型(fit)
- 评估模型
二.这里以乳腺癌症数据分析为例
这里没有分开训练集和测试集
导入包
from keras.models import Sequential
from keras.layers import Dense
import numpy
from matplotlib import pyplot as plt
from keras.utils.vis_utils import plot_model
1.加载数据集
#numpy.loadtxt指定文本数据间的间隔符号是什么
#X:[:,0:8]取所有行的第0到第7列(特征值)Y:[:,8]所有行的最后一列,是否得病的列(类别值)
dataset = numpy.loadtxt("pima-indians-diabetes.csv", delimiter=",")
X = dataset[:,0:8]
Y = dataset[:,8]
2.定义模型
激活函数是建模型的时候使用的
#Sequential:为最简单的线性、从头到尾的结构顺序,不分叉。
模型的基本组件一般需要:
- 1、model.add,添加层;
- 2、model.compile,模型训练的BP模式设置;
- 3、model.fit,模型训练参数设置 + 训练;
- 4、模型评估
- 5、模型预测
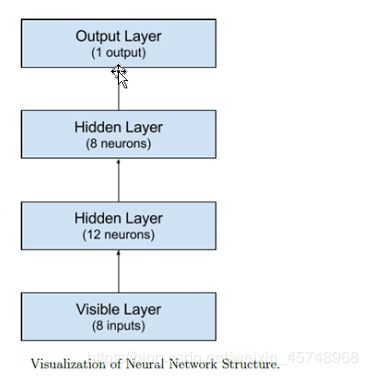
add函数添加全连接层
第一层输入维度是8,因为有8个属性值
第二层为隐含层第一层,12个神经元连接到第二层的8个神经元
第三层为隐含层第二层,8个神经元
第四层输出层,1个神经元
sigmoid函数:用于隐层神经元输出,取值范围为(0,1),它可以将一个实数映射到(0,1)的区间,可以用来做二分类。
relu函数:用于隐层神经元输出
二分类中间层relu
最后一层sigmoid
附上链接:关于Sequential这块讲的很详细
#create model
model = Sequential()
model.add(Dense(12, input_dim=8, activation='relu'))
model.add(Dense(8, activation='relu'))
#model.add(Dense(4, activation='relu'))
model.add(Dense(1, activation='sigmoid'))#sigmoid函数
这里注释了神经元为4的一层
3.编译模型
损失函数;优化函数;精确度
损失函数:二分类的损失函数:直接指定用这个函数binary crossentropy
优化函数:默认的梯度下降算法adam
精确度:分类的精确度放在矩阵里面accuracy
#Compile model
model.compile(loss='binary_crossentropy',optimizer='adam',metrics=['accuracy'])
4.训练模型
要指定epoches和batch_size
fit模型就是训练模型
epochs参数是迭代的次数,batch_size是批处理的个数
#fit the model
history=model.fit(X,Y,epochs=100,batch_size=10,verbose=2)
5.评估模型
evaluate()并且画出神经元的模型图
#evaluate the model
scores=model.evaluate(X,Y)
print("\n%s: %.2f%%" % (model.metrics_names[1], scores[1]*100))
model.summary()
plot_model(model, to_file='model1.png', show_shapes=True)
结果输出:
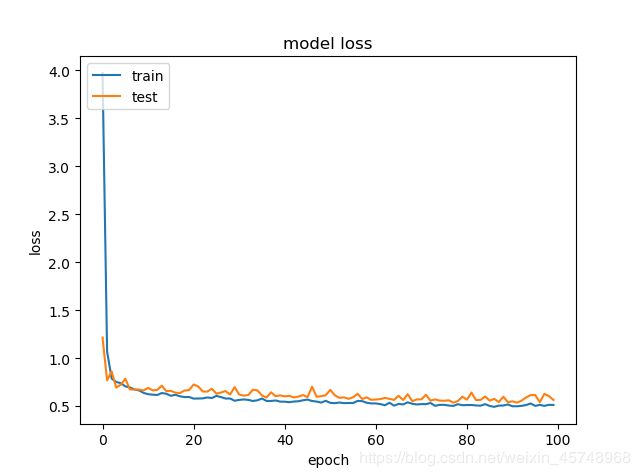
三.画出损失函数和精确度的变化曲线
用上述例子不能直接画出损失函数和精确度的变化,因为这里的训练集同时作为为验证集,必须吧训练集和验证集分开才可,同样用该数据集,只需要改改部分代码就可画出损失函数和精确度的变化曲线
添加代码:
导入的包部分:
from sklearn import model_selection#分训练集和训练集
加载数据集的部分:
这里数据集训练集和测试集2 8分
validation_size=0.20
seed =7
scoring= 'accuracy'
X_train, X_validation, Y_train,Y_validation = model_selection.train_test_split(X, Y, test_size=validation_size, random_state=seed)
训练模型部分:
训练模型中注意加上validation_split=0.2
history=model.fit(X,Y,validation_split=0.2, epochs=100,batch_size=10,verbose=2)
最后画图曲线图:
# summarize history for accuracy
plt.figure('acc')
plt.plot(history.history['accuracy'])
plt.plot(history.history['val_accuracy'])
plt.title('model accuracy')
plt.ylabel('accuracy')
plt.xlabel('epoch')
plt.legend(['train', 'test'], loc='upper left')
plt.show()
# summarize history for loss
plt.figure('loss')
plt.plot(history.history['loss'])
plt.plot(history.history['val_loss'])
plt.title('model loss')
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['train', 'test'], loc='upper left')
plt.show()
可能存在报错:
KeyError: 'val_acc’或者KeyError: 'val_accuracy’
可能跟keras库版本相关,这俩不行相互改就完事了。