Prodigal:原核基因识别和翻译起始位点鉴定
文章目录
- Prodigal:原核基因识别和翻译起始位点鉴定
- 热心肠日报
- 摘要
- 背景
- 结果
- 结论
- 实现 Implementation
- 图1. Prodigal算法的伪代码方式描述
- 表1. Prodigal中的动态规划连接类型
- 表2. Prodigal中的Shine-Dalgarno RBS基序
- 图2. 动态规划连接模式图
- 结果与讨论 Results and Discussion
- 表3. 基因预测效果
- 表4. 与Genbank注释比较
- 表5. 主流基因预测方法在一些微生物组基因组上的比较
- 扩展阅读
- 猜你喜欢
- 写在后面

Prodigal:原核基因识别和翻译起始位点鉴定
Prodigal: prokaryotic gene recognition and translation initiation site identification
BMC Bioinformatics, [2.448]
2010-3-8 Software
DOI: https://doi.org/10.1186/1471-2105-11-119
第一作者:Doug Hyatt1,2*
通讯作者:Doug Hyatt1,2*
其它作者:Gwo-Liang Chen1, Philip F LoCascio1, Miriam L Land1,3, Frank W Larimer1,2, Loren J Hauser1,3
作者主要单位:
1美国田纳西州橡树岭,橡树岭国家实验室,计算生物学与生物信息学小组(Computational Biology and Bioinformatics Group, Oak Ridge National Laboratory, Oak Ridge, TN, 37831, USA)
2美国田纳西州诺克斯维尔,田纳西大学基因组科学技术研究生院(Genome Science and Technology Graduate School, The University of Tennessee, Knoxville, TN, 37996, USA)
热心肠日报
Prodigal鉴定细菌基因组和宏基因组中的基因
- Prodigal是细菌基因预测领域的主流软件,在宏基因组领域也有较多应用;
- 软件主要采用动态规划算法,较其它主流工具相比有更接近参考注释的水平;
- 软件引用超3千多次,并被众多分析工具整合,如抗生素抗生基因鉴定流程rgi、基因注释流程prokka、分箱流程das_tool和、基因簇鉴定流程antismash等;
- 软件运行速度快,支持多线程,原生安装或conda安装都非常方便。
点评:由橡树岭国家实验室计算生物学与生物信息学小组开发的Prodigal是原核生物基因鉴定的流行软件,引用3千多次可谓神作。而且此软件被众多分析流程整合,如抗生素抗生基因鉴定rgi、分箱结果去冗余drep(https://www.mr-gut.cn/papers/read/1066969984)、宏基因组流程anvio、基因注释流程prokka(https://www.mr-gut.cn/papers/read/1076111428)、基因组质量评估checkm-genome、分箱流程das_tool(https://www.mr-gut.cn/papers/read/1036660372)、基因簇鉴定antismash等,引用被严重低估。目前在细菌菌组、宏基因组领域有非常广泛的应用。
摘要
背景
在过去的十年中,微生物中自动基因预测的质量稳步提高,但仍有改进的空间。理想的目标是增加基因以及每个基因的翻译起始位点的正确识别的数量,并减少假阳性的总数。
结果
凭借我们在联合基因组研究所手动管理基因组方面的多年经验,开发了一种新的基因预测算法,称为Prodigal(原核动态规划/编程基因发现算法)。借助Prodigal,我们特别关注改善基因结构预测,改善翻译起始位点识别和减少假阳性的三个目标。我们将Prodigal的结果与现有的基因发现方法进行了比较,以证明它满足了这些目标。
结论
我们建立了一个快速,轻量级的开源基因预测程序,称为Prodigal http://compbio.ornl.gov/prodigal/ 。与现有方法相比,Prodigal取得了良好的结果,我们认为这将是自动化微生物注释流程的宝贵资源。
原始网站已经无法打开,软件github主页:https://github.com/hyattpd/Prodigal ,2016年2月版更新 2.6.3
实现 Implementation
图1. Prodigal算法的伪代码方式描述
Pseudocode description of the Prodigal algorithm
表1. Prodigal中的动态规划连接类型
Table 1 Dynamic Programming Connections in Prodigal
表2. Prodigal中的Shine-Dalgarno RBS基序
Table 2 Shine-Dalgarno RBS Motifs in Prodigal
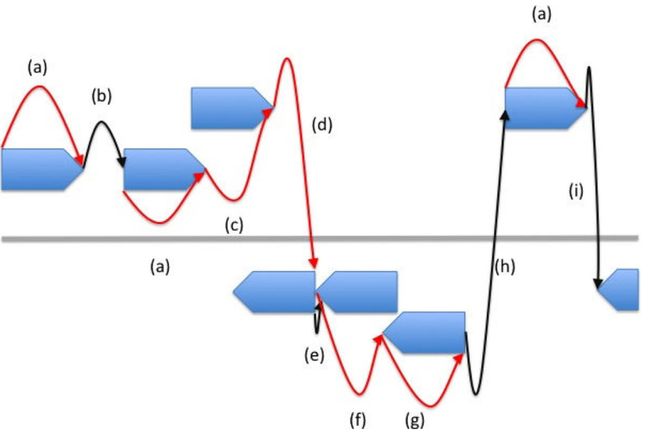
图2. 动态规划连接模式图
Illustration of the dynamic programming connections in Prodigal
结果与讨论 Results and Discussion
表3. 基因预测效果
Table 3 Gene Prediction Performance
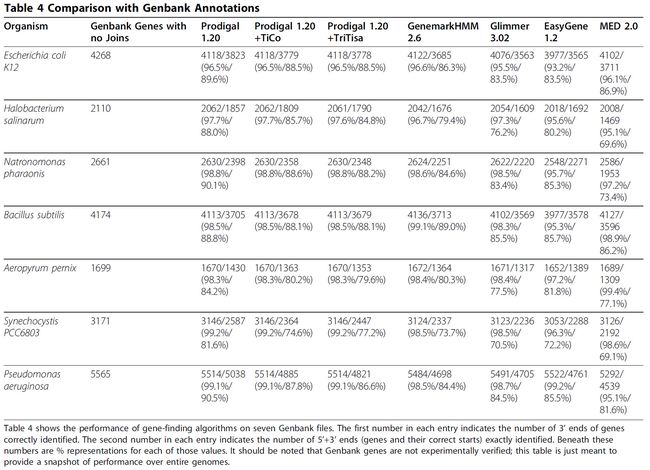
表4. 与Genbank注释比较
Table 4 Comparison with Genbank Annotations
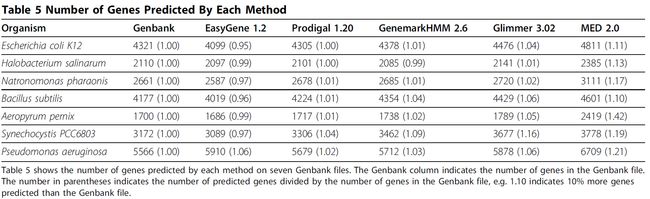
表5. 主流基因预测方法在一些微生物组基因组上的比较
Table 5 Number of Genes Predicted By Each Method
扩展阅读
- 基因注释Prokka、Prodigal
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA