SPIEC-EASI的微生物网络构建示例
SPIEC-EASI的微生物网络构建示例
续前文“微生物网络”,本篇继续简介SPIEC-EASI的网络构建方法。
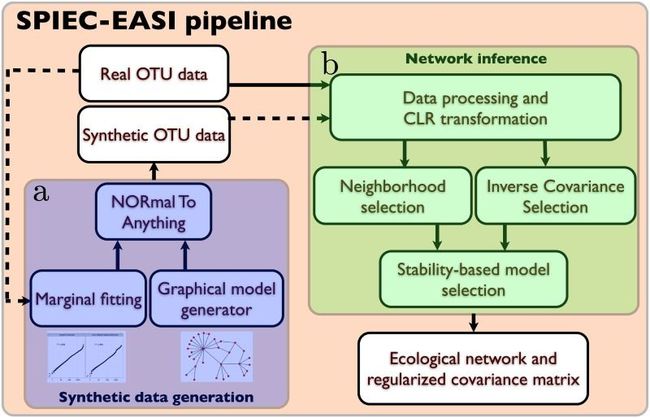
16S rRNA测序技术为揭示了微生态系统中的系统发育和微生物种群丰富度提供了便利。微生物通常不是独立存在的,且微生物群落结构的变化与环境密切相关,传统关联网络的推理功能严重不足,因此需要新的统计工具确定潜在的机制。SPIEC-EASI(Sparse InversE Covariance estimation for Ecological Association and Statistical Inference,生态关联的稀疏逆协方差估计与统计推断)是一种从16S rRNA扩增子测序数据集推断微生物生态网络的新工具。SPIEC-EASI将为成分数据分析开发的数据转换与图形化的模型推理框架相结合,该框架假设底层的生态关联网络是稀疏的,并依赖于稀疏邻域和逆协方差选择算法重建网络(Kurtz et al, 2015)。
SPIEC-EASI推断包括两个步骤。首先,将来自成分数据分析领域的转换应用于OTU数据。随后,SPIEC-EASI使用(1)邻域选择或(2)稀疏逆协方差选择方法之一从转换后的数据进行交互图估计。与SparCC和CCREPE中使用的经验相关性或协方差估计不同,SPIEC-EASI使用条件独立性的概念寻求推断底层图形模型。
此外,除了应用于16S细菌群落测序,对于ITS、18S,或者蛋白质组学数据等的联合推断,SPIEC-EASI同样能够适用。
接下来主要以微生物网络为例,展示SPIEC-EASI工作的一般过程。
SPIEC-EASI的微生物共发生网络构建
SPIEC-EASI的github链接,包含源码及操作文档(本篇也主要按该文档的过程来简介):
https://github.com/zdk123/SpiecEasi
首先安装加载SpiecEasi包。
#链接到 github 安装 SpiecEasi 包
library(devtools)
devtools::install_github('zdk123/SpiecEasi')
library(SpiecEasi)
1、准备数据
使用SpiecEasi提供的测试数据作演示。
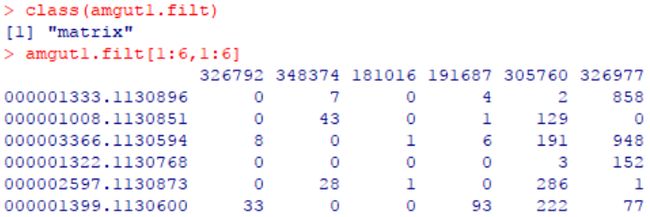
数据集“amgut1.filt”,为来自某肠道微生物群落的测序数据,由289个样本(行)和127种OTU(列)丰度组成的矩阵。
#示例数据,详情 ?amgut1.filt
data(amgut1.filt)
class(amgut1.filt)
amgut1.filt[1:6,1:6]
2、构建非含权的无向网络
SpiecEasi提供了一个整合函数spiec.easi(),可直接应用于该微生物群落组成丰度数据集,其能够完成数据标准化、稀疏逆协方差估计、模型选择等在内的多步过程,最后得到关联矩阵,以用于构建网络。
因此SpiecEasi使用起来非常方便简单。
#构建网络,详情 ?spiec.easi
#data 指定未作任何预转化的 OTU 丰度表,将通过内部方法标准化
#method 提供了两种估计方法,即 glasso 和 mb,分别展示(二者间可能会差别较大)
se.gl.amgut <- spiec.easi(data = amgut1.filt, method = 'glasso', lambda.min.ratio = 0.01,
nlambda = 20, pulsar.params = list(rep.num = 50))
se.mb.amgut <- spiec.easi(amgut1.filt, method = 'mb', lambda.min.ratio = 0.01,
nlambda = 20, pulsar.params = list(rep.num=50))
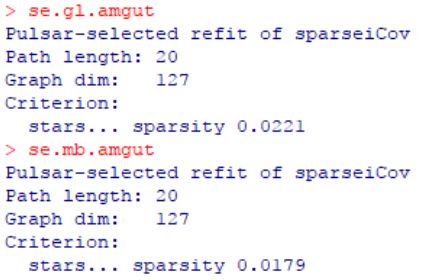
se.gl.amgut
se.mb.amgut
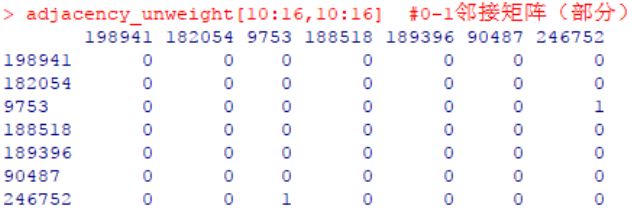
#0-1 矩阵获得,即非含权的邻接矩阵(1 代表二者互作,0 代表无互作,不包含互作类型或强度)
#以 glasso 的结果为例
summary(se.gl.amgut)
adjacency_unweight <- data.frame(as.matrix(se.gl.amgut$refit$stars))
rownames(adjacency_unweight) <- colnames(amgut1.filt)
colnames(adjacency_unweight) <- colnames(amgut1.filt)
adjacency_unweight[10:16,10:16]
#如需输出 0-1 关系的邻接矩阵
write.table(adjacency_unweight, 'adjacency_unweight.glasso.txt', col.names = NA, sep = '\t', quote = FALSE)
如此获得了OTU间是否存在互作关联,即0-1类型的网络邻接矩阵(1代表二者互作,0代表无互作),并根据这种关系构建网络。
igraph包提供了便利的网络操作方法,SPIEC-EASI允许将识别的OTU间互作结果转化为igraph网络对象,便于后续的统计分析,或作图。
#构建 igraph 网络,便于后续的统计分析,或作图
#将上述邻接矩阵转化为 igraph 的邻接列表,详情 ?adj2igraph
library(igraph)
ig.gl <- adj2igraph(getRefit(se.gl.amgut), vertex.attr = list(label = colnames(amgut1.filt)))
ig.mb <- adj2igraph(getRefit(se.mb.amgut), vertex.attr = list(label = colnames(amgut1.filt)))
ig.gl
ig.mb
#简单作图展示下
#本篇只展示网络生成,关于 R 的网络图可视化方法略,还请自行了解
vsize <- rowMeans(clr(amgut1.filt, 1)) + 6
am.coord <- layout.fruchterman.reingold(ig.mb)
par(mfrow = c(1, 2))
plot(ig.gl, layout = am.coord, vertex.size = vsize, vertex.label = NA, main = 'glasso')
plot(ig.mb, layout = am.coord, vertex.size = vsize, vertex.label = NA, main = 'MB')
#非含权的网络输出,以 glasso 的结果为例
#节点属性列表
node.gl <- data.frame(id = as.vector(V(ig.gl)), label = V(ig.gl)$label)
write.table(node.gl, 'unweight.glasso_node.txt', sep = '\t', row.names = FALSE, quote = FALSE)
#边列表
edge.gl <- data.frame(as_edgelist(ig.gl))
edge.gl <- data.frame(source = edge.gl[[1]], target = edge.gl[[2]], weight = E(ig.gl)$weight)
write.table(edge.gl, 'unweight.glasso_edge.txt', sep = '\t', row.names = FALSE, quote = FALSE)
#graphml 格式,可使用 gephi 软件打开并进行可视化编辑
write.graph(ig.gl, 'unweight.glasso.graphml', format = 'graphml')
#gml 格式,可使用 cytoscape 软件打开并进行可视化编辑
write.graph(ig.gl, 'unweight.glasso.gml', format = 'gml')
这些操作输出了其它类型的网络文件(如边列表、graphml、gml等),便于由不同的网络分析软件识别,满足进一步的分析或可视化需要。
3、估计边的权重,获得含权的无向网络
在上一步中,获得的网络中仅包含0-1关系(1代表二者互作,0代表无互作,不包含互作类型或强度)。如果有需要,可以使用基础模型中的术语评估边的权重。如下示例,将获得物种关系的互作类型(可视为正负相关)以及互作强度(绝对值可视为关联强度的大小)。
备注:由于SPIEC-EASI基于惩罚估计量(penalized estimators),因此边权重请勿直接与Pearson/Spearman等相关系数进行比较。
#估计边的权重
library(Matrix)
secor <- cov2cor(getOptCov(se.gl.amgut))
sebeta <- symBeta(getOptBeta(se.mb.amgut), mode = 'maxabs')
elist.gl <- summary(triu(secor*getRefit(se.gl.amgut), k = 1))
elist.mb <- summary(sebeta)
head(elist.gl)
head(elist.mb)
i和j分别代表网络中两个互作的节点,即它们之间存在边,x就是求得的边的权重。
随后,可以将这种权重信息添加到网络中,获得并输出含权网络。
#将权重合并到网络中,以 glasso 的结果为例
elist.gl <- data.frame(elist.gl)
names(elist.gl) <- c('source', 'target', 'weight')
elist.gl <- elist.gl[order(elist.gl$source, elist.gl$target), ]
E(ig.gl)$weight <- elist.gl$weight
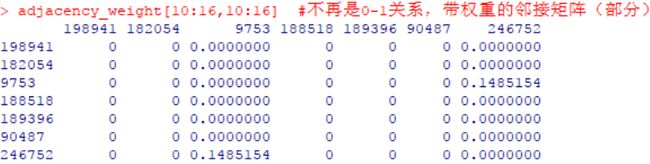
#输出带权重的邻接矩阵,不再是 0-1 关系,而替换为具体数值(可表示两变量关联程度)
adjacency_weight <- as.matrix(get.adjacency(ig.gl, attr = 'weight'))
rownames(adjacency_weight) <- colnames(amgut1.filt)
colnames(adjacency_weight) <- colnames(amgut1.filt)
write.table(data.frame(adjacency_weight), 'adjacency_weight.glasso.txt', col.names = NA, sep = '\t', quote = FALSE)
#以及含权重的边列表
edge.gl <- data.frame(as_edgelist(ig.gl))
edge.gl <- data.frame(source = edge.gl[[1]], target = edge.gl[[2]], weight = E(ig.gl)$weight)
write.table(edge.gl, 'weight.glasso_edge.txt', sep = '\t', row.names = FALSE, quote = FALSE)
#graphml 格式,可使用 gephi 软件打开并进行可视化编辑
write.graph(ig.gl, 'weight.glasso.graphml', format = 'graphml')
#gml 格式,可使用 cytoscape 软件打开并进行可视化编辑
write.graph(ig.gl, 'weight.glasso.gml', format = 'gml')
如此获得的网络邻接矩阵中,包含了OTU关系的互作类型(可视为正负相关)以及互作强度(绝对值可视为关联强度的大小)。
此外,上述也输出了其它类型的网络文件(如边列表、graphml、gml等),便于由不同的网络分析软件识别,满足进一步的分析或可视化需要。
4、常见网络拓扑特征计算
通常是为了识别网络属性,寻找关键的节点或互作的边等,可参考前文等描述:
微生物共发生网络中节点度的幂律分布
网络拓扑结构-节点和边特征
网络拓扑结构-网络图的凝聚性特征
网络模块内连通度(Zi)和模块间连通度(Pi)
* 5、与phyloseq包的结合
phyloseq包是目前在微生物组分析领域非常流行的R包。无独有偶,SpiecEasi提供了包装器,可直接与phyloseq对象一起使用。参考以下示例。
#与 phyloseq 结合使用
library(SpiecEasi)
library(phyloseq)
#示例数据,内置的 phyloseq 对象
data('amgut2.filt.phy')
#构建非加权的无向网络
se.gl.amgut2 <- spiec.easi(amgut2.filt.phy, method = 'glasso', lambda.min.ratio = 0.01,
nlambda = 20, pulsar.params = list(rep.num = 50))
#邻接矩阵转化为 igraph 的邻接列表
ig2.gl <- adj2igraph(getRefit(se.gl.amgut2), vertex.attr = list(name = taxa_names(amgut2.filt.phy)))
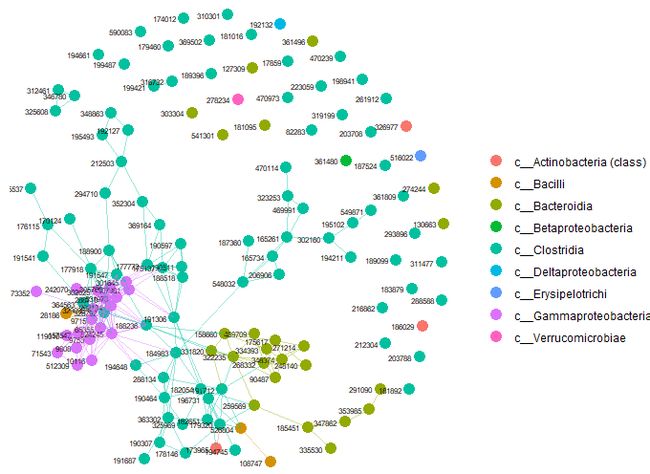
#可视化
plot_network(ig2.gl, amgut2.filt.phy, type = 'taxa', color = 'Rank3')
SPIEC-EASI的跨组学数据的关联推断
除了单纯的基于16S/ITS/18S微生物物种的互作外,SpiecEasi同样能够用于跨组学的分析。SpiecEasi提供了包装器,可用于处理在相同样本上测序的多种类型数据。
参考以下示例,评估16S细菌物种和蛋白质组学功能的相关性,这里同样与phyloseq对象一起使用。
#跨组学数据的关联推断
library(SpiecEasi)
library(phyloseq)
#示例数据,内置的 phyloseq 对象(16S 的 hmp216S、蛋白质组的 hmp2prot)
data(hmp2)
#关联网络推断
se.hmp2 <- spiec.easi(list(hmp216S, hmp2prot), method = 'glasso', lambda.min.ratio = 0.01,
nlambda = 40,pulsar.params = list(thresh = 0.05))
#可视化
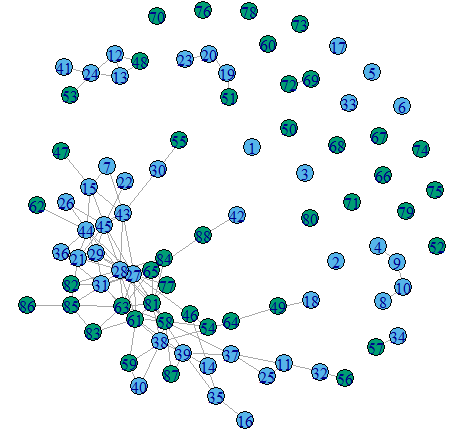
dtype <- c(rep(1,ntaxa(hmp216S)), rep(2,ntaxa(hmp2prot)))
plot(adj2igraph(getRefit(se.hmp2)), vertex.color = dtype + 1, vertex.size = 9)
参考文献
Kurtz Z D, Müller, Christian L, Miraldi E R, et al. Sparse and Compositionally Robust Inference of Microbial Ecological Networks. PLOS Computational Biology, 2015, 11(5):e1004226.
友情链接
相关性和网络分析基础
Pearson、Spearman、Kendall、Polychoric、Polyserial相关系数简介及R计算
网络分析概述之网络基础简介
网络拓扑结构-节点和边特征的简介和R计算
网络拓扑结构-网络图的凝聚性特征和R计算
微生物关联网络推断及一个简单的相关网络示例
CoNet的关联网络推断过程演示
分子生态网络分析(MENA)构建微生物网络示例
微生物共发生网络中节点度的幂律分布和在R中拟合
网络模块内连通度(Zi)和模块间连通度(Pi)及在R中计算
基础统计图(以R作图为主)
柱形图类:堆叠柱形图 分组柱形图 双向柱形图 蝴蝶图
箱线图类:箱线图 提琴图 密度提琴图
饼图类:饼图扇形图 圆环图 蜘蛛饼图 旭日图 星形图
面积图类:堆叠面积图
散点图类:二维散点图 三维散点图 火山图 曼哈顿图
曲线图类:折线图和拟合线
雷达图类:雷达图
集合可视化:在线韦恩图 韦恩图 花瓣图 UpSet图
圈图:关联弦图 简单弦图 基因组变异圈图
三元相图:三元图
树形图:聚类树 聚类树+堆叠柱形图 聚类树+排序散点图
相关图:相关图
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读